摘要
eLife摘要
介绍
结果
Fam60a与Sin3a-Hdac复合物的成分相互作用
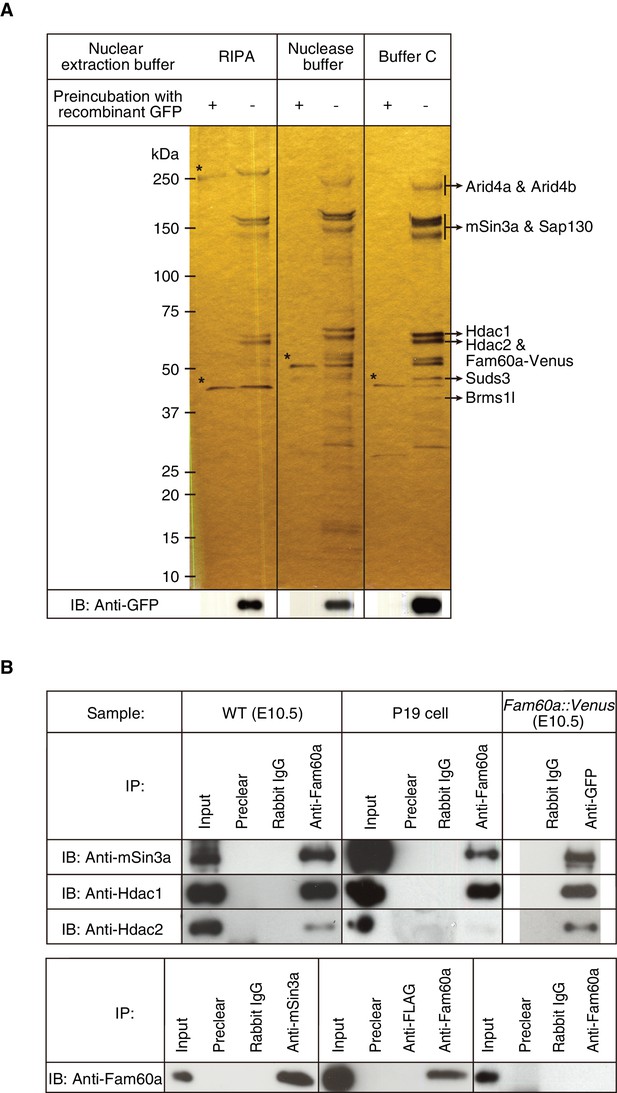
小鼠胚胎中Fam60a-相互作用蛋白的鉴定。
Fam60a系列 小鼠胚胎和成年肠道中的表达
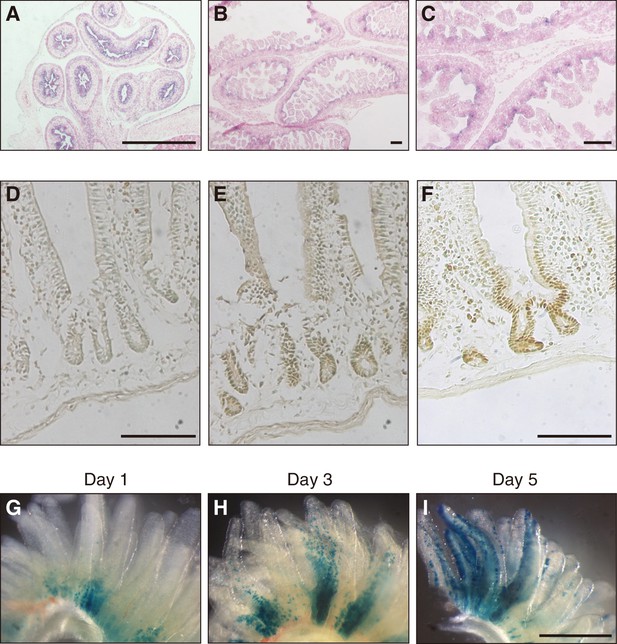
的表达式 Fam60a系列 在胚胎和成年小鼠肠道中。
发育缺陷 Fam60a系列 突变小鼠
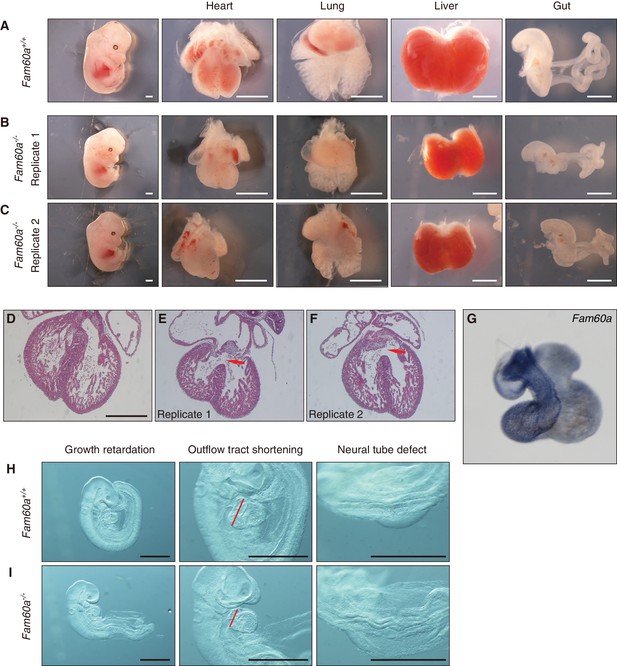
内脏器官生长迟缓 Fam60a系列 –/– 老鼠。
Fam60a被募集到启动子区域并调节基因表达
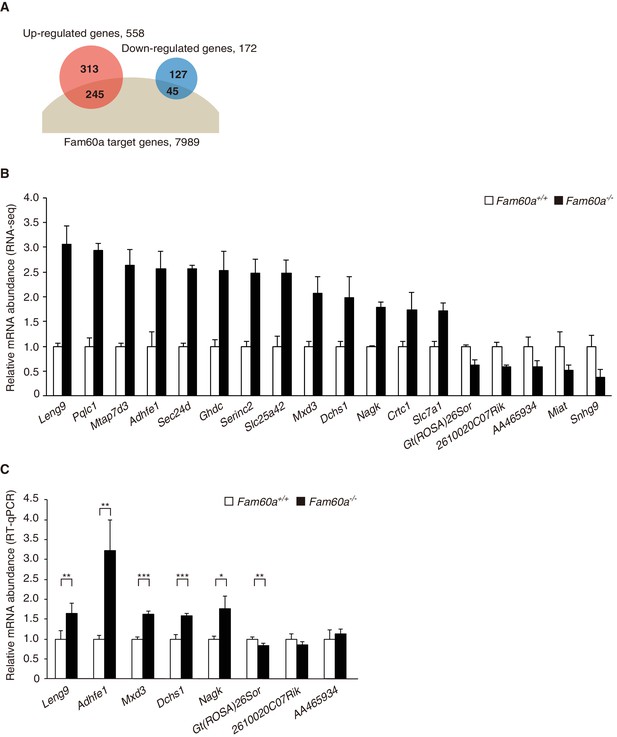
改变基因表达谱 Fam60a系列 –/– 胚胎。
-
图4——源数据1 的靶基因列表 Fam60a系列 . -
https://doi.org/10.7554/eLife.36435.019 -
图4——源数据2 的数值数据 图4B . -
https://doi.org/10.7554/eLife.36435.020 -
图4——源数据3 的数值数据 图4C . -
https://doi.org/10.7554/eLife.36435.021 -
图4-源数据4 的数值数据 图4——补充图2B . -
https://doi.org/10.7554/eLife.36435.022 -
图4-源数据5 的数值数据 图4补充图2C . -
https://doi.org/10.7554/eLife.36435.023 -
图4——源数据6 的数值数据 图4——图补充2D . -
https://doi.org/10.7554/eLife.36435.024
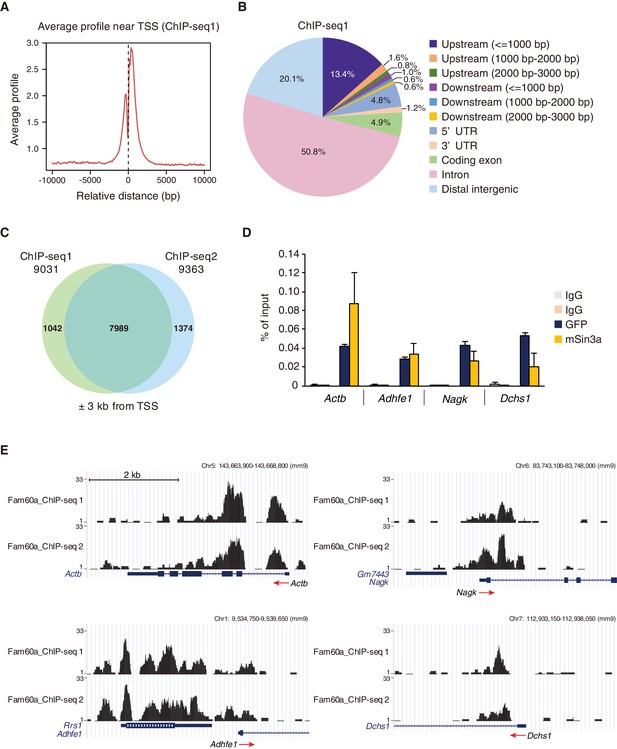
Fam60a对基因启动子的全基因组定位。
-
图5-源数据1 ChIP-seq分析确定的目标基因组区域列表。 -
https://doi.org/10.7554/eLife.36435.028 -
图5-源数据2 的数值数据 图5D . -
https://doi.org/10.7554/eLife.36435.029 -
图5-源数据3 的数值数据 图5——补充图2 . -
https://doi.org/10.7554/eLife.36435.030
Fam60a与DNA甲基化和Tet的关联
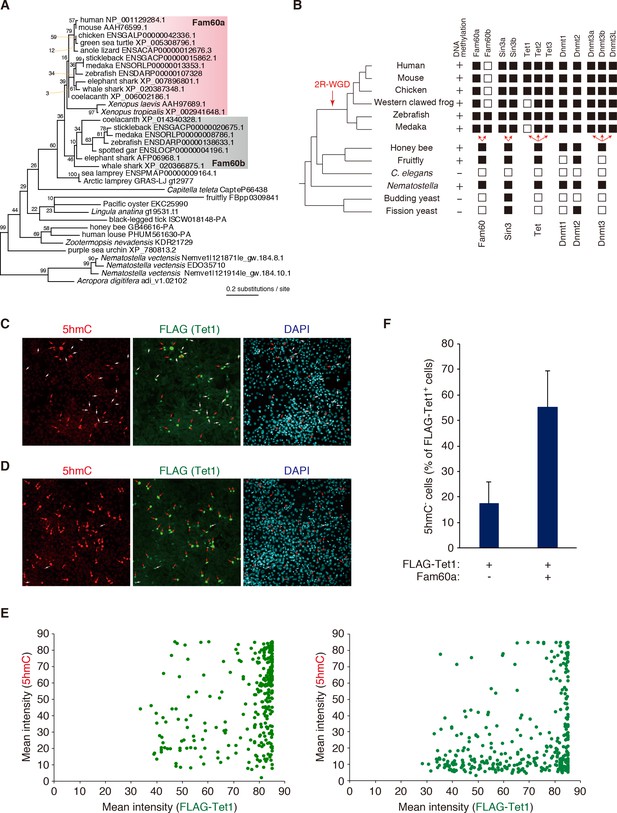
Fam60a和Tet1的系统发育和功能关系。
异常启动子低甲基化 Fam60a系列 –/– 小鼠胚胎
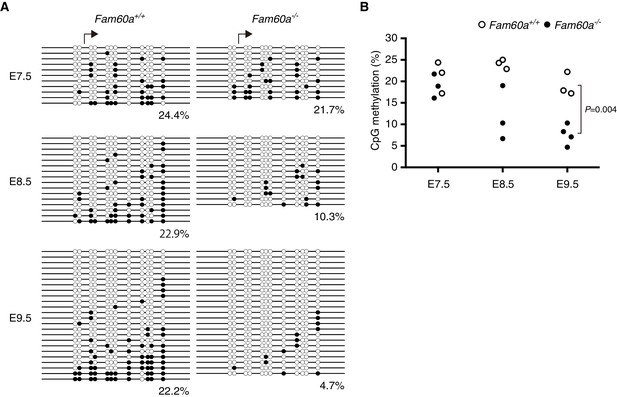
甲基化状态 Adhfe1公司 野生型和 Fam60a系列 –/– 小鼠胚胎。
基因组中不同的甲基化区域 Fam60a系列 –/– 胚胎
高甲基化和低甲基化DMR的数量与ChIP-seq峰重叠。
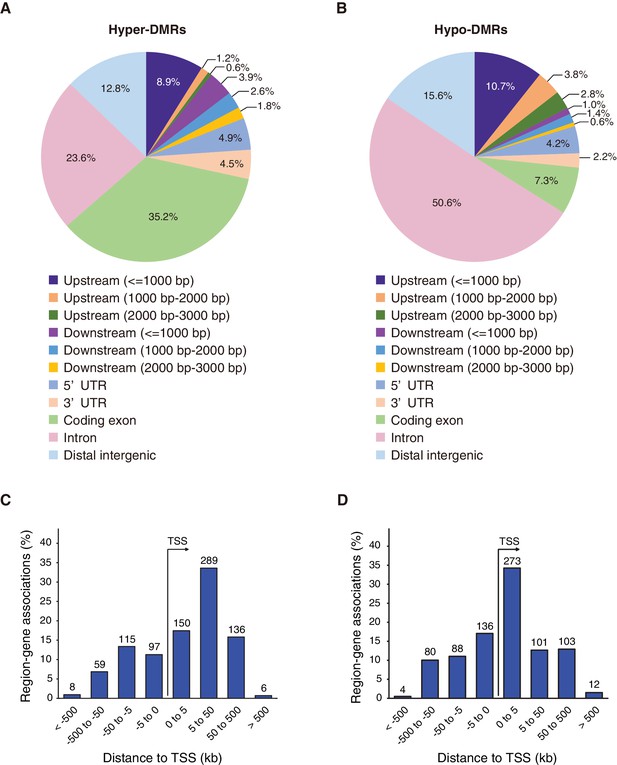
不同甲基化区域(DMRs) Fam60a系列 –/– 胚胎。
-
图8-源数据1 低甲基化和高甲基化DMR列表。 DMR按列areaStat排序,该列是CpG的t-统计量之和。 请注意 Adhfe1公司 被包括在前500个低甲基化DMR中。 -
https://doi.org/10.7554/eLife.36435.047
讨论
材料和方法
老鼠
细胞系来源和验证
Fam60a-相互作用蛋白的鉴定
免疫沉淀和免疫印迹分析
世系追踪
原位杂交和组织学
抗体
胚胎免疫染色
BrdU掺入分析
RT-qPCR分析
ChIP-seq和RNA-seq
亚硫酸氢盐测序
Fam60a和FLAG-Tet1的强制表达
ChIP-qPCR
hMeDIP分析
甲基seq文库建设
甲基seq和DMR的检测
分子系统发育学
数据可用性
统计分析
数据可用性
-
Fam60a是mSin3A-HDAC转录共抑制复合物的组成部分,抑制Tet-介导的DNA去甲基化 在NCBI BioProject上公开提供(注册号:PRJDB4950)。 -
Fam60a是mSin3A-HDAC转录共抑制复合物的组成部分,抑制Tet-介导的DNA去甲基化 可在NCBI BioProject上公开获取(注册号:PRJDB6732)。
工具书类
-
ING2以不同于组蛋白H3赖氨酸4和9的甲基化位点特异性招募组蛋白甲基转移酶活性 生物化学与生物物理学报(BBA)-分子细胞研究 1783 :1673–1680. https://doi.org/10.1016/j.bbamcr.2008.04.019 -
Bismark:一种适用于亚硫酸氢盐-Seq应用的灵活对准器和甲基化调用者 生物信息学 27 :1571–1572. https://doi.org/10.1093/bioinformatics/btr167 -
作为mSin3A/p33(ING1b)/HDAC1脱乙酰酶复合物成分的新型BRMS1同源蛋白p40的鉴定 生物化学和生物物理研究通讯 323 :1216–1222. https://doi.org/10.1016/j.bbrc.2004.08.227 -
一种在成熟卵母细胞中保持Cre重组酶活性的转基因小鼠系,与Cre转基因传递无关 生物化学和生物物理研究通讯 237 :318–324. https://doi.org/10.1006/bbrc.1997.7111 -
序列相似的人类家族60成员A(FAM60A)蛋白:Sin3脱乙酰酶复合物的一个新亚单位 分子和细胞蛋白质组学 11 :1815–1828. https://doi.org/10.1074/mcp.M112.020255 -
Fam60a定义了胚胎干细胞自我更新所需的变异Sin3a-Hdac复合物 欧洲分子生物学组织 36 :2216–2232. https://doi.org/10.15252/embj.201696307
文章和作者信息
作者详细信息
基金
教育、文化、体育、科学和技术部(25251029)
滨田浩史
鸣谢
伦理学
版本历史记录
收到日期:2018年3月6日 接受日期:2018年7月10日 发布的记录版本: 2018年8月2日(第1版)
版权
度量标准
-
1,550 意见
-
169 下载
-
9 引用
下载链接
进一步阅读
-
计算与系统生物学 发育生物学
TALK-1 K基因的获得功能突变 + 通道(p.L114P)与年轻人成熟型糖尿病(MODY)相关。 TALK-1是β细胞电活动和葡萄糖刺激胰岛素分泌的关键调节器。 这个 KCNK16型 编码TALK-1的基因是最丰富的β-细胞限制性钾 + 通道转录本。 调查 KCNK16号机组 L114P对葡萄糖稳态的影响,并证实其与MODY(一种含有 Kcnk16号机组 产生L114P突变。 杂合和纯合 Kcnk16号机组 L114P小鼠在C57BL/6J和CD-1(ICR)基因背景下的新生儿死亡率分别增加。 致死率可能是纯合子严重高血糖的结果 Kcnk16号机组 L114P新生儿由于缺乏葡萄糖刺激的胰岛素分泌,可以通过胰岛素治疗减少。 Kcnk16号机组 L114P增加了全细胞β细胞K + 导致葡萄糖刺激钙钝化的电流 2+ 葡萄糖诱导的钙的进入和损失 2+ 振荡。 因此,成年人 Kcnk16号机组 L114P小鼠降低了葡萄糖刺激的胰岛素分泌和血浆胰岛素水平,这显著损害了葡萄糖稳态。 综上所述,本研究表明,MODY与 Kcnk16号机组 L114P突变破坏了类似MODY表型的成年小鼠的葡萄糖稳态,并在发育过程中通过抑制胰岛胰岛素分泌导致新生儿死亡。 这些数据表明,TALK-1是治疗糖尿病的一个胰岛受限靶点。 -
发育生物学 结构生物学和分子生物物理学
有性生殖中的一个关键事件是单倍体精子和卵子在受精时融合形成一个新的二倍体生物。 在哺乳动物中,卵子JUNO和精子IZUMO1之间的直接相互作用介导配子膜粘附,但它们在融合中的作用仍然是个谜。 我们使用AlphaFold预测其他对受精至关重要的细胞外蛋白的结构,以确定它们是否可以形成一种可能介导融合的复合物。 我们首先鉴定了TMEM81,其基因由小鼠和人类精子细胞表达,该蛋白与IZUMO1和另一种配子融合所必需的精子分子SPACA6具有结构同源性。 然后,我们使用一组已知对受精和TMEM81很重要的蛋白质,采用非指导性方法系统地搜索预测的二元相互作用,并确定了涉及精子IZUMO1、SPACA6、TMEM81和卵子JUNO、CD9的五元复合物。 该复合物在结构上与对侧配子膜上的预期拓扑结构和未用AlphaFold-Multimer建模的预测N-聚糖的位置一致,表明其成分在融合点可以组织成突触样集合。 最后,这里描述的结构建模方法可能更普遍地有助于深入了解难以实验检测的瞬时蛋白质复合物。






