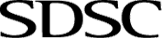介绍
生物分子的功能遵循其形式(或形状)。这反过来意味着具有相似形状或结构的分子具有相似的功能。蛋白质数据库(PDB)中的结构数量、大小和实验结构的复杂性每年都在增长。许多实验结构是多个蛋白质或蛋白质的多个拷贝的集合。装配坐标可以是模型的特定子集或放置坐标,也可以通过应用特定类型的对称操作导出。查询沉积坐标和组装坐标使得寻找结构相似的蛋白质和组装具有挑战性。
RCSB.org还提供了超过一百万个计算结构模型(CSM)的访问权限。这些模型的坐标不包含任何与对称相关的信息,因此模型和装配坐标相同,默认情况下包含在基于结构的搜索中。
什么是结构相似性搜索?
结构相似性搜索选项允许您使用蛋白质结构的3D形状查询PDB存档。该RCSB PDB开发方法(Guzenko等人,2020年)将蛋白质视为原子填充的空间体积(即密度分布),而不是原子坐标和链连接性的集合。蛋白质体积使用称为3D泽尼克多项式的数学工具进行分解,并被描述为泽尼克矩的向量。这种方法有助于使用对旋转和平移不变的紧凑描述符来描述体积(Novotni和Klein,2004年). 该搜索使用BioZernike描述符来评估全局3D形状的相似性,以捕获蛋白质的全局体积形状,并且对于单个蛋白质链和组装体都非常快速。
为什么运行结构相似性搜索?
发现和分类PDB中的结构是理解功能和进化关系的基础。虽然基于序列的搜索可以揭示蛋白质中的保守结构域,但在生物学中有许多例子表明,尽管序列发生了变化,但蛋白质的形状(和功能)是相似的。此外,有时同一蛋白质可能采用一种以上的构象,例如酶的开放和封闭形式。使用基于序列的搜索无法识别这些结构,并且需要结构相似性搜索选项。
此外,一些蛋白质是稳定的和/或作为组装的一部分发挥作用的,在组装中它与自身或其他蛋白质的一个或多个拷贝相互作用。结构相似性搜索选项允许您识别相似的集合,从而可以探索蛋白质(或其复合物)的形状和相互作用。
文档
有几个不同的选项可以组合起来运行基于结构或形状的搜索。这些选项列在此处的查询和结果部分下:
- 查询-这将描述输入查询形状的选项(例如,链或组件);定义搜索类型(例如,严格和宽松);并指定用于查找匹配的结构层次结构(即链或程序集)
- 结果-这将描述您希望在结果页面中看到的可用选项
查询选项
可以对类似以下形状启动结构相似性搜索:
- 给定的聚合物链ID-指定的形状可以与聚合物链(默认)或组件(需要指定)匹配
- 给定的程序集ID-指定的程序集可以与另一个程序集匹配(默认)或聚合物链(需要指定)
对于任何结构相似性搜索,可以使用下拉菜单在两种匹配模式之间进行选择:
- 严格:如果你想确保所有匹配项都是相关的,就使用这个选项,这样可能会导致找不到更遥远的匹配项
- 放松:如果你想确保你的匹配包含所有类似的结构,就使用这个,这样可能会带来一些假阳性
请注意,虽然可以为从“高级搜索”面板启动的结构相似性搜索选择严格或宽松选项,但从“结构摘要页面”启动的搜索会自动选择严格搜索选项。
除了指定查询形状外,还有一些选项可用于指定与输入形状进行比较的对象。这些选项包括:
- 程序集:使用它来匹配查询以完成程序集(如果您对复杂的整体形状感兴趣,但对其组成不感兴趣,则此项很重要)
- 链:使用此选项将查询与蛋白质结构的单个链匹配(如果您希望链是更大复合体的一部分,则此选项特别有用)
虽然会自动设置合理的默认值,即使用程序集ID定义的搜索将搜索程序集,而按链ID搜索将搜索链,但如果查询没有返回任何结果或预期结果,则可能需要更改这些选项。
注意:“Search for”选项独立于“Return”选项,后者可以在Advanced Query Builder的左下角指定。“搜索”选项决定查询找到的结构,而“返回”选项则改变结果的显示方式。
可以为RCSB.org中可用的结构定义查询,也可以为RCSB.org中不可用的结构定义查询。
可从RCSB.org查询结构
这些结构的聚合物链和基于组装的结构相似性搜索都可以从-(a)高级搜索面板和(b)3D结构的结构摘要页面启动。
使用“高级搜索”面板进行查询
结构相似性搜索选项可从“高级搜索”面板获得,可以通过在“结构相似性”下列出的框中键入PDB ID或RCSB.org分配的CSM ID来访问(图1)。
|
| 图1:从高级搜索查询生成器启动结构相似性搜索的选项。 |
一旦在框中键入3D结构ID(实验结构的PDB ID或RCSB.org指定的CSM ID),一些附加选项就可用了。这些选项允许您通过选择合适的组件ID或链ID和搜索层次来指定查询形状,即搜索匹配的组件或聚合物链。
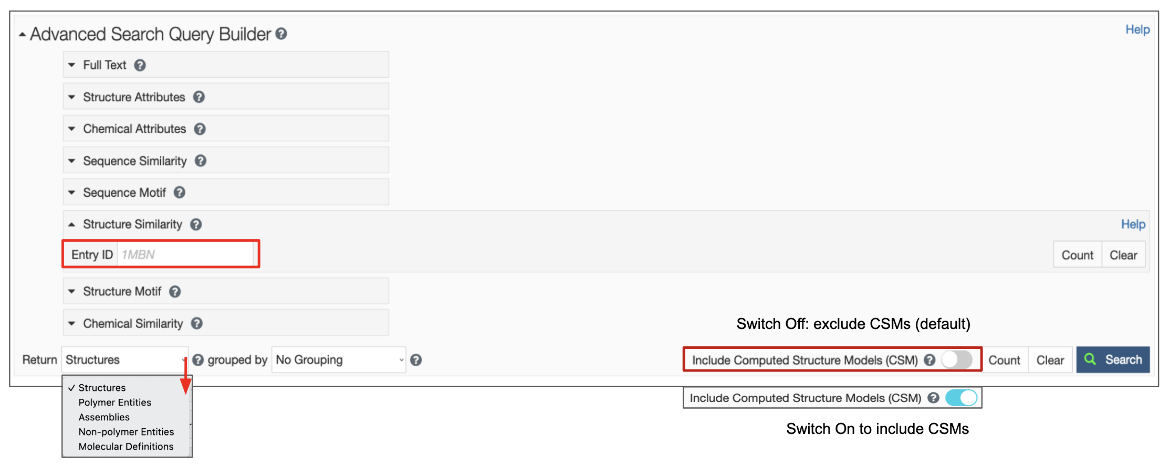
通过上传PDB ID并选择程序集ID指定查询时,可以使用以下选项:
- 默认情况下,从“搜索”下拉菜单中默认选择“装配”选项。决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮以启动搜索。预计您还将在结果“Return”选项中选择“Assemblies”(装配)(图2A)。可酌情选择其他选项。
- 如果您希望找到与指定组件匹配的聚合物链,请在“搜索”下拉选项中选择“链”,然后选择适当的“返回”选项。(请参见例子)
如果RCSB.org分配的CSM ID用于此搜索,请记住打开Include CSM toggle开关(见图2B)。请注意,对于CSM,装配坐标与模型坐标相同,因此装配表示为存放的装配。
|
| 图2:指定结构相似性搜索的选项-a.使用PDB ID和程序集ID并决定是否包含或排除CSM;B.使用RCSB.org分配的CSM ID,打开Include CSM toggle switch。在这两种情况下,在启动搜索之前,将结果Return类型指定为Assemblies。 |
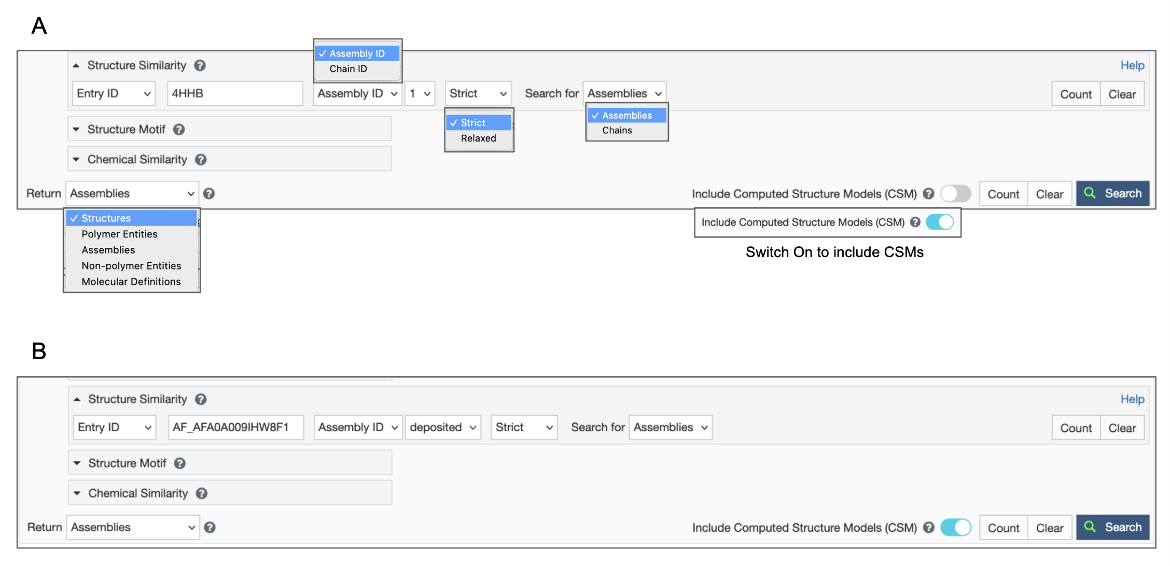
通过上传PDB ID并选择链ID指定查询时,可以使用以下选项:
对于基于蛋白质链的结构相似性搜索,在查询结构中选择感兴趣蛋白质的链ID,在结果返回选项中选择“Polymer entities”,决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮启动搜索(图3A)。如果此搜索使用CSM ID,请记住打开Include CSM toggle开关(见图3B)。
- 默认情况下,从“搜索”下拉菜单中默认选择选项“链”。决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮以启动搜索。预计您还将在结果“返回”选项中选择“聚合物实体”(图3A)。可酌情选择其他选项。
- 如果您希望找到与指定聚合物链匹配的组件,请在“搜索”下拉选项中选择“组件”,然后选择适当的“返回”选项。(请参见例子)
如果RCSB.org分配的CSM ID用于此搜索,请记住打开Include CSM toggle开关(见图3B)。
|
| 图3:指定结构相似性搜索的选项-a.使用PDB ID和链ID并决定是否包含或排除CSM;B.使用RCSB.org分配的CSM ID,打开Include CSM toggle switch。在这两种情况下,在启动搜索之前,将结果返回类型指定为“聚合物实体”。 |
从“结构摘要”页面查询
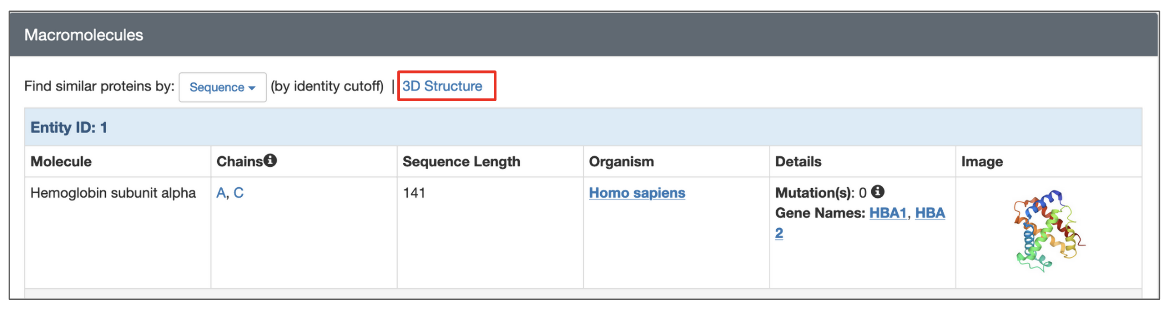
RCSB.org上提供的所有3D结构(实验结构和CSM)都有一个专用的结构摘要页面,其中显示了该条目实体和组件的信息。要搜索与结构中任何一个聚合物实体相似的结构,请单击高分子详细信息上方的“结构”链接(图4)。
|
| 图4:从结构摘要页面(在红色框中突出显示)启动基于结构的搜索的选项。 |
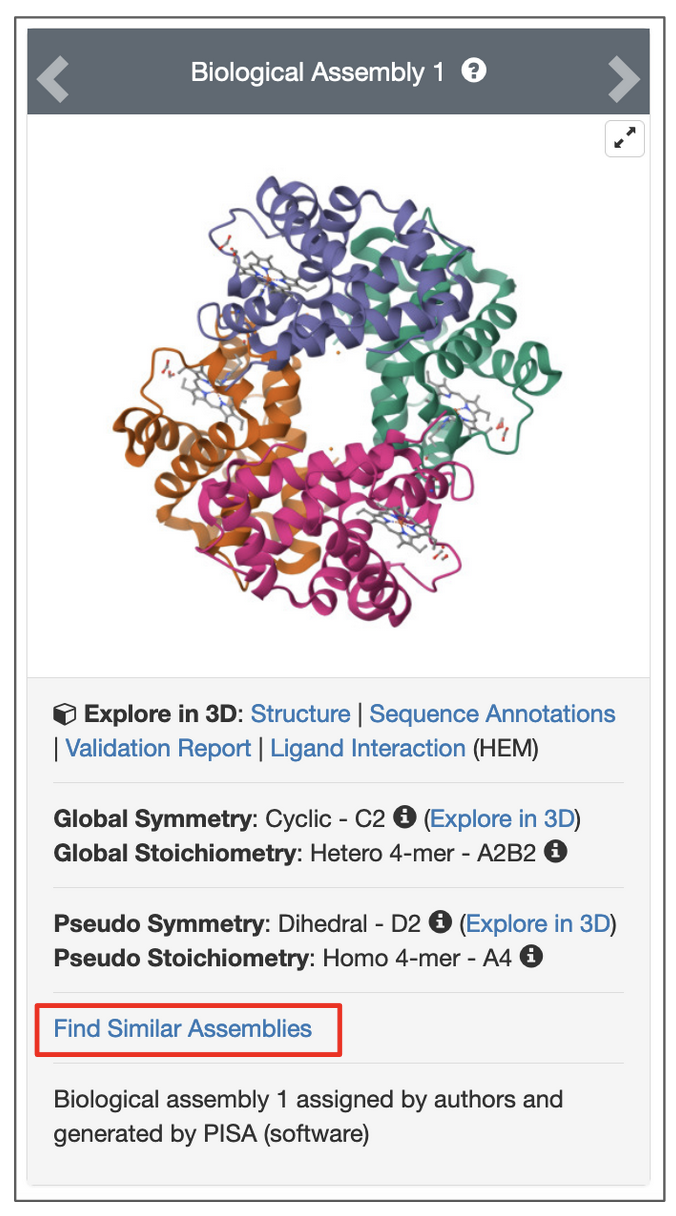
要搜索与结构的特定程序集类似的程序集,请单击页面上程序集快照下方的“查找类似程序集”链接(图5)。
|
| 图5:从结构摘要页面启动程序集搜索的选项。单击红色框中突出显示的链接。 |
RCSB.org上没有结构查询
查询其他公共数据资源中可用的结构
此选项可用于查找与RCSB.org以外的公共数据资源中包含的3D结构类似的结构,例如AlphaFold、RoseTTAFold或ESMFold预测。可以使用URL输入查询结构来构造此查询。
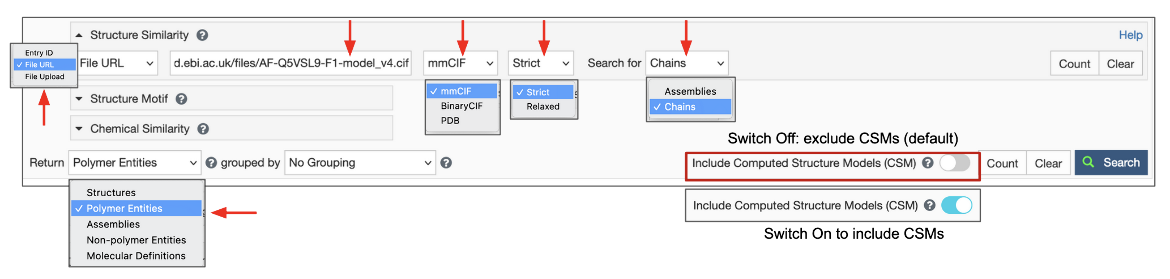
要使用此功能,请打开高级搜索查询生成器并滚动到“结构相似性”选项。将输入模式从“条目ID”切换到“文件URL”(图6)。确保将URL指定为“http”或“https”协议。指定文件格式,默认为mmCIF,但也支持BinaryCIF和PDB文件。根据需要,在结果返回选项中选择“聚合物实体”或“结构”。决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮以启动搜索
搜索将基于放置的坐标,也称为“非对称单元”。注意:这与基于3D实验或CSM条目ID的查询不同,后者允许您选择特定的程序集或链标识符进行搜索。
注意:在具有局部低置信区域的CSM结构中,即对于来自AlphaFold、RoseTTAFold、ESMFold的CIF文件,其中存在“ma_qa_metric_local”CIF类别,并且本地pLDDT分数小于70,则应用预筛选步骤从查询中删除这些区域。排除这种非结构化或高度灵活的CSM区域可以减少查询结果中的误报和漏报数量。
|
| 图6:使用文件URL指定非RCSB.org 3D结构作为查询的结构相似性搜索选项。 |
查询本地驱动器上可用的结构
此选项可用于通过上传您自己的本地文件来指定自定义查询,以搜索与文件中分子形状类似的结构。
要使用此功能,请将输入模式切换为“文件上载”。这将为您提供菜单,允许您从文件系统中选择文件(图7A)。支持扩展名为“.cif”、“.bcif”,“.pdb”和“.ent”的文件及其gzipped(“.gz”)变体。选择文件后,它将自动上传到我们的服务器,输入模式将切换到“文件URL”(图7B)。您的文件将被一个唯一的URL引用。其他用户无法猜测这个随机URL;但是,请注意,任何知道此URL的人都可以访问您的数据。
上传的文件将在90天内可用,这意味着您可以在有限的时间内为搜索添加书签或与同事共享。如果你想在出版物、博客文章或类似文章中持续引用搜索,你应该将文件上传到Dropbox或Google Drive等文件共享服务,并使用“文件URL”功能引用它。存储在MyPDB中的查询也是如此。支持的最大文件大小为10 MB,较大的文件还需要使用外部文件共享服务。

|
| 图7:结构相似性搜索选项定义为(A)从本地驱动器上传文件到(B)创建临时web链接。 |
注意:上传的文件的行为与URL引用的外部文件类似,对于处理pLDDT置信度较低的程序集和区域,也存在相同的限制。
结果
根据选定的选项,结构相似性搜索结果会列出类似的实体或程序集。
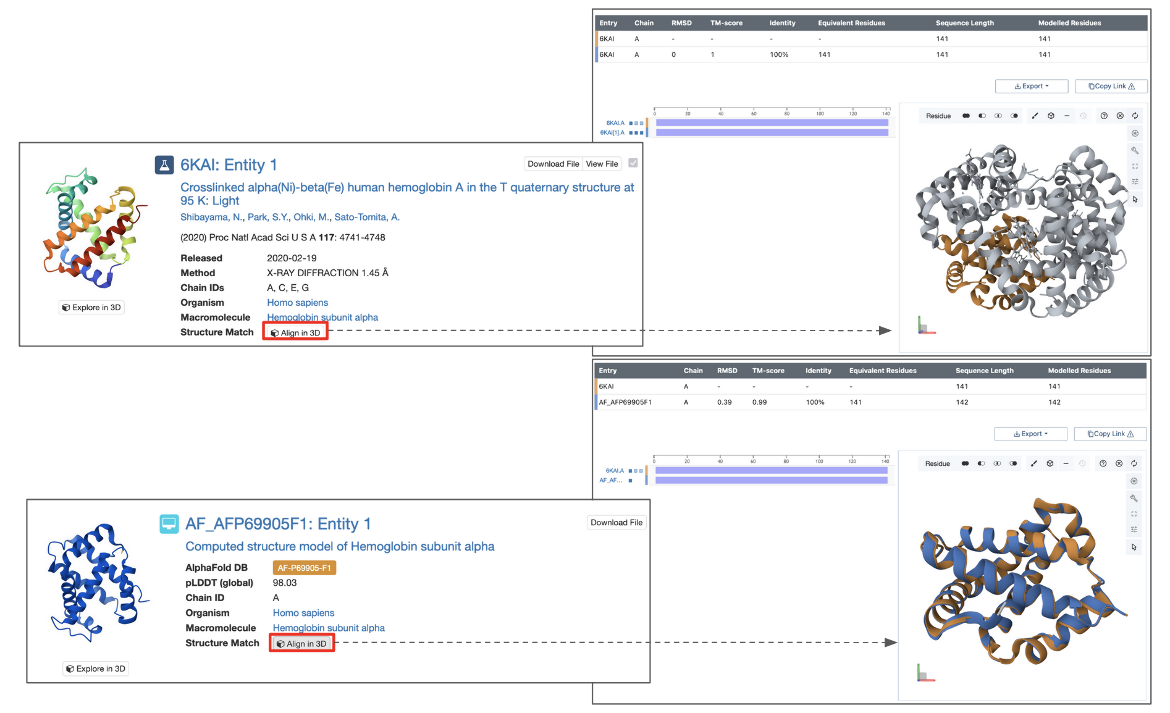
对于基于实体的搜索,可以将每个匹配的实体叠加在查询实体上,并通过单击“结构匹配”旁边的视图按钮,使用成对对齐工具在3D中查看(图8)。
注意:“视图”按钮仅在使用3D实验或基于CSM条目ID的查询搜索结构时可用。
|
| 图8:查询结果页面的一部分,显示了查看结构匹配的选项(右侧的面板)和描述匹配程度的一些度量(图顶部和底部的红色轮廓框)。 |
对于基于程序集的搜索,每个匹配的程序集都会被分配一个结构匹配分数,以匹配查询结构的概率百分比表示。因此,分数为100表示完全匹配,而数字越小表示组件中的相似程度越低(图9)。

|
| 图9:基于装配的匹配结果列表的一部分,显示了结构匹配得分 |
注意:使用“文件URL”或“文件上载”选项上载的搜索结果被视为基于程序集的搜索。由于这些搜索返回的结果是程序集,因此会报告“结构匹配分数”。
结构相似性搜索的局限性
结构相似性搜索系统有一些局限性:
- 该方法无法报告RMSD,因为它只生成体积的全局最优叠加,而不了解路线中成对的残差。相反,该方法输出一个分数,指示匹配的可能性。
- 高度对称的程序集通常会产生误报(分数较低),例如,搜索D3点组对称程序集可能会匹配一些分数较低的无关D3程序集。
- 由于长而灵活的尾部,柔性核磁共振结构通常是不匹配的
- 长而突出的尾巴将导致无法匹配全球相似的形状。
- 匹配是全局的,因此没有发现局部相似性。例如:
- 搜索链时:仅在某些公共域中相似的两条链通常不匹配,
- 搜索程序集时:在链的某些子集中相似但不全局的两个程序集通常不匹配。
示例
1.搜索与肌红蛋白类似的实体
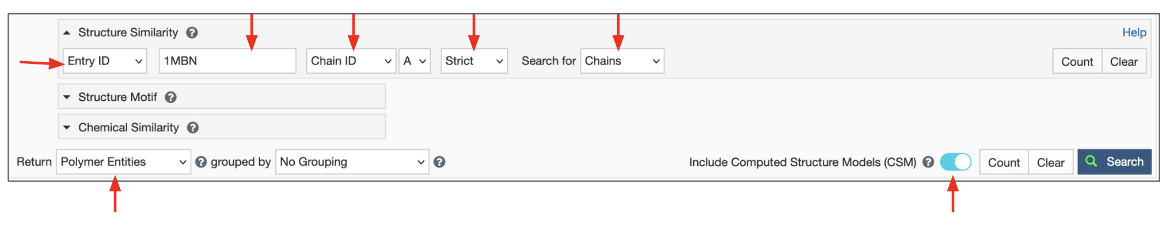
- 从PDB ID 100亿、链ID A的高级搜索界面启动此搜索
- 选择strict search选项,Display results as Polymer Entities,include CSM,然后启动搜索(图10)
|
| 图10:在PDB条目100亿中运行基于结构的链ID a搜索以返回聚合物实体的选项。搜索包括CSM。 |
- 这个搜索结果显示许多肌红蛋白实体,一些血红蛋白实体,一些脑红蛋白和其他实体。
2.搜索构象类似己糖激酶开放形式的实体
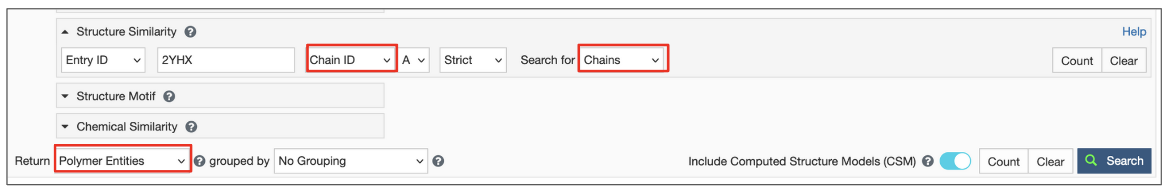
- 使用“开放”构象中己糖激酶的结构作为查询。从PDB ID 2yhx、Chain ID A的Advanced search界面启动此搜索(图11)
- 选择严格搜索选项,将结果显示为聚合物实体,包括CSM,然后启动搜索。
|
| 图11:搜索构象类似己糖激酶开放形式的结构的选项 |
- 这个搜索结果显示其他己糖激酶和相关蛋白。请注意,较好的匹配是具有开放构象的己糖激酶实体,而结果列表末尾列出的匹配包括闭合构象中的相同或相关酶实体。
3.搜索与SARS-CoV-2 Spike蛋白三聚体类似的组装体
- SARS-CoV-2尖峰蛋白由三条聚合物链组成,每条聚合物链都有一个受体结合域,该域可以是开放(或向上)构象,用于与细胞受体或封闭(或向下)构象相互作用。结构相似性搜索功能可用于识别具有这些域类似排列的尖峰结构。
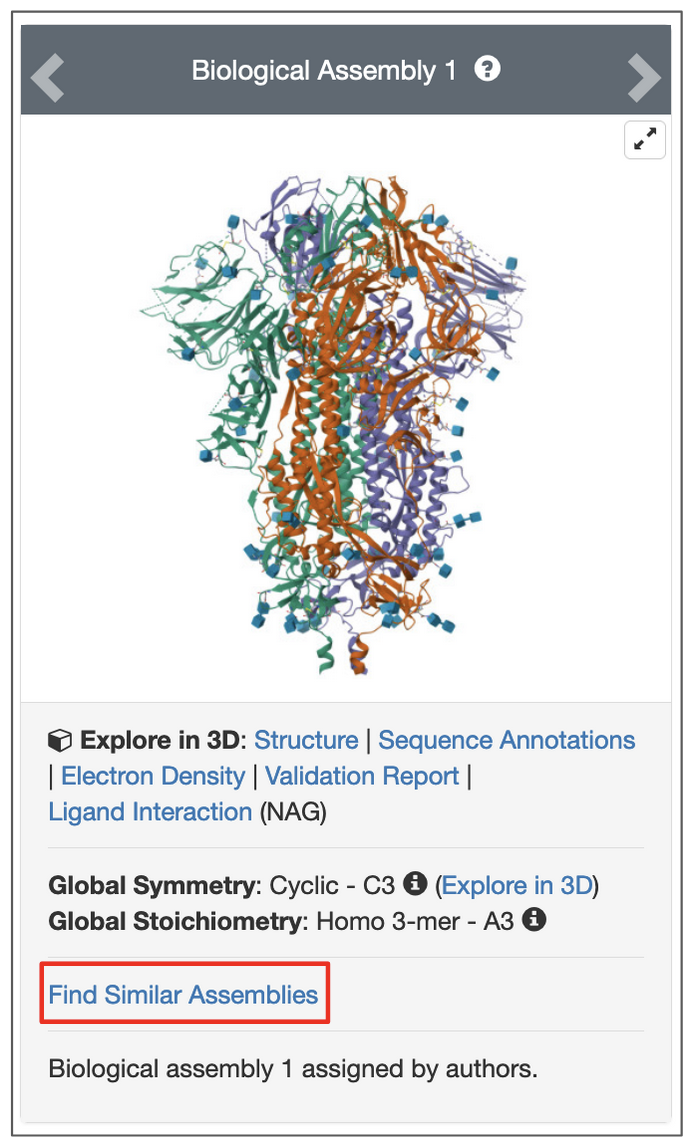
- 要找到所有三个受体结合结构域都闭合的刺突结构,请从结构摘要页面启动PDB ID 6vxx,生物组件1的结构相似性搜索(图12)。
|
| 图12:PDB ID 6vxx的结构摘要页面中搜索具有相同组件的结构的选项。 |
- 这个搜索结果显示具有闭合构象的类似spike蛋白组合。
4.寻找与胰岛素六聚体类似的组装物
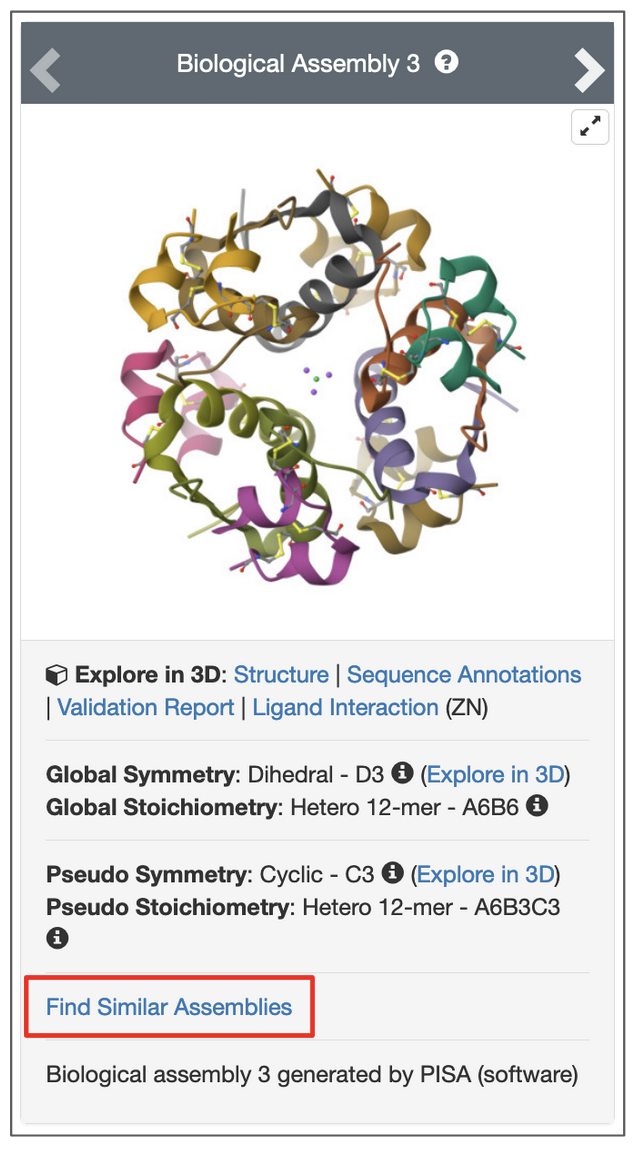
- 从Structure Summary页面启动此搜索,查找PDB条目1trz,Biological Assembly 3(图13)
|
| 图13:从PDB ID 1trz的结构摘要页面启动基于结构(组件)的搜索的选项。 |
这个搜索结果显示了许多其他类似的胰岛素组件,以及约12%的结构匹配分数的一些不相关的结构。
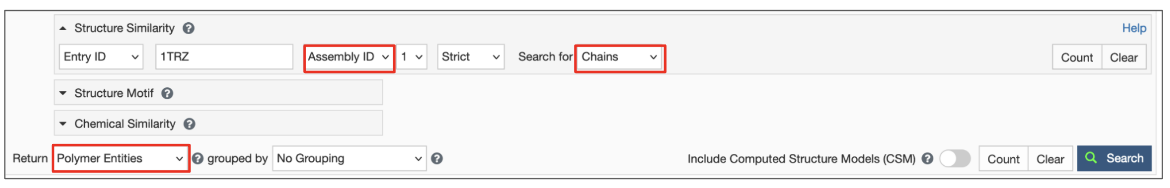
5.寻找形状类似成熟胰岛素的单链胰岛素(由两条聚合物链组成)
- 从PDB ID 1trz、Assembly ID 1的Advanced search界面启动此搜索
- 在“搜索”下拉菜单中选择严格搜索选项和链选项
- 将结果(即设置“返回”选项)显示为聚合物实体。
|
| 图14:高级搜索查询生成器选项,用于搜索与PDB条目1trz中的程序集匹配的单链。 |
这个搜索结果显示了一些单链胰岛素分子。
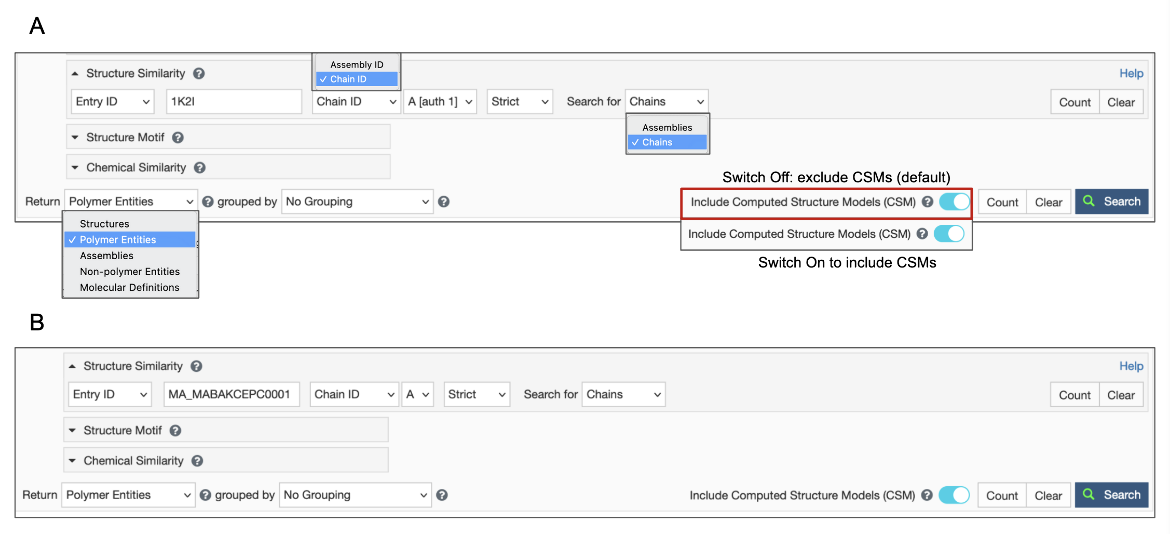
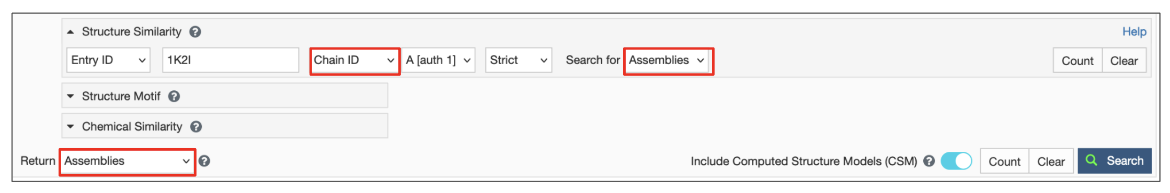
6.寻找与胰凝乳蛋白酶聚合物类似的组装物
- 从PDB ID 1k2i、Chain ID A的高级搜索界面启动此搜索
- 在“搜索”下拉菜单中选择严格搜索选项和装配选项
- 将结果(即设置“返回”选项)显示为程序集,包括CSM,然后启动搜索。
|
| 图15:Advanced Search Query Builder选项,用于启动对PDB条目1k2i中链接的程序集的搜索。 |
这个搜索结果显示了许多其他糜蛋白酶结构,一些是单链,一些是组装体(由多个聚合物链组成),但与查询中指定的整体形状匹配。
工具书类