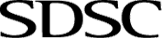介绍
什么是结构主题?
结构基序是少量具有重要意义的氨基酸(至少2个)的空间或三维排列-例如。,形成催化或结合位点。构成基序的氨基酸残基在1D序列中可能彼此远离,甚至可能位于不同的聚合物链中,只要它们在3D空间中彼此靠近(彼此之间在20Å以内)。结构主题搜索服务(Bittrich等人,2020年)检索可从RCSB.org获得的3D结构中出现的所有特定结构基序。
此处以烯醇化酶超家族的活性位点为例(图1,Meng等人,2004年). 烯醇化酶超家族是一组序列不同但在3D结构上大体相似的蛋白质,所有这些蛋白质都催化从羧酸中去除质子(巴比特等人,1996年).
|
| 图1:这里显示了代表烯醇化酶超家族的五个残基。注意,在其中三个位置可以看到多种氨基酸。氨基酸通过其氨基酸名称3个字母的缩写、链ID(label_asym_ID)和残基编号(label_seq_ID)进行识别。 |
此搜索何时有用?
当您对探索蛋白质结构的局部结构特性感兴趣时,结构基序搜索服务特别有用。该搜索服务补充了结构搜索服务,并发现蛋白质之间的局部结构相似性。搜索结果仅依赖于查询中指定的残基,因此即使蛋白质的序列或整体结构相似性有限,它也可以识别局部结构相似性。因此,例如,这种搜索可以在无关蛋白质中找到类似的配体结合位点,而不管这些结构是否在该邻域中有配体结合。
对这种结构基序的检测可以对以前未经特征化的蛋白质的功能提供有价值的见解,特别是那些在序列或整体结构水平上与其他蛋白质不相似的蛋白质。
文档
结构模体搜索服务可通过Mol*界面访问,查询残基(氨基酸和核苷酸)可在可视化分子结构中指定'高级搜索'面板中,可以通过在界面中键入结构文件标识符或位置和查询残留物详细信息来指定它们。
使用Mol定义查询*
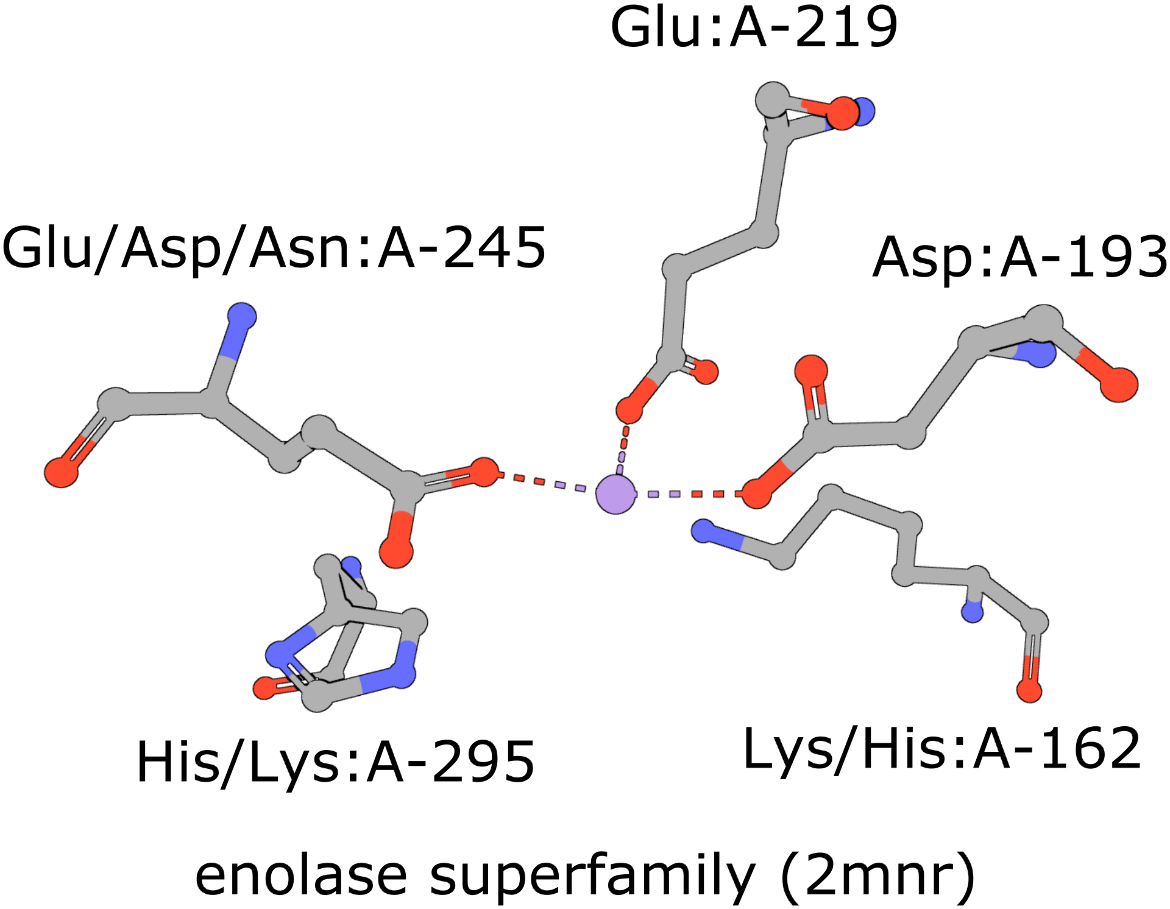
RCSB-Mol*插件提供了一种方便的方式来可视化结构和定义结构主题查询。一般Mol*文档可以找到在这里这里描述了指定结构motif查询(图2)的步骤。
|
| 图2:Mol*用户界面与Structure Motif Search面板展开。 |
定义基于扁桃酸消旋酶(PDB ID)的烯醇化酶超家族的结构基序查询2个)并使用中描述的模板(孟,2004)使用以下步骤。
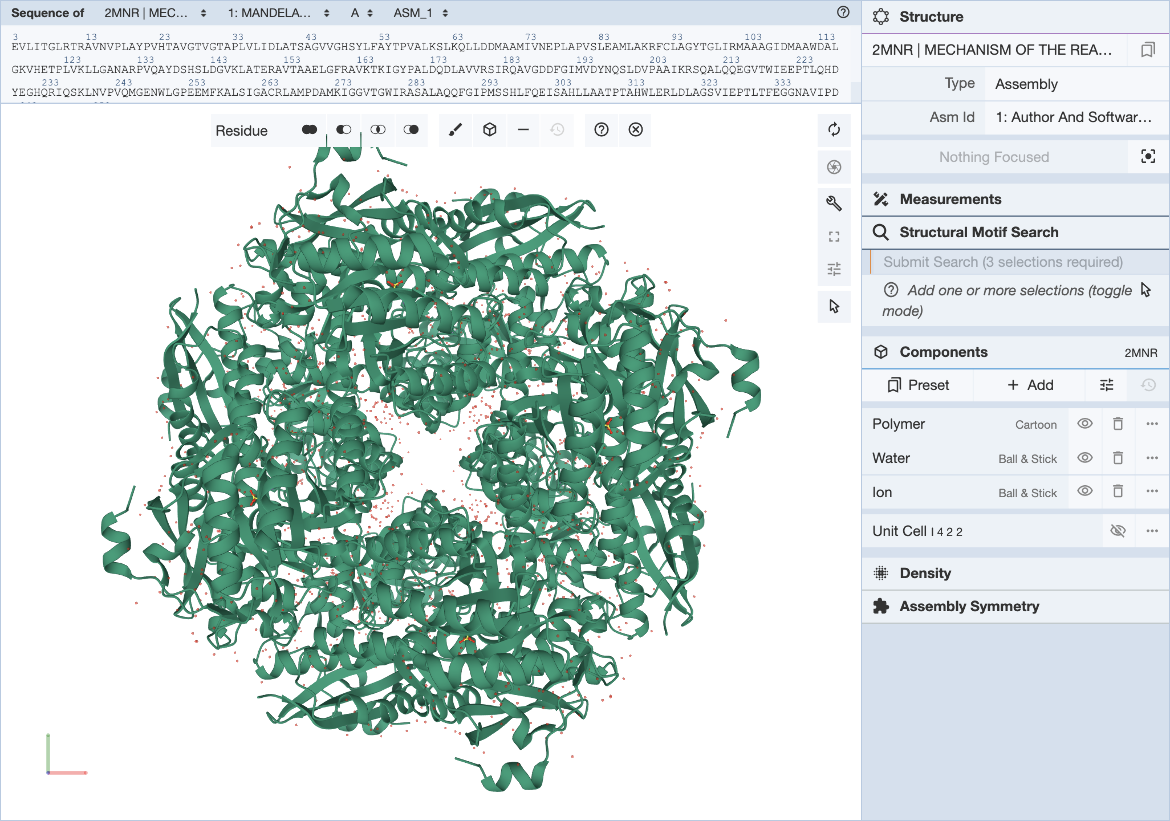
在Mol*界面中,单击并展开右侧控制面板中的“Structure Motif Search”菜单。通过单击鼠标指针图标激活Mol*的选择模式,并将选择级别设置为“残留物”(默认值)。这允许您选择将定义查询的单个残留物(图3)。
|
| 图3:选择motif的Mol*用户界面。 |
本文使用文献中描述的构成模板的5个残基来定义查询基序。
通过在3D画布或序列面板中单击单个残留物来选择它们。所选残留物将填充在控制面板的“结构基序搜索”列表中。此列表中最多可包含10个残留物。通过单击其他残留物添加到选择,或通过单击残留物列表中的垃圾图标删除残留物。Mol*的“Structure Motif Search”元素的行为类似于“Measurements”面板。
将鼠标悬停在感兴趣的剩余部分上以进行验证标签_符号_id和标签_设备id。信息将显示在Mol*面板右下角的工具提示中。如果标签和作者标识符不同,作者定义的链ID和残数将显示在方括号中。当通过作者编号选择残基时,顶部的序列视图特别有用(图4)。之间的差异标签_设备id和授权seq_id将以方括号中的Mol*表示。学习有关PDB中标识符的更多信息.
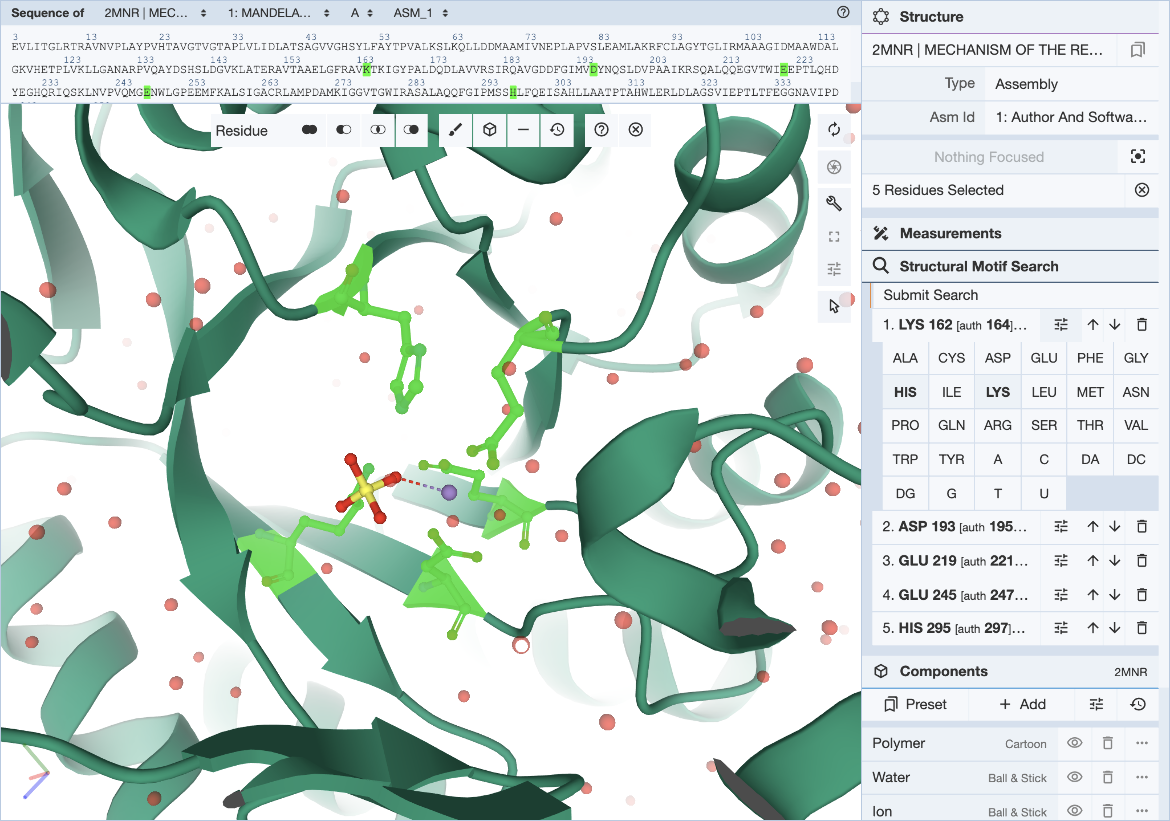
|
| 图4:Mol*用户界面,交换面板已展开并指定了选项。 |
如果一系列氨基酸(或核苷酸)可能实现相同的生物功能或结合相同的配体,则可以在查询中定义特定位置的交换,以适应查询结构基序特定位置的可能变化。
对于剩余列表的每个条目,可以通过单击选项图标(三个带有短垂直线的水平条与它们相交)分别指定交换。这将打开一个包含20个氨基酸和8个核苷酸名称的面板。单击应视为相应位置有效交换的所有三个字母代码。如果未定义交换,则只有原始剩余类型有效。定义其他交换时,请确保包括原始残留物类型。每个职位的交换次数限制为4次。
单击“提交搜索”按钮。这将打开一个新的浏览器选项卡,您的查询将显示在“高级搜索”面板.
除了使用链接到RCSB.org三维结构的结构摘要页面的Mol*可视化选项外,Mol*独立工具中还提供了文件上传功能(/三维视图). 从本地驱动器上传结构文件或指定URL后,可以如上所述定义结构主题查询。Mol*将检测您的文件是否是存档结构(并使用其条目ID引用它)、使用外部URL加载的结构(并通过该链接引用它),或者您是否正在可视化本地文件(在这种情况下,您的文件将上载到我们的服务器),并将适当的ID/链接保存到“高级搜索”面板.
使用“高级搜索”面板定义查询
也可以直接在“高级搜索”面板。有几个不同的选项可用于定义查询。
使用RCSB.org提供的结构ID
要使用此选项,您可以在“条目ID”框中输入结构ID(PDB ID或RCSB.org分配的CSM ID)(图5)。旁边会出现一个新按钮,名为“打开摩尔*”。点击此按钮可打开Mol*中的结构,允许您选择查询残留物(如上所述)。使用选项选择残留物,使用Mol*向导自动填充“高级搜索”面板。该面板还为您提供了手动验证、优化或扩展搜索的机会。
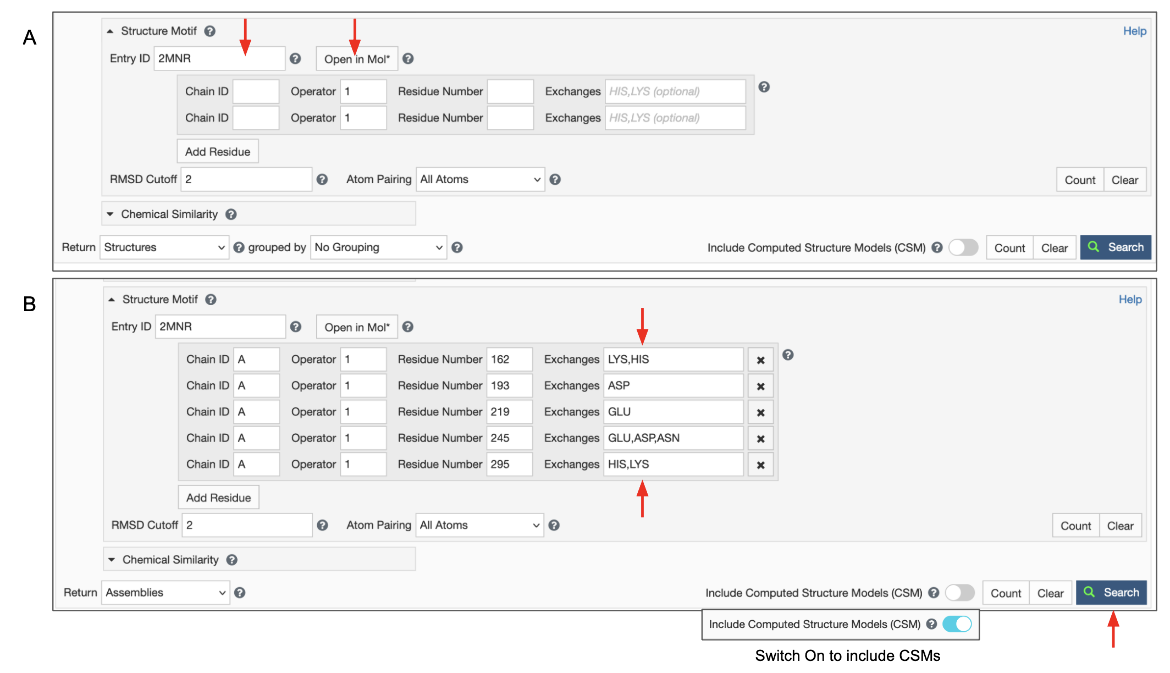
|
| 图5:显示Structure Motif搜索面板的Advanced Search面板。A.输入条目ID后,出现“Open in Mol*”按钮(用红色箭头标记)。B.结构主题搜索面板由Mol*自动填充并手动扩展。 |
注:再次单击“在分子*中打开”按钮(即,在指定了残留物选择后)将在分子*内打开结构,并仅显示“结构主题搜索”面板中列出的残留物。注:要优化残基选择,请更多地打开选择并选择显示的所有氨基酸。现在,使用“控制”面板中的选项显示聚合物,并选择其他(或更少)残留物来修改/完善查询,然后单击“结构主题搜索”>>“提交搜索”按钮。注:如果手动扩展高级搜索面板以包含特定位置的替代残留物,“在分子中打开*”按钮将仅显示结构中存在的残留物。面板中列出的可能备选方案将不会显示,因为这些坐标在可视化的结构中不存在。
对其他公共数据资源中可用的结构使用文件链接
此选项有助于找到与公共数据资源(例如AlphaFold、RoseTTAFold或ESMFold预测)中提供的3D结构类似但未包含在RCSB.org中的结构。
要使用此功能,请将输入模式从“条目ID”切换到“文件链接”(图6)。确保将URL指定为“http”或“https”协议。指定文件格式,默认为mmCIF,但也支持BinaryCIF文件。像往常一样指定图案中的残留物。决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮以启动搜索。
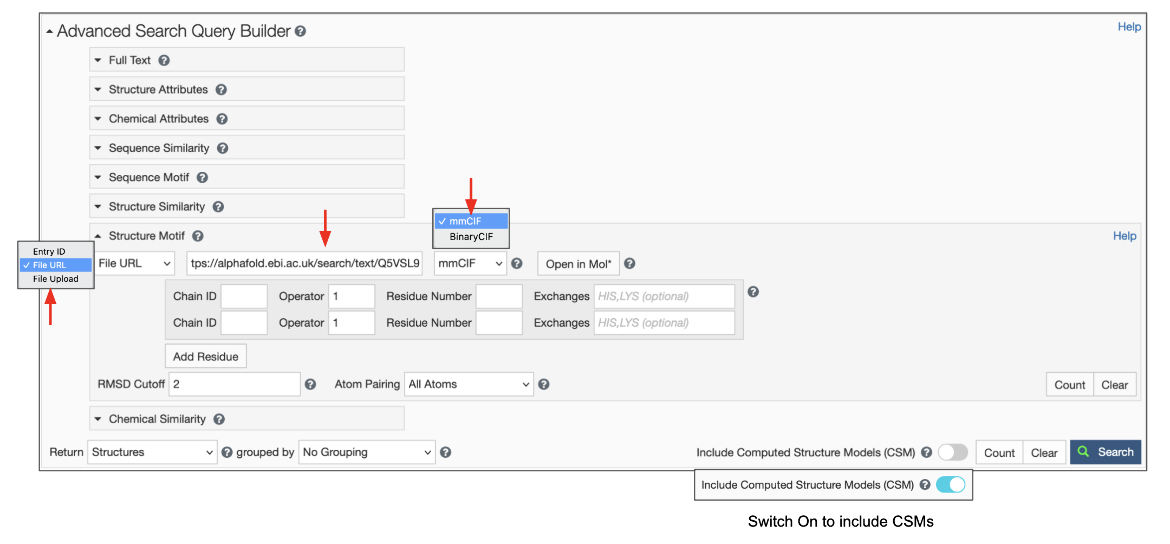
|
| 图6:Advanced Search面板显示了Structure Motif搜索面板,用于使用file Link选项上载文件。 |
上传您自己的本地文件
这将允许您访问一个按钮,该按钮允许您从文件系统中选择文件。要使用此功能,请将输入模式切换为“File Upload”(图7)。支持扩展名为“.cif”、“.bcif”,“.pdb”和“.ent”的文件及其gzipped(“.gz”)变体。选择文件后,它将自动上传到我们的服务器,输入模式将切换到“文件链接”。您的文件将被一个唯一的URL引用。其他用户无法猜测这个随机URL;但是,请注意,任何知道此URL的人都可以访问您的数据。
上传的文件将在90天内可用,这意味着您可以在有限的时间内为搜索添加书签或与同事共享。如果你想在出版物、博客文章或类似内容中持续引用搜索,你应该将你的文件上传到Dropbox或Google Drive等文件共享服务,并使用“文件链接”功能引用它。存储在MyPDB中的查询也是如此。最后,支持的最大文件大小为10 MB,较大的文件也需要使用外部文件共享服务。
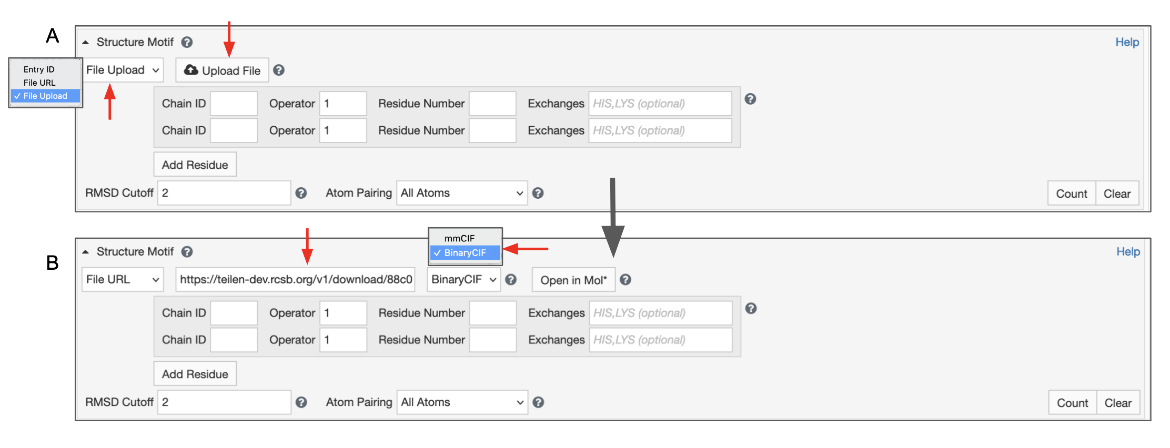
|
| 图7:高级搜索面板显示了用于从本地驱动器上载文件的Structure Motif搜索面板。A.初始视图B.加载本地驱动器中的文件以创建临时web链接后。 |
无论使用何种方法,一旦指定了结构坐标,就会出现一个按钮“Open in Mol*”。点击此按钮可用于指定结构图案中的残留物。再次单击此按钮(即,在指定了残留物选择后)将在Mol*中打开结构,并仅显示“结构主题搜索”面板中列出的残留物。
注:
- 要优化残留物的选择,请更多地打开选择并选择显示的所有氨基酸。现在,使用“控制”面板中的选项显示聚合物,并选择其他(或更少)残留物来修改/完善查询,然后单击“结构主题搜索”>>“提交搜索”按钮。
- 如果手动扩展高级搜索面板以包含特定位置的替代残留物,则“在分子中打开*”按钮将仅显示结构中存在的残留物。面板中列出的可能备选方案将不会显示,因为这些坐标在可视化的结构中不存在。
要手动填充结构图案残留物,请完成以下步骤:
- 插入包含查询三维结构主题的PDB ID或RCSB.org指定的CSM ID。
- 指定2到10个残留物,这些残留物组成了要在存档中的其他结构中查找的残留物组。
- 第一个框用于聚合物链ID(
标签_符号_id)残留物。注意,一个基序可能包括来自多个聚合物链的氨基酸。
- 操作员框用于选择性地指定用于生成生物组装的转换操作(参见PDB ID2个例如)。通过他们的
每个id的结构。运算符组合的注释类似于1x61或Px61。如果引用原始坐标,请将值设置为“1”。
- 查询中包含的残渣编号由其
标签_设备id注意,在出版物中,残留物很可能被其引用授权seq_id,由作者分配的标识符。然而,为了定义查询和报告结果,RCSB PDB网站使用标签_设备id.
- 交换-可选择定义特定于位置的交换或替换。注意,默认情况下,只有参考结构中观察到的残留物类型才被视为有效。一组逗号分隔的三字母代码允许在指定位置搜索不同的氨基酸(或核苷酸)。必须包括原始残留物类型,才能在特定位置进行考虑。
- 使用“添加残基”按钮扩展选择范围,在结构基序中包含其他氨基酸残基,或使用右侧的“x”按钮删除单个残基。
- “RMSD截止”参数可用于筛选不太可能具有生物相关性的高RMSD点击。
- “Atom Pairing”参数对用于对齐的原子集进行细粒度控制。默认情况下,对所有原子进行求值。或者,仅主干、仅侧链或仅Cα/C4′和Cβ/可以选择C1′原子进行RMSD计算。
- 确保将结果类型设置为“Assemblies”,以获取结果页面上的详细信息,其中包括匹配的剩余标识符,并报告此次命中的分数。注意:一个条目可能会出现多个查询结构主题。由于基序可能跨越多个聚合物链,因此每次出现都是一个集合。如果未选择此选项,则结果中仅列出包含查询主题的PDB条目。
决定是否包括或排除CSM,然后单击带有绿色放大镜图标的蓝色搜索按钮以启动搜索。
如何解释成绩分数?
结果显示为“程序集”。
返回PDB档案中包含类似查询基序的残基组的所有程序集,并通过其标签_符号_id和标签_设备id.之间的差异标签_设备id和授权seq_id将在方括号中报告。这个标签_压缩id报告了每种残留物的含量。还提供了比赛的RMSD分数(见图8)。
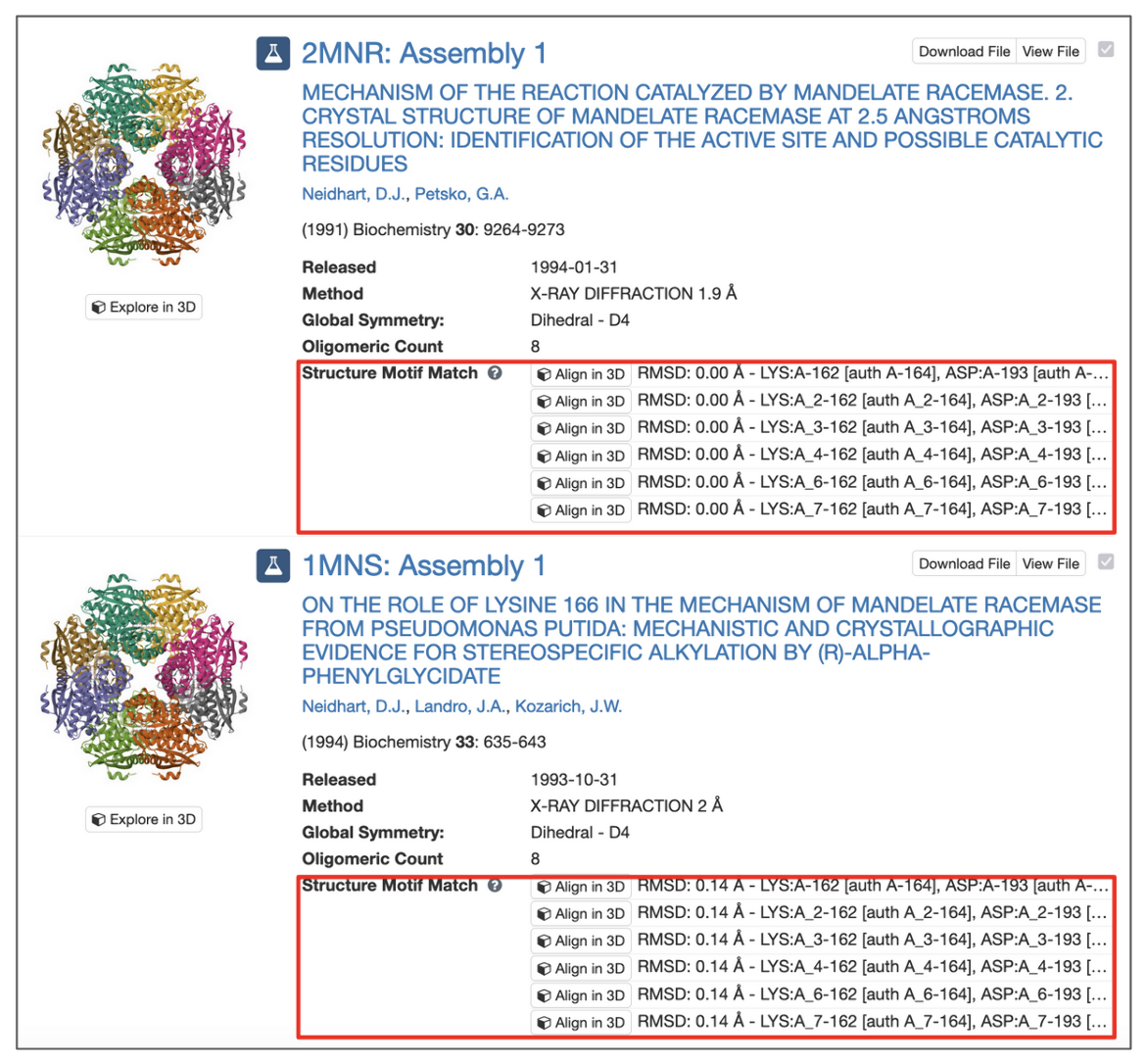
|
| 图8:具有匹配上下文的结构Motif搜索结果。 |
所有潜在匹配都用根平方偏差(RMSD)分数报告,该分数是通过将每个识别的匹配与查询基序对齐并测量每个匹配原子的位移来计算的。值0.0Å表示最佳对齐,不同残基组的值较高。
基元可能出现在沉积坐标的对称伙伴中。在这些情况下,链标识符将包括相应的每个id的结构在下划线之后(例如,LYS:A_2-162)。
每行开头的“Align”按钮启动Mol*视图,显示查询主题和选定匹配的叠加。
结构主题搜索的局限性
结构模体搜索服务是一种启发式搜索,假阴性率<2%。这意味着,与较慢的穷举搜索策略相比,每50个相关点击中就会有1个被遗漏。该服务使用3个特征来描述查询模式中所有剩余对的几何属性(图9):主干距离(db条),侧链距离(d秒)和C之间的角度θαC类β这两种氨基酸的载体。如果其中一个属性差异过大,则会错过命中。距离的公差值为1Å,角度特性的公差值是20°。
低RMSD值<0.5Å的点击的假阳性率趋于0,但高RMSD值的点击假阳性率增加。这也意味着在只包含C的结构中不会发现命中α跟踪。
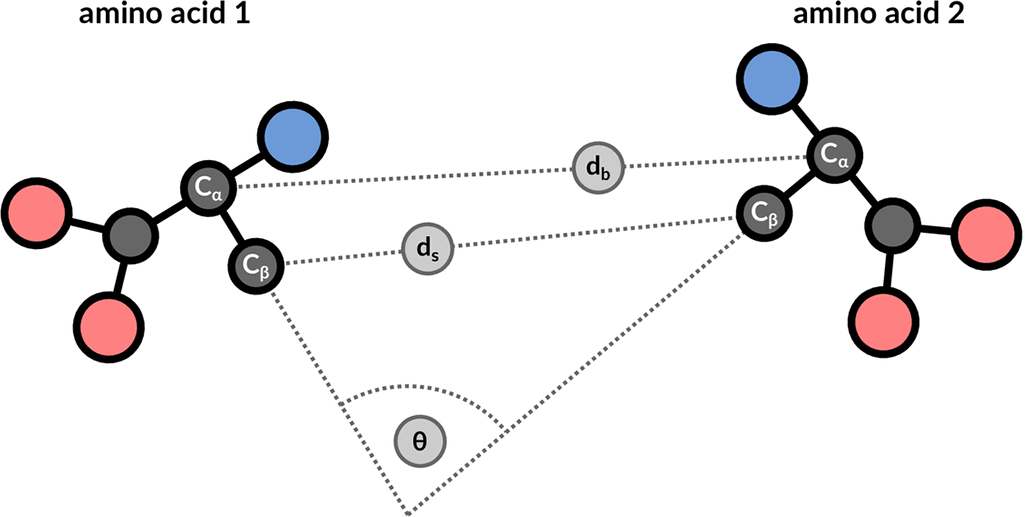
|
| 图9:3几何性质用于描述残数对:C之间的主干距离α原子,C之间的侧链距离β原子和相应矢量之间的角度。 |
有关搜索算法和评分的详细信息,请参阅Bittrich等人,2020年特别是,请参见图3和随附的对观察到的假阴性的讨论。“针对高级用户”部分提供了有关如何使用增加的容差值来运行结构基序查询的信息,该容差值以牺牲较高的运行时间为代价来降低假阴性率。
示例
结构基序搜索服务发现2到10个空间邻近的残基的相似性。有趣的图案在文献中有定义,并可在诸如催化位点图谱(CSA)。它适用于许多示例查询。所有给定的标识符都是标签_符号_id和标签_设备id.
表1:结构主题搜索示例
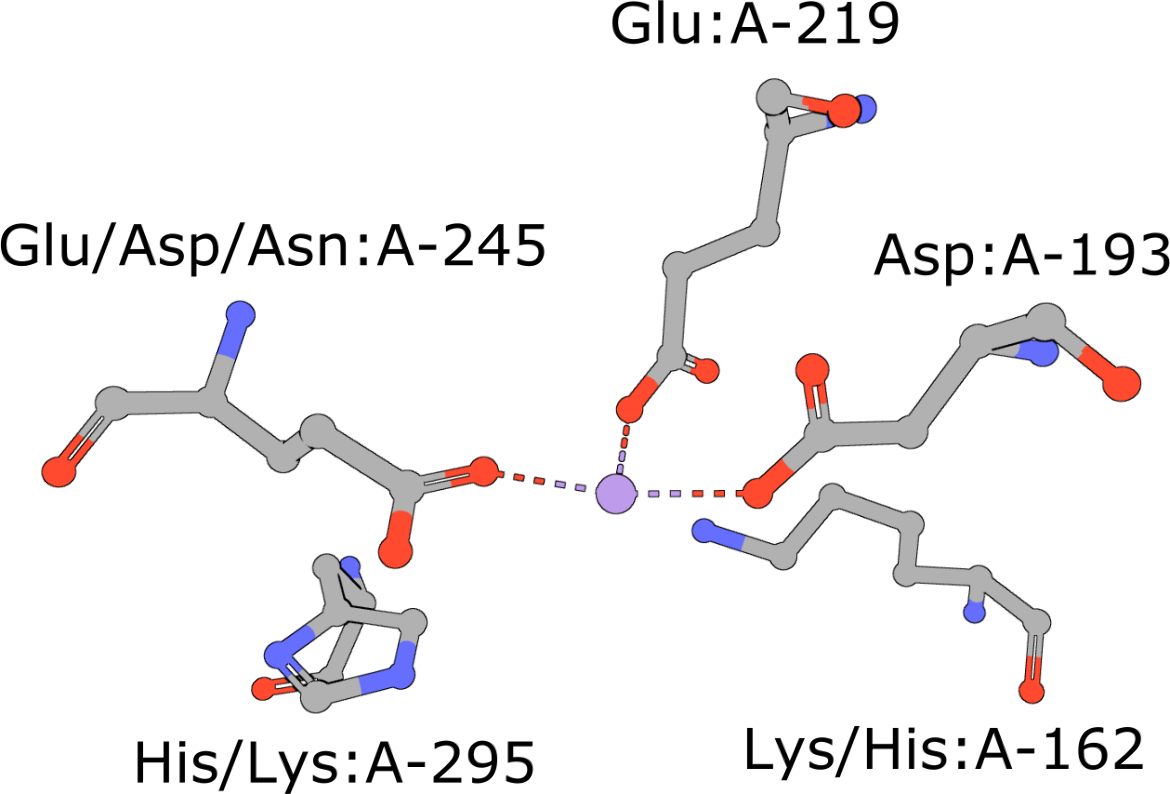 |
烯醇化酶超家族模板
(执行查询)
烯醇化酶超家族是一组序列不同但在3D结构上大体相似的蛋白质,所有这些蛋白质都催化从羧酸中去除质子(巴比特,1996年). 支持这种催化功能的结构基序(孟,2004)以PDB ID表示2个. |
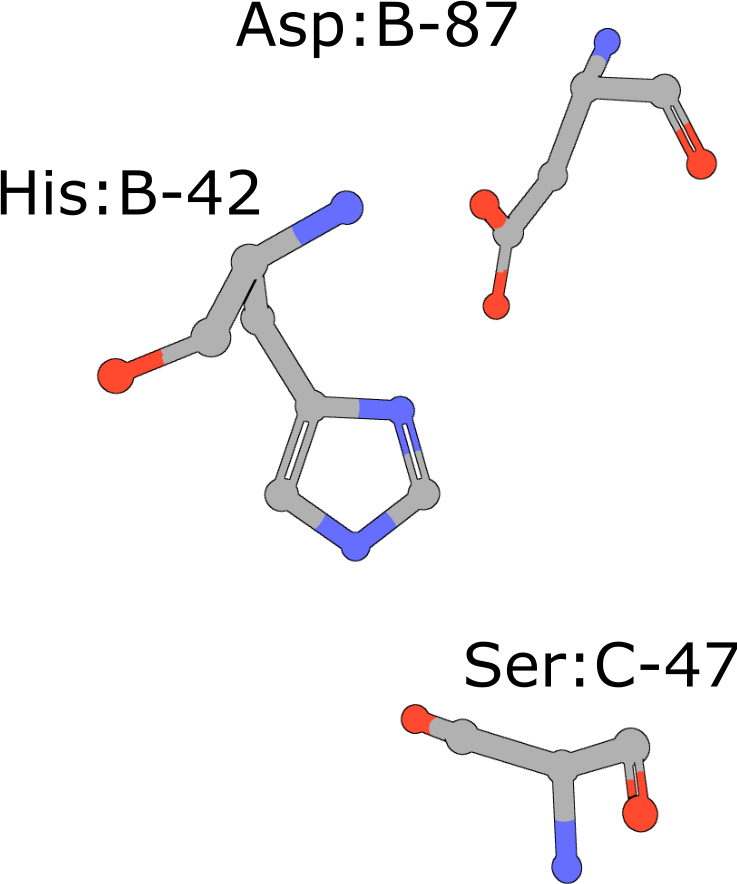 |
丝氨酸蛋白酶催化三联体
(执行查询)
许多水解酶在催化过程中使用丝氨酸亲核试剂。典型丝氨酸蛋白酶催化三联体由His、Asp和Ser残基(PDB ID)组成4cha码). 通常,这些残基出现在两条多肽链中,因为许多这些蛋白酶最初是作为需要通过蛋白水解过程激活的酶原制造的(海德斯特罗姆,2002年)防止细胞内蛋白质被不受控制的消化。
您还可以将使用关键字查询缩小结果集的范围,找到更多有趣的查询主题。 |
 |
氨肽酶
(执行查询)
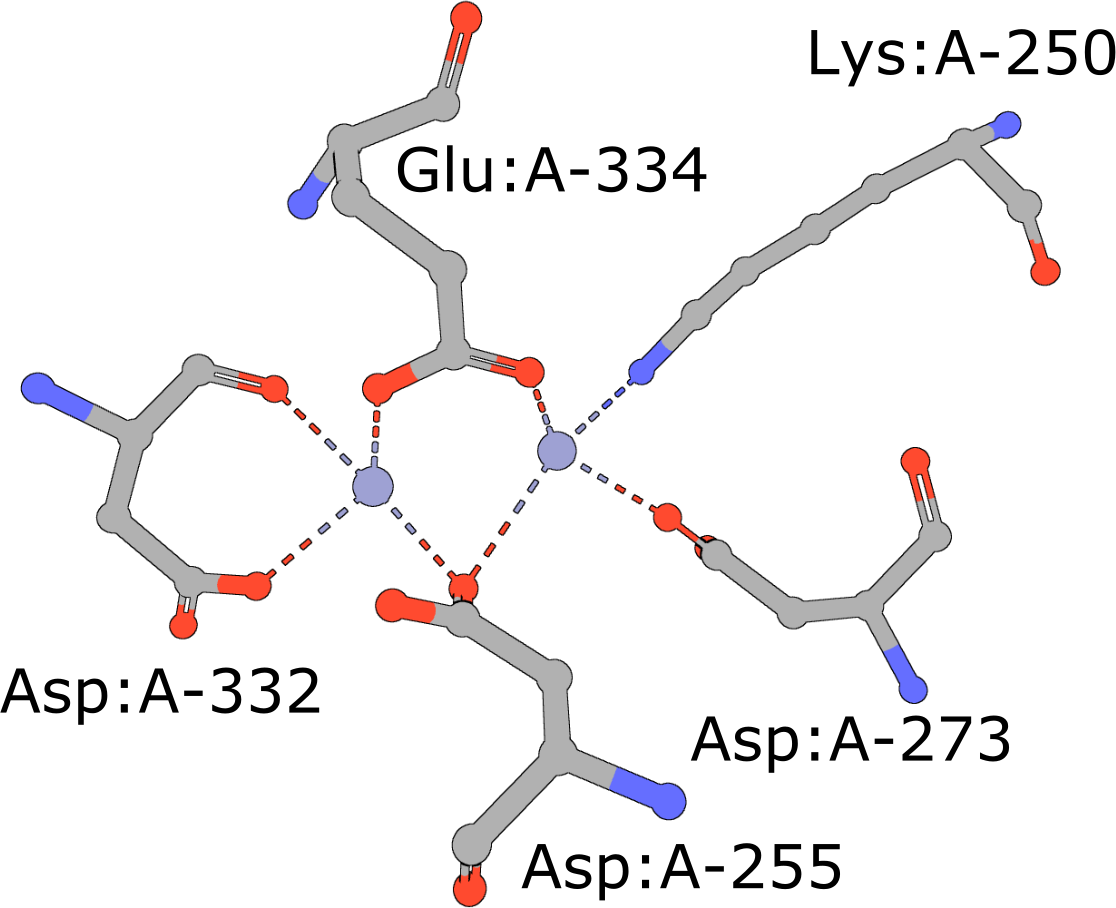
氨肽酶通过清除多肽链N或氨基末端的残基在蛋白质降解中发挥重要作用(白肋烟,1990年). 牛亮氨酸氨肽酶(BLLAP)是一种含有32四元对称。BLLAP的活性位点包含两个相邻的锌离子,由~2.9Å分隔,并由五个保守残基Lys、Asp、Asp和Glu(PDB ID)的侧链协调1圈). |
 |
锌指
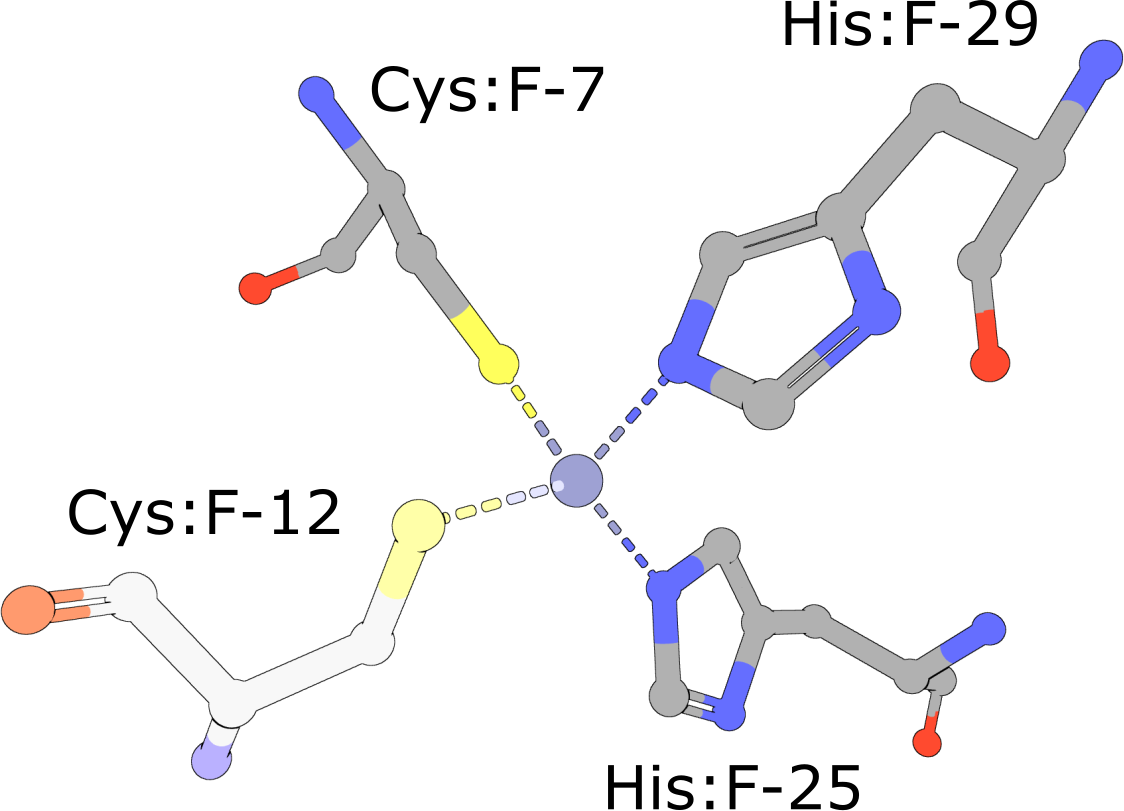
(执行查询)
真核转录因子通常含有His2/Cys公司2锌指结构域(PDB ID1克2夫)结合DNA的。这些基序由两个半胱氨酸和两个组氨酸残基组成,它们稳定包裹和协调单个锌离子的小ββα结构域结构(Pabo,2001年). 在没有锌离子的情况下,这些结构域不采用紧凑的折叠结构,并且不能结合DNA。 |
 |
RNA G-四分体
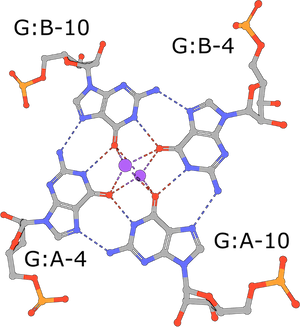
(执行查询)
G-四分体是一种常见的核酸结合基序(PDB-ID300万). 它们由鸟嘌呤组成,并由Hoogsteen碱基对稳定。四个O6氧原子协调单价离子,如K+,单个四分体往往一个叠一个(Burge,2006年). |
 |
镉的配位
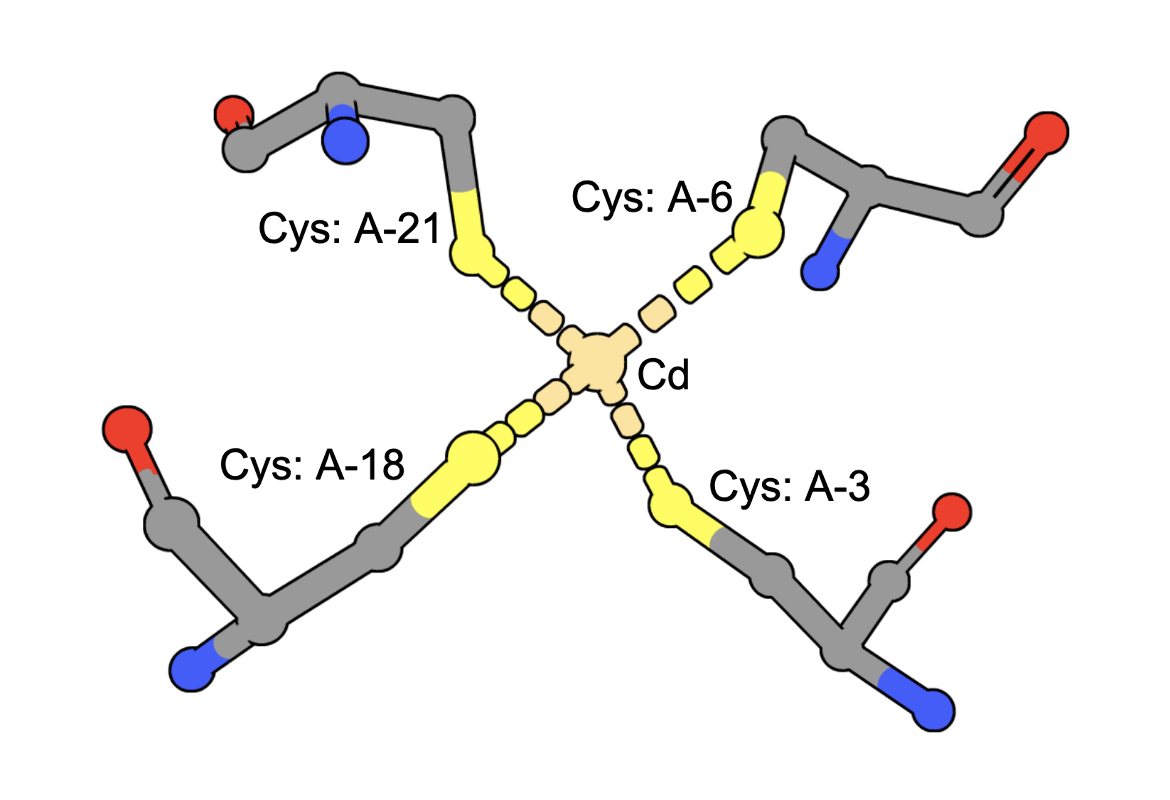
(执行查询)
镉离子可以与蛋白质中的含硫氨基酸(例如Cys)结合。可以通过组合两种类型的查询来构造一个查询,以查找由四个Cys残基结合的Cd结构:
a.结构基序搜索-寻找离子周围有4个Cys残基的结构(通常在这些几何结构中发现锌),以及
b.化学属性搜索-包含镉离子的结构
该查询查找(结构与Cd)和(结构与4个Cys残基定位以协调离子)的交集。结果中的一个示例显示了一个结构,该结构具有与PDB ID中的四个Cys氨基酸结合的Cd5sbj个.
注:结构可能具有Cd,但它不与4个Cys残基配位。因此,应仔细检查此查询的结果,以确保其中至少包含Cys协调的一种镉4. |
对于高级用户
所有Java源代码在GitHub上都是公开的(github.com/rcsb/structmotif-search),该项目作为Maven工件分发。
我们鼓励感兴趣的用户在本地安装结构主题搜索服务。这允许您为您的确切需求配置工具,并对所有参数进行细粒度控制,其中一些参数没有在上公开rcsb.org网站。其他功能包括:
- 允许检索更多不同点击的增加容差值
- 使用不属于PDB存档的自定义结构(例如AlphaFold结构)定义查询主题
- 未知功能结构中已知基序出现的筛选
工具书类
- Bittrich S,Burley SK,Rose AS(2020)使用反向索引策略在蛋白质中实时结构基序搜索。公共科学图书馆计算生物学。16(12):e1008502,doi:10.1371/日记.pcbi.1008502
- Meng EC、Polaco BJ、Babbitt PC(2004)《超级家族活性站点模板》。蛋白质:结构、功能和生物信息学。55(4):962–976,doi:10.1002/2009年保护.
- Babbit PC、Hasson MS、Wedekind JE、Palmer DR、Barrett WC、Reed GH等(1996)烯醇化酶超家族:酶催化提取羧酸α质子的一般策略。生物化学。35(51):16489–16501,doi:10.1021/bi9616413.
- Hedstrom L.(2002)丝氨酸蛋白酶的机制和特异性。化学评论。102(12):4501–4524,doi:10.1021/cr000033倍.
- Burley SK,David PR,Taylor A,Lipscomb WN(1990),亮氨酸氨肽酶2.7-A分辨率的分子结构。美国国家科学院院刊。87(17): 6878–6882.
- Pabo CO,Peisach E,Grant RA(2001)新型Cys2His2锌指蛋白的设计和选择。生物化学年鉴。70(1):313–340,doi:10.1146/anurev.biochem.70.1313.
- Burge S、Parkinson GN、Hazel P、Todd AK、Neidle S(2006)《四倍体DNA:序列、拓扑和结构》。核酸研究。34(19):5402–5415,doi:10.1093/nar/gkl655.