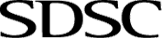介绍
PDB配体结构质量指标的完整描述见“蛋白质数据库中小分子配体的简化质量评估”结构30(2):252-262.e4。 文件编号:10.1016/j.str.2021.10.003(Shao等人,2022)。
PDB档案中的配体是什么?
PDB结构中与生物聚合物(如蛋白质和核酸)相关的小分子,如离子、溶剂分子、辅因子等,被称为配体。超过70%的PDB结构在大分子复合物中包含一个或多个小分子配体,不包括水分子。PDB中的小分子配体可大致分为“功能性”(即可能发挥生物/生化作用的配体)或“非功能性”。功能配体包括辅因子、活化剂、抑制剂、底物、产物和中间体,以及其中任何一种的类似物,而非功能配体通常是溶剂、盐、离子和结晶剂。与蛋白质或核酸靶标结合的配体结构已被证明有助于理解其相互作用模式、结合强度和选择性(Burley 2021)。wwPDB化学成分词典(CCD)定义了PDB中发现的每一种独特的小分子配体,其具有不同的标识符CCD ID和详细的化学描述(Westbrook等人,2015)。每个唯一定义的CCD配体可以存在于多个PDB结构中,并且可以在一个PDB结构中具有多个拷贝。
什么是配体结构质量?
PDB中配体结构的质量可以根据其与实验数据的匹配程度以及几何参数(如键长和角度)的准确性进行评估。wwPDB验证报告提供了单个配体质量的验证(Feng等人,2021),包括实际空间R因子(RSR)的局部电子密度良好指标(Jones等人,1991)和实际空间相关系数(RSCC)(Brändén和Jones,1990)EDS计算的X射线结构(Kleywegt等人,2004);Mogul(Bruno等人,2004)基于剑桥结构数据库(CSD)的小分子参考结构计算的化学和几何结构质量指标键长(RMSZ-bond-length)和键角(RMSZ-bond-angle)的均方根偏差Z分数(Groom等人,2016);以及MolProbity计算的原子间不适当碰撞的度量(Chen等人,2010)。
目前,配体质量分析的重点是X射线共晶结构中的配体,其完整的RSR、RSCC、RMSZ-bond-length和RMSZ-bond-angle验证数据来自wwPDB验证报告。请注意,以下类别的配体目前未进行配体质量分析:(a)未通过X射线衍射法求解的结构(b)没有几何特征的配体,例如具有单个非氢原子的离子配体(例如钙或钠离子),缺乏键长和角度的验证数据,和(c)缺乏相关结构因子数据的X射线结构中的配体(通常在2008年之前沉积),因为没有结构因子数据就无法计算实验数据的质量。(d) 以分支实体表示的低聚糖目前不包括在质量报告中。未来将对所有低聚物小分子进行验证分析。
为什么了解PDB配体结构质量很重要?
过去五十年来,PDB中X射线共晶体结构的持续增长为生物医学研究提供了大量开放存取数据。它还为PDB数据使用者在决定使用哪个PDB结构以及用于什么目的方面带来了相当大的挑战。PDB档案中配体的每个实例与实验数据和化学/几何参数的一致性可能不同。配体结构质量分析允许PDB用户快速选择合适质量的特定配体实例进行分析、实验或分子设计。
文档
如何评估PDB配体结构质量?
为了便于PDB用户快速评估配体结构质量,RCSB PDB构建了综合排名分数,并在易于解释的2D配体质量图中实现了评分算法,以进行可视化总结。配体质量综合排名得分(1)将相关质量指标聚合为一维指标,(2)建立均匀分布的综合排名得分,以简化解释。
将相关质量指标汇总为综合指标-在统计学中,可以使用主成分分析(PCA)将两个或多个相关变量简化为一个变量。对于多个变量,主成分分析找到一组正交向量来表示数据在多维空间中的分布。每个向量都是多个变量的线性组合,称为主成分(PC)。如果第一主成分(PC1)解释了所有变量的大部分方差,则可将其用作代表变量。对RSR和RSCC进行PCA(两者都是配体模型与电子密度拟合程度的指标)。他们的第一个主成分被指定为PC1-拟合,解释了RSR和RSCC 84%的方差,并用作一维(1D)测量配体结构与局部电子密度的匹配程度。对RMSZ-bond-length和RMSZ-bond-langle(两者都是分子几何结构的指标)进行另一个主成分分析,得出PC1-几何结构,解释了82%的总方差。因此,PC1几何结构被用作1D复合指标,以评估化学和几何参数的准确性。由于此处描述的两个PC1相对独立,配体拟合质量和几何质量必须分别通过PC1拟合和PC1几何进行评估。有关此分析的更多信息,请参见(Shao等人,2022)。
RCSB PDB配体质量综合排名得分:计算PDB中所有配体的PC1-拟合和PC1-几何结构的综合指标,以便在整个档案中对配体结构质量进行排名。特定配体结构的综合排名分数被定义为与被查询配体拟合或几何质量较差的PDB配体的百分比,这与wwPDB验证报告中为整体模型质量定义的排名一致(Feng等人,2021)。由于排名是均匀分布的,所以综合排名分数具有最简单的解释:0%表示最差,100%表示最好,50%表示中等质量。例如,对于不同PDB条目中的化合物Flavin腺嘌呤二核苷酸(FAD),PC-fitting的综合排名得分如图所示。
|
| 电子密度拟合的综合排名分数。所有配体实例都是以1.5Å的分辨率确定的CCD ID FAD结构,沿着彩色垂直条从最佳(顶部)到最差(底部)(蓝色:优;红色:劣)。PC1-拟合综合排名分数在PDB ID、链ID和残渣数下方的括号中提供。注意:从PDB ID 2QWX中选择了两个实例:链A的残基#232(从顶部起第二个)和链B的残基#232(自顶部起第三个)。 |
如何查看和解释配体结构质量评估?
对于PDB中接受配体结构质量分析的每个配体,RCSB PDB提供以下评估:
- 配体结构与实验数据拟合优度的1D滑块-在结构摘要页面上,在整体质量滑块下,添加了一个滑块,以显示结构中“感兴趣配体”(LOI)的拟合优度。LOI是结构作者或RCSB PDB根据以下标准在实验中指定为研究重点的大分子复合物中的功能配体:(1)公式重量>150 Da,和(2)不在可能的非功能配体排除列表中(Shao等人,2022)。滑块的红色末端标记为0(0%),表示拟合不良,而蓝色末端标记为1(100%),表示良好拟合。结构中每种配体的最佳拟合实例显示为实心垂直蓝色条。将光标悬停在每个条形图上,将显示其对应的配体名称。单击滑块上的垂直条形图将打开“配体”选项卡,以显示有关配体质量评估的更多详细信息。
- PC1拟合和PC-1几何结构的2D配体质量图-在PDB条目的新“配体”选项卡中,结构中配体的所有实例的实验数据拟合和几何评估显示在第一个(最左边)2D图中。X轴显示了PC1拟合的综合排名分数,以及描述配体结构的原子坐标在多大程度上得到了实验数据的支持;Y轴显示了PC1几何结构的综合排名分数,即配体结构与已知化学结构的符合程度。绿色菱形符号表示条目中此配体的最佳匹配实例,而所有其他实例均显示在绿色圆圈中。演示是交互式的。将鼠标悬停在菱形符号上可以提供配体实例信息,并在页面的表格中突出显示有关它的其他详细信息。将鼠标悬停在圆圈符号上只会突出显示页面上表格中有关配体的详细信息。点击菱形符号(配体的最佳实例)将在Mol*中打开配体的3D视图(Sehnal等人,2021),并显示配体以及实验确定的电子密度图。
- 配体的三维显示和实验数据拟合-在“3D视图”选项卡中,根据Mol*中的实验数据可视化配体和局部电子密度,PDB用户可以检查配体结构及其与电子密度的匹配情况。用户可以通过2D配体质量图中的电子密度图快速调用3D配体显示。
- PDB条目的配体结构质量比较-在特定PDB条目的“配体”选项卡中,排名信息以更多2D图和表格摘要形式显示,以帮助用户比较配体结构,并从不同条目中选择感兴趣化合物的最佳配体模型结构。在当前条目的最佳配体模型和(1)具有相同蛋白质靶标的其他PDB条目中相同化合物的最佳拟合配体模型之间进行了两次比较;(2) 任何其他PDB条目中相同化合物的最佳拟合配体模型。其他重要的质量指标,包括碰撞和手性错误,也在本页的表格报告中提供。
例子
RCSB PDB配体结构质量评估提供了各种查看和选择配体最佳实例的方法,以供进一步分析,并作为设计和开发的起点。PDB条目6wjc中CCD ID为Y01和OIN的配体的结构质量评估如下所示:
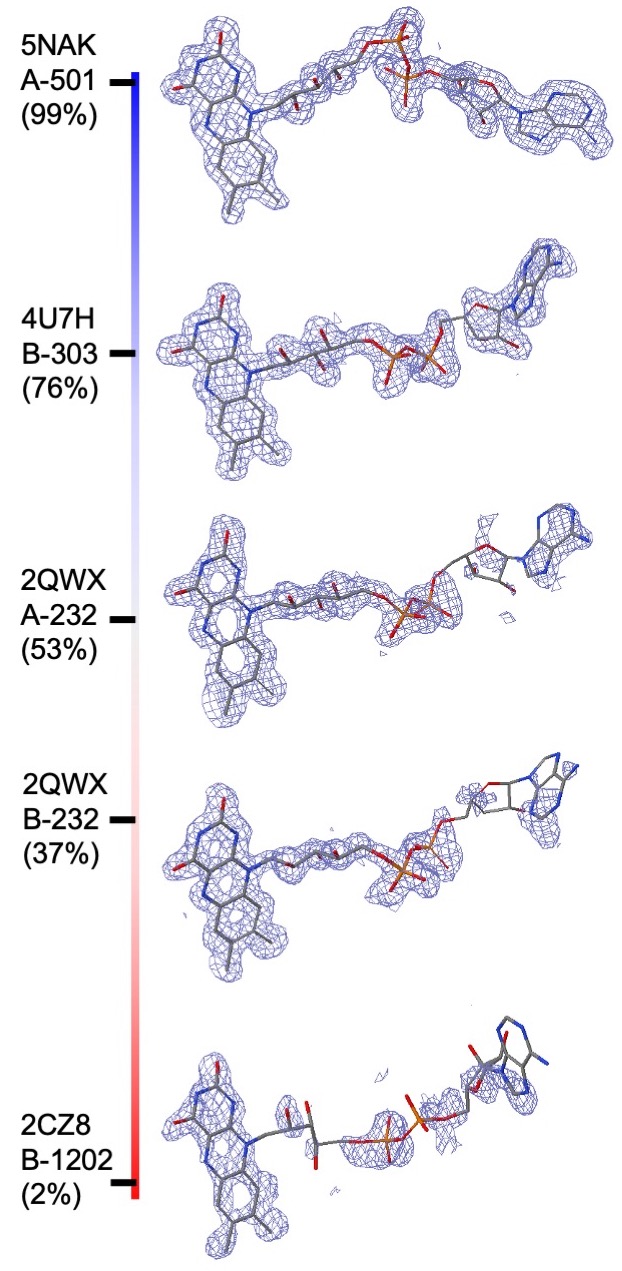
在结构摘要页面上:1D滑块
下图显示了CCD ID Y01和OIN配体最佳实例的配体拟合质量表示。滑块的红色末端标记为0(0%),表示拟合不良,而蓝色末端标记为1(100%),表示良好拟合。
|
| 结构摘要页面上的配体质量摘要滑块。此处所示的1D图具有从最差(0%,红色)到最佳(100%,蓝色)的颜色编码等级,用于PC1拟合。两个深蓝色条分别代表PDB ID 6WJC中Y01和OIN的最佳拟合配体模型。点击任何一个条形图,用户就会看到下面的2D配体质量图。 |
在配体选项卡页上:2D配体质量图和表格报告
您可以通过单击配体Y01的最佳实例栏,或直接单击配体选项卡,然后选择Y01配体子选项卡,从而在配体选项卡中访问配体Y01-的质量分析。
配体结构质量指标和实际配体结构以及实验数据(电子密度)可以如下所示进行检查。
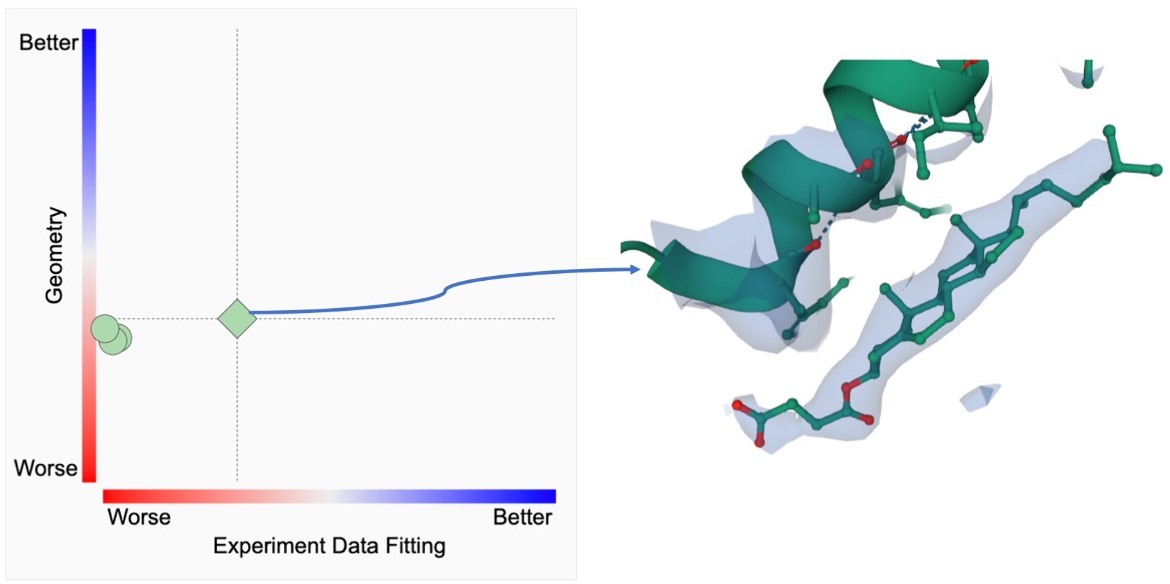
|
| RCSB PDB配体质量图。所示2D图具有从最差(0%,红色)到最佳(100%,蓝色)的PC1-拟合(水平轴)和PC1-几何体(垂直轴)的彩色编码等级。每个符号表示PDB ID 6WJC中CCD ID Y01的配体实例,其水平位置由PC1拟合标记,其高度由PC1几何标记。菱形符号表示最合适的实例,单击它会显示聚焦于配体结构的电子密度的3D显示。 |
几个2D图和一个表格报告显示了给定PDB条目中配体结构的质量,并将其与同一页面上的其他实例进行了比较。
A.本页的第一(左)个图显示了结构中所有Y01实例的2D配体质量图。单击绿色菱形符号(表示配体Y01_A_502)可在Mol*中打开此配体的3D视图。
B.第二个(中间)图将结构中配体的最佳实例与配体与相同聚合物(例如,蛋白质或核酸)结合的其他结构中的前五个最佳拟合实例进行比较。
C.第三张图(右)将最佳配体实例与配体与任何聚合物复合体中的前五个最佳匹配实例进行比较。
D.配体结构质量评估的完整细节见图表下方的表格。该总结可用于识别最符合分析需要的配体PDB条目和实例。
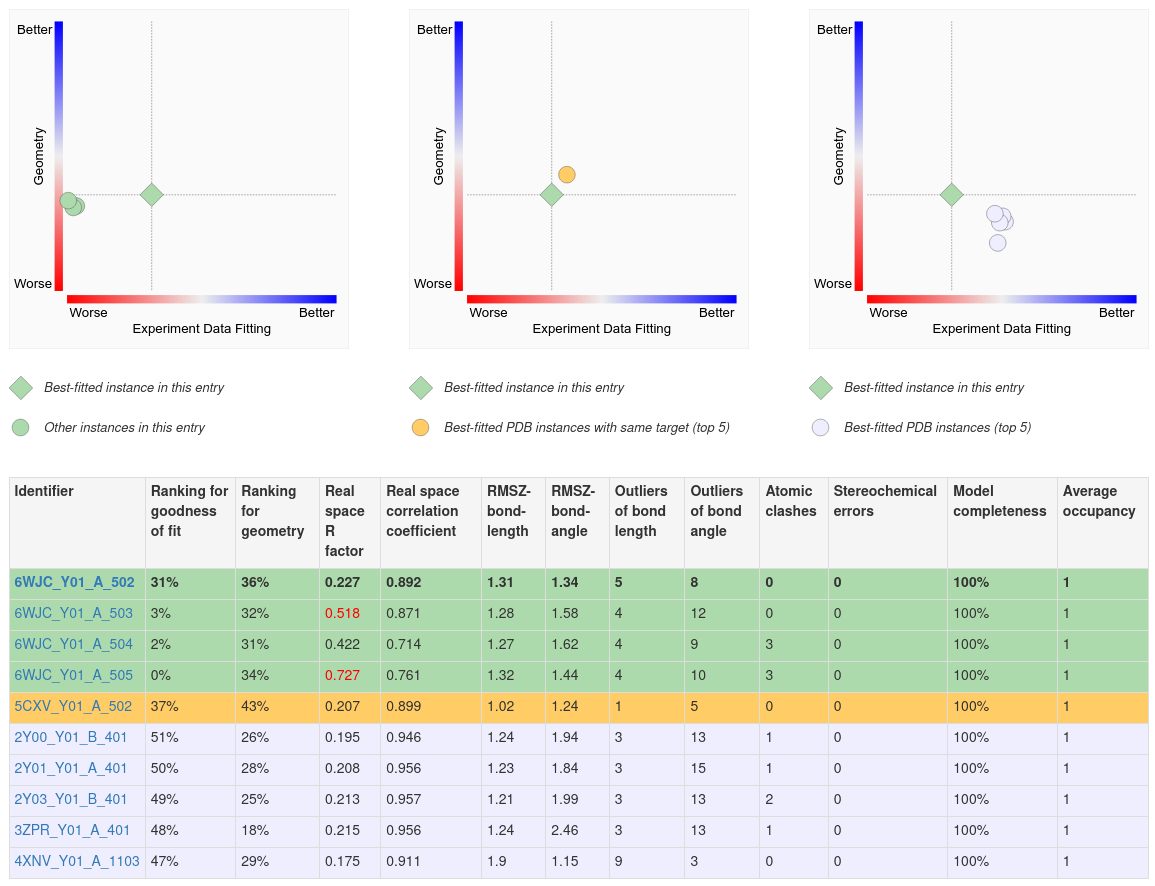
|
| 配体结构质量比较. 顶部的三个2D图分别显示了相同结构内CCD ID Y01配体结构的质量测量值、与同一蛋白质靶结合的同一化合物配体结构比较值以及PDB条目中同一化合物的结构。只有包含配体的前五个PDB条目被选中用于中间和最右侧的绘图。每个图中的绿色菱形符号表示当前PDB ID 6WJC中最适合的实例,对应底部表格报告的顶行。表格报告中以绿色、黄色和灰色突出显示的其他行对应于上述2D图中以相同颜色的圆圈符号显示的配体质量指标。将鼠标悬停在二维图中的任何符号上都会高亮显示表格报告中的相应行。在表中,每个配体质量指标中最差的1%离群值以红色字体突出显示,第一列中的标识符是蓝色的超链接,导致Mol*3D电子密度视图聚焦于标识符标记的配体。 |
工具书类
- Brändén C,Jones T.(1990年)。在客观性和主观性之间。自然。343:687-689.
- Bruno IJ、Cole JC、Kessler M、Luo J、Motherwell WD、Purkis LH、Smith BR、Taylor R、Cooper RI、Harris SE等人(2004年)。晶体衍生分子几何信息的检索。化学信息与计算科学杂志。44(6):2133-2144. doi:10.1021/ci049780b
- Burley SK.(2021年)。结构生物学家和蛋白质数据库对小分子药物发现和开发的影响。生物化学杂志100559。doi:10.1016/j.jbc.2021.100559
- Chen VB、Arendall WB、3rd、Head JJ、Keedy DA、Immormino RM、Kapral GJ、Murray LW、Richardson JS、Richards DC。(2010). 分子完整性:用于大分子晶体学的全原子结构验证。《结晶学报》D.66系列(Pt 1):12-21。doi:10.1010/S0907444909042073
- Feng Z、Westbrook JD、Sala R、Smart OS、Bricogne G、Matsubara M、Yamada I、Tsuchiya S、Aoki-Kinoshita KF、Hoch JC等人(2021年)。加强蛋白质数据库中小分子配体和碳水化合物的验证。结构。29:393-400.e391。doi:10.1016/j.str.2021.02.004
- Groom CR、Bruno IJ、Lightfoot MP、Ward SC.(2016年)。剑桥结构数据库。《晶体学报》B.72(Pt 2):171-179。doi:10.1107/S2052520616003954
- Jones TA、Zou J-Y、Cowan SW、Kjeldgaard M.(1991)。改进了在电子密度图中建立蛋白质模型的方法以及这些模型中的错误位置。《结晶学报》A.47:110-119。
- Kleywegt GJ、Harris MR、Zou JY、Taylor TC、Wahlby A、Jones TA。(2004). uppsala电子密度服务器。《结晶学报》D.60系列(第12部分第1部分):2240-2249。doi:10.1107/S090744490413253
- Sehnal D、Bittrich S、Deshpande M、Svobodova R、Berka K、Bazgier V、Velankar S、Burley SK、Koca J、Rose AS(2021)。Mol*viewer:用于大型生物分子结构三维可视化和分析的现代网络应用程序。核酸研究doi:10.1093/nar/gkab314
- Shao C、Westbrook JD、Lu C、Bhikadiya C、Peisach E、Young JY、Duarte J、Lowe R、Wang S、Rose Y、Feng Z、Burley SK.(2022)。蛋白质数据库中小分子配体的简化质量评估。结构30(2):252-262.e4。文件编号:10.1016/j.str.2021.10.003
- Westbrook JD、Shao C、Feng Z、Zhuravleva M、Velankar S、Young J.(2015)。化学成分词典:对蛋白质数据库中实验测定的三维大分子中的组成分子的完整描述。生物信息学。31(8):1274-1278. doi:10.1093/bioinformatics/btu789