摘要
1简介
系统发育学研究生物体群之间的进化关系。如今,系统发育学受益于分子测序数据技术,以收集大量数据进行分析,旨在改善生命进化树等领域的研究(麦迪逊等。, 2007;Schuh和Brower,2009年)生物的分组等等。随着基因组测序和基因组数据库的出现,大量信息可用于计算处理,使得世界范围内的研究能够解码DNA序列中的信息结构。
由于世界各地许多个人和研究机构的大量合作,大量DNA信息正在被收集和解码,并可用于科学研究。在马查多(2010))通过将数学工具应用于基因组数据,揭示了新的信息模式,解决了这一不断发展的领域。
在本研究中,我们从识别核基因组和线粒体基因组结构模式的角度分析DNA代码。理解DNA可能是人类知识面临的最具挑战性的问题之一(诺贝尔奖网站,http://nobelprize.org/nobel_prizes/medicine/laurates/1968/). DNA复合体结构的解码可能不仅具有初级的生化细节,还具有其他级别的信息(Seitz,2007年). 这一愿景推动了逻辑和数学概念的关联,即直方图、相关性和分析/可视化工具,如多维分析、有向图和数据图。一旦建立了几个物种的方法,它的DNA数据就被用来追求这一愿景。在选定的DNA库中,相当一部分与基因和短重复序列相对应(如加利福尼亚大学圣克鲁斯基因组生物信息学网站所定义),被组织成染色体,这是我们的输入数据。在本研究中,我们考虑了18种物种的可用核和线粒体基因组:10种哺乳动物、2种鸟类、2种鱼类、1种昆虫、2种线虫和1种真菌。我们注意到,在大多数物种中,基因与染色体的关联还不完全。在表1,我们展示了这些物种的染色体特征。
| 物种. | 组. | 核/线粒体染色体. |
|---|
| 人类(Ho) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 19 20 21 22 X Y M |
| 黑猩猩(Ch) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 1 14 15 16 17 19 20 22 X年 |
| 猩猩(Orangutan) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 13 14 15 16 18 19 20 22 X M |
| 猪(Po) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 X米 |
| 负鼠(Op) | 哺乳动物 | 1 2 3 4 5 6 7 8 X M |
| 马(Eq) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 18 20 21 22 24 26 28 29 2 31 X M |
| 狗(Dg) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 19 20 21 22 23 25 27 29 30 31 32 34 36 38 X米 |
| 公牛(Ox) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 23 25 27 29 X M |
| 老鼠(Mm) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 X Y M |
| 大鼠(Rn) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 X M |
| 鸡肉(Ga) | 大道 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 27 28西元 |
| 斑马雀(Tg) | 大道 | 1a b 1 2 3 4 a 5 6 7 9 11 12 14 17 19 20 22 24 26 28 Z M公司 |
| 斑马鱼(Zf) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 24 25 M |
| 四齿龙(Tn) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 21个月 |
| 蚊子(Ag) | 昆虫 | 2l 2r 3r X M |
| 秀丽隐杆线虫(证书) | 蠕虫 | 1 2 3 4 5 X M |
| 线虫(抄送) | 蠕虫 | 1 2 3 4 5 X M |
| 酵母(Sc) | 真菌 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16百万 |
| 物种. | 组. | 核/线粒体染色体. |
|---|
| 人类(Ho) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 19 20 21 22 X Y M |
| 黑猩猩(Ch) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 1 14 15 16 17 19 20 22 X年 |
| 猩猩(Orangutan) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 13 14 15 16 18 19 20 22 X M |
| 猪(Po) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 X米 |
| 负鼠(Op) | 哺乳动物 | 1 2 3 4 5 6 7 8 X M |
| 马(Eq) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 23 24 25 26 27 28 29 2 3 31 X M |
| 狗(Dg) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 19 20 21 22 23 25 27 29 30 31 32 34 36 38 X米 |
| 公牛(Ox) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 23 25 27 29 X M |
| 老鼠(Mm) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 X Y M |
| 大鼠(Rn) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 X M |
| 鸡肉(Ga) | 大道 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 27 28西元 |
| 斑马雀(Tg) | 大道 | 1a b 1 2 3 4 a 5 6 7 9 11 12 14 17 19 20 22 24 26 28 Z M公司 |
| 斑马鱼(Zf) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 20 21 22 23 24 25米 |
| 四齿龙(Tn) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 21个月 |
| 蚊子(Ag) | 昆虫 | 2l 2r 3r X M |
| 秀丽隐杆线虫(证书) | 蠕虫 | 1 2 3 4 5 X M |
| 线虫(抄送) | 蠕虫 | 1 2 3 4 5 X M |
| 酵母(Sc) | 真菌 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16百万 |
| 物种. | 组. | 核/线粒体染色体. |
|---|
| 人类(Ho) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 19 20 21 22 X Y M |
| 黑猩猩(Ch) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 1 14 15 16 17 19 20 22 X年 |
| 猩猩(Orangutan) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 13 14 15 16 18 19 20 22 X M |
| 猪(Po) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 X米 |
| 负鼠(Op) | 哺乳动物 | 1 2 3 4 5 6 7 8 X M |
| 马(Eq) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 18 20 21 22 24 26 28 29 2 31 X M |
| 狗(Dg) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 19 20 21 22 23 25 27 29 30 31 32 34 36 38 X米 |
| 公牛(Ox) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 23 25 27 29 X M |
| 老鼠(Mm) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 X Y M |
| 大鼠(Rn) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 X M |
| 鸡肉(Ga) | 大道 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 27 28西元 |
| 斑马雀(Tg) | 大道 | 1a b 1 2 3 4 a 5 6 7 9 11 12 14 17 19 20 22 24 26 28 Z M公司 |
| 斑马鱼(Zf) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 24 25 M |
| 四齿龙(Tn) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 21个月 |
| 蚊子(Ag) | 昆虫 | 2l 2r 3r X M |
| 秀丽隐杆线虫(证书) | 蠕虫 | 1 2 3 4 5 X M |
| 线虫(抄送) | 蠕虫 | 1 2 3 4 5 X M |
| 酵母(Sc) | 真菌 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16百万 |
| 物种. | 组. | 核/线粒体染色体. |
|---|
| 人类(Ho) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 19 20 21 22 X Y M |
| 黑猩猩(Ch) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 1 14 15 16 17 19 20 22 X年 |
| 猩猩(Orangutan) | 哺乳动物 | 1 2a b 3 4 5 6 7 8 9 10 11 13 14 15 16 18 19 20 22 X M |
| 猪(Po) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 X米 |
| 负鼠(Opossum) | 哺乳动物 | 1 2 3 4 5 6 7 8 X M |
| 马(Eq) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 23 24 25 26 27 28 29 2 3 31 X M |
| 狗(Dg) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 15 16 17 19 20 21 22 23 25 27 29 30 31 32 34 36 38 X米 |
| 公牛(Ox) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 14 15 16 17 18 20 21 22 23 25 27 29 X M |
| 老鼠(Mm) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 X Y M |
| 大鼠(Rn) | 哺乳动物 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 X M |
| 鸡肉(Ga) | 大道 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 27 28西元 |
| 斑马雀(Tg) | 大道 | 1a b 1 2 3 4 a 5 6 7 9 11 12 14 17 19 20 22 24 26 28 Z M公司 |
| 斑马鱼(Zf) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 19 20 21 22 23 24 25米 |
| 四齿龙(Tn) | 鱼 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 20 21个月 |
| 蚊子(Ag) | 昆虫 | 2l 2r 3r X M |
| 秀丽隐杆线虫(证书) | 蠕虫 | 1 2 3 4 5 X M |
| 线虫(抄送) | 蠕虫 | 1 2 3 4 5 X M |
| 酵母(Sc) | 真菌 | 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16百万 |
DNA实现了由符号{T,C,A,G}组成的字母表。任何简单的数值转换都可能产生偏差并破坏内在信息。因此,决定直接处理非数字代码。由于信息量巨大,因此采用了基于直方图的测量方法。然而,总的来说,直方图并不反映动态。为了克服这一局限性,考虑了一种基于符号序列计数的灵活模式检测算法(文加和阿尔梅达,2003年). “灵活”是指算法可以计算长度序列n个项,每个项由四个基本符号之一组成。
除酵母菌(Sc)外,可用的染色体数据包括第五个符号(“N”),与不属于基因组的屏蔽DNA符号相对应,通常出现在较大的连续序列中。例如,人类Y染色体文件中有59 373 566 bp,其中33 710 000 bp是“N”(56.78%),排列在17个序列中,最大的序列有30 000 000个符号。另一个例子是Chicken Ga25染色体,有2051 775 bp,其中663 879个是“N”(32.67%),排列在274个序列中,最大的是50万个符号。HoY和Ga25只是“N”符号百分比>10%的染色体的两个例子,但大多数染色体的百分比较小。
我们决定不在序列中使用“N”作为第五个符号,或者不将其替换为任何符号{T、C、G、a},因为这将在序列处理中引入未知偏差。然后我们考虑了两种方法:(a)在预处理步骤中删除所有“N”符号,或(b)处理序列,但忽略任何带有“N”的序列。虽然(a)和(b)看起来可能不同,但我们得出的结论是,差异最小,并且(a)可以在不影响结果和结论质量的情况下得到有利的使用。
以{Ho,Ck,Tn,Ag}核染色体和序列长度为例n个= 8,表2最右边的列综合了(a)和(b)方法的差异。对于Ga25,皮尔逊相关系数第页在(a)和(b)序列之间n个=8收益率第页>0.9999717,而对于HoY,相应的系数第页大于0.9999999。我们的结论是,这两种方法在统计上对于设想的DNA解码是等效的。因此,我们选择在构建直方图之前丢弃“N”符号。
| 染色体. | 删除“N”的序列[α]. | 过滤了“N”的序列[β]. | (α − β)/β (%). |
|---|
| 镓25 | 1367889 | 1366030 | 0.136088 |
| 镓3 | 110204947 | 110177075 | 0.025297 |
| Tn1型 | 20304845 | 20315377 | 0.051869 |
| Tn15型 | 6235253 | 6236842 | 0.025484 |
| AgX公司 | 21470369 | 21477782 | 0.034527 |
| 年龄2岁 | 48065434 | 48071405 | 0.012423 |
| 霍伊 | 25653559 | 25653447 | 0.000437 |
| Ho5公司 | 177695253 | 177695218 | 0.000020 |
| 染色体. | 删除“N”的序列[α]. | 过滤了“N”的序列[β]. | (α−β)/β(%). |
|---|
| 镓25 | 1367889 | 1366030 | 0.136088 |
| 镓3 | 110204947 | 110177075 | 0.025297 |
| Tn1型 | 20304845 | 20315377 | 0.051869 |
| Tn15型 | 6235253 | 6236842 | 0.025484 |
| AgX公司 | 21470369 | 21477782 | 0.034527 |
| 氯化银 | 48065434 | 48071405 | 0.012423 |
| 霍伊 | 25653559 | 25653447 | 0.000437 |
| Ho5公司 | 177695253 | 177695218 | 0.000020 |
| 染色体. | 删除“N”的序列[α]. | 过滤了“N”的序列[β]. | (α − β)/β (%). |
|---|
| 镓25 | 1367889 | 1366030 | 0.136088 |
| 镓3 | 110204947 | 110177075 | 0.025297 |
| Tn1型 | 20304845 | 20315377 | 0.051869 |
| Tn15型 | 6235253 | 6236842 | 0.025484 |
| AgX公司 | 21470369 | 21477782 | 0.034527 |
| 氯化银 | 48065434 | 48071405 | 0.012423 |
| 霍伊 | 25653559 | 25653447 | 0.000437 |
| Ho5公司 | 177695253 | 177695218 | 0.000020 |
| 染色体. | 删除'N'的序列[α]. | 过滤了“N”的序列[β]. | (α − β)/β (%). |
|---|
| 镓25 | 1367889 | 1366030 | 0.136088 |
| 镓3 | 110204947 | 110177075 | 0.025297 |
| Tn1型 | 20304845 | 20315377 | 0.051869 |
| Tn15型 | 6235253 | 6236842 | 0.025484 |
| AgX公司 | 21470369 | 21477782 | 0.034527 |
| 氯化银 | 48065434 | 48071405 | 0.012423 |
| 霍伊 | 25653559 | 25653447 | 0.000437 |
| 霍5 | 177695253 | 177695218 | 0.000020 |
当考虑长度范围为n个=1,仅表示静态计数米= 41州,最多n个=8,表示系统的动力学米= 48(65 536)个州。对于箱子计数,使用一个底座滑动窗口(即移动一个底座和重叠n个采用-1个连续碱基)方法。
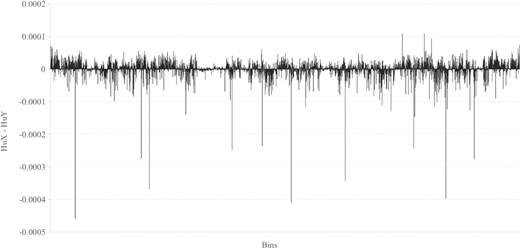
图1显示了人类X和人类Y染色体直方图相对频率之间的差异n个=6。大量的箱子(4096)直观地帮助理解两条染色体DNA碱基序列的差异。这些差异是我们研究基因组DNA并找出是否会出现一些高级结构信息的主要动机。
图1。
人类X和Y染色体直方图相对频率的差异n个=6(4096个箱子)。
简而言之,对于我们的DNA序列分析过程,我们采用了(i)直方图,将T、C、A、G符号转换为数值;(ii)通过n个-元组序列;(iii)使用相关方法进行序列相似性比较;以及(iv)数字序列中隐藏模式的识别以及这些模式的后续高级可视化。
在这项研究中,我们证明了DNA序列分析的四阶段方法能够揭示种内或种间核染色体之间意外的结构模式。结果还揭示了染色体/物种之间的重要高层关系,表明了所提方法的优点,并激励了涉及更完整和广泛的DNA数据以及其他补充科学工具的进一步研究。
2方法
从加州大学圣克鲁斯基因组生物信息学网站下载DNA数据(FASTA格式)后,对每个DNA染色体序列进行处理,以删除“N”符号,并将所有碱基符号转换为{A、C、G、T}字母表。
对于箱子计数,考虑了两种可能的方法,即无重叠窗口和部分重叠窗口n个基本序列。几项测试表明,这两种方法都会产生类似的结果,尽管在处理较小的染色体时会出现一些细微的差异。因此,为了获得更稳健的计数,单基滑动窗口(即一个基的移位和n个−1个连续基地)。为此,我们开发了“文学家'应用程序,在中可用补充材料,它需要所选序列长度作为参数(n个)并过滤DNA序列文件,生成相应的直方图文件。
获取给定值的直方图后n个和一组染色体,我们分析的第二步是评估它们的相似性。这种任务有很多方法(Chaa和Srihari 2002;凌和冈田2006;沃曼等。1985). 最后,我们得到了一个相关矩阵S公司= [秒ij公司],其中秒ij公司定义为秒ij公司=(f)(H(H)我,H(H)j个) ∧我,j个= 1,…,n个(秒ij公司=秒吉∧我,j个= 1,…,n个;秒ii(ii)= 1 ∧我= 1, …,n个)其中H(H)我和H(H)j个是两个长度直方图米,函数(f)(H(H)我,H(H)j个)为实值且0≤秒ij公司≤ 1. 由于对定性相似性感兴趣,我们选择了一种方法来测量任意两个直方图之间匹配的等级部分。因此,我们采用了统计“Kendallτ”秩相关法(肯德尔,1938年),它计算两个直方图的排名之间的对应关系,并根据总体上的“一致对”、“不一致对”和“联系”的数量评估其重要性{H(H)我(一),H(H)j个(一)}和{H(H)我(b条),H(H)j个(b条)}对,其中一,b条= 1,…,米和一<b条我们注意到Kendallτ相关法的计算成本很高。因此,我们采用了中描述的高效算法克里斯滕森(2005)). 为了生成相关相似性矩阵文件,我们开发了金雀花'应用程序,也可在补充材料,它需要所选序列长度作为参数(n个)以及直方图文件,生成相应的相关矩阵文件。
分析的第三步是揭示相关矩阵数据中的嵌入模式。为此,我们首先考虑多维缩放(MDS)技术(Borg和Groenen,2005年;考克斯和考克斯,2001年;克鲁斯卡尔和维什,1978年;谢泼德,1962年;曾等。, 2008). MDS是一种数学工具,它在低维地图中表示一组数据点,其相似性(或替代距离)通过对称矩阵在高维空间中定义S公司= [秒ij公司]. 在相似度和经典MDS的情况下,矩阵主对角线由一个组成,而其余矩阵元素必须遵守0≤的限制秒ij公司≤ 1 (秒ij公司≥ 0),我,j个= 1,…,米通常,为了便于图形表示,使用2D和3D MDS图,并通过Shepard和/或应力图验证其一致性。为了创建MDS图,我们选择了GGobi包,因为它简单、快速和健壮(网址:http://www.ggobi.org).
除MDS图外,相关性矩阵可用于生成链接最相关项的图形,以便可视化它们之间的潜在模式。因此,我们选择了GraphViz包(http://www.graphviz.org)这是一个开源软件,用于将结构信息表示为抽象图和网络图,以创建显示染色体或物种如何相关的有向图。相关矩阵也可以用于生成树形图,树形图描述了由某些层次聚类方法产生的聚类。为了在本研究中生成树状图,我们选择了MultiDendograms分层聚类包,该包配置为“内部联合”聚类方法(Fernández和Gómez,2008年).
这个补充材料包含我们自定义开发的应用程序的源代码和可执行文件,以及输入数据文件(序列直方图)、输出数据文件(用于GGobi、GraphViz或MultiDendogram包、图像和视频)和一些实用程序(主要是用于UNIX或GNU/Linux平台的转换脚本)。
3结果
3.1核染色体的相关分析
使用长度序列为18个物种的核染色体生成384组直方图后n个={1,…,8},对于的每个值n个,我们应用了Kendallτ相关方法(肯德尔,1938年)生成相应的384×384S公司原子能的如前所述,为了创建MDS图,我们使用GGobi包。
图2显示了18个物种的384条核染色体的3D MDS序列长度图n个= {3, 6}. 即使长度低至n个=3,3D图案很容易被注意到。同样的模式也可以在n个=6个绘图,但更好地定义和分隔。
图2。
384条核染色体的3D MDS图n个=3(左侧)和n个=6(右侧)。
图3显示了对18个物种的384条核染色体进行序列长度3D MDS的结果n个=8,有两个不同的二维投影。中的图像分析图2和三显示出空间模式的出现与染色体分组到物种中密切相关。尽管MDS图是为n个={1,…,8},我们注意到n个(7或8)生成的MDS图具有更好的染色体分组和更清晰的物种分离。我们还观察到,染色体分组和物种分离的质量随着n个增加,周围稳定n个= 8.
图3。
384条核染色体的3D MDS图的两个不同视图n个=8。阴影点代表核染色体,有些标记为可读性。
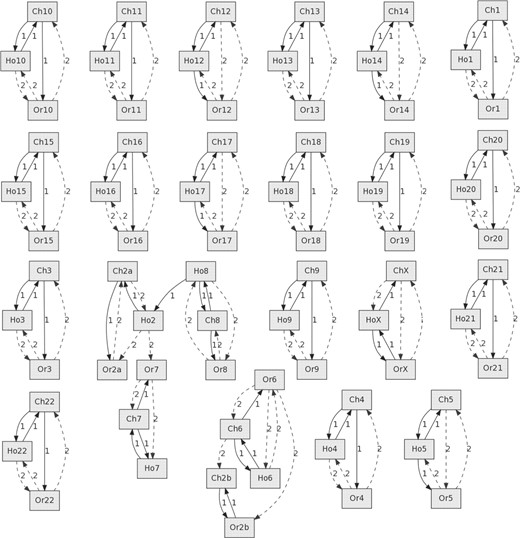
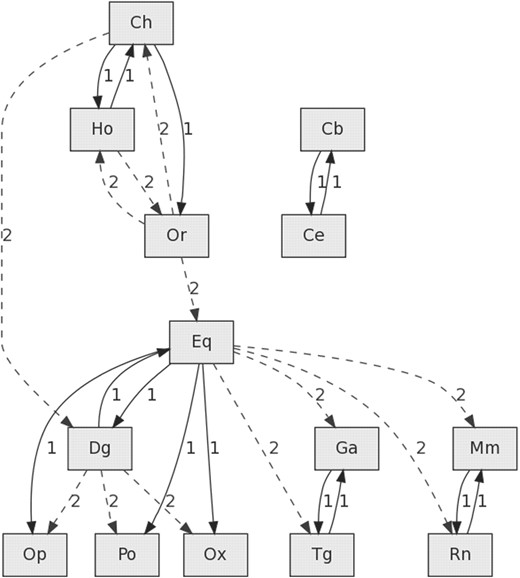
相似矩阵S公司原子能的也可以用于生成连接最相关染色体的图形,以便可视化它们之间的潜在结构模式。的有向图图4由GraphViz生成的,显示了{Ho、Ch和Or}的染色体如何与n个=8。通过箭头结束的连续线(标记为“1”)指向另一条染色体的染色体代表与“指向”染色体最相关的染色体。如果这条线是虚线(标记为“2”),那么指向的染色体是相关性第二大的染色体。在图4,只有相关性≥95%的链接可见,这表明具有相同“数目”的{Ho、Ch、Or}染色体之间的相关性更强,尽管也有一些例外:例如,{Ch、Or}中的Y染色体和2a、2b染色体。
图4。
{Ho,Ch,Or}染色体的两条最相关的染色体图n个=8。灰色矩形:染色体,链接第页:相似性连接第页在两条染色体之间。
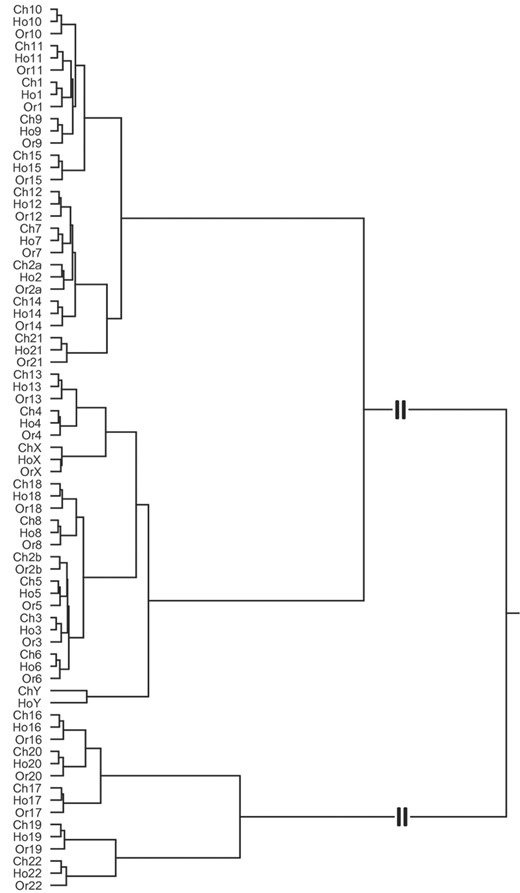
这个S公司原子能的相似度矩阵还可以用于生成dendogram,这是一个树状图,描述由某些层次聚类方法产生的聚类。丹麦图5由MultiDendograms分层聚类包创建(Fernández和Gómez,2008年),使用n个=8和{Ho,Ch,Or}染色体。在图5,我们可以观察到{Ho,Ch,Or}聚类的几个级别(例如,位于图5).
图5。
{Ho,Ch,Or}染色体的树突状图n个=8(压缩了最右边的聚类)。
3.2核基因组的相关性分析
到目前为止,我们分别关注核染色体。另一种方法是将核基因组作为一个整体来考虑。这可以通过组合每个物种的所有核染色体直方图来实现,从而生成相应的“全球核直方图”。随后,将Kendallτ相关方法应用于18个物种的全球核直方图,得到物种相似矩阵S公司全球性的生成,然后进行处理以生成一些高级可视化。
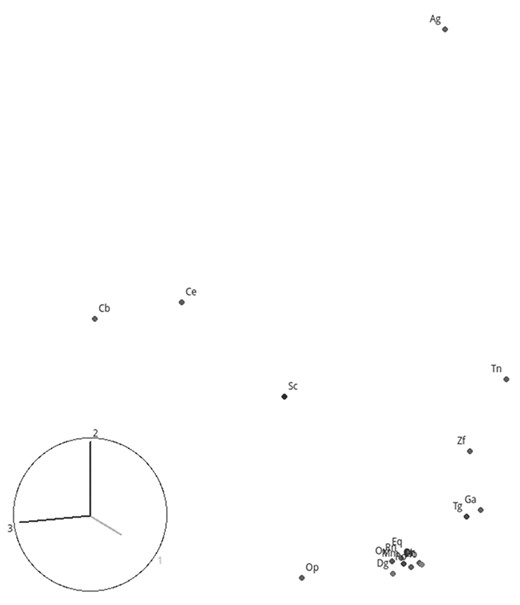
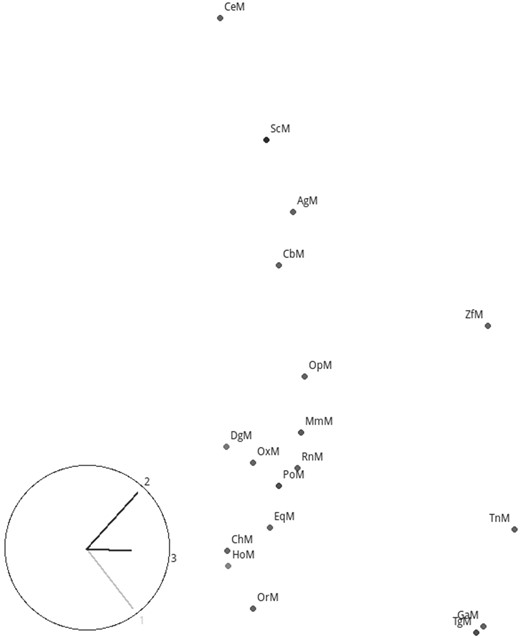
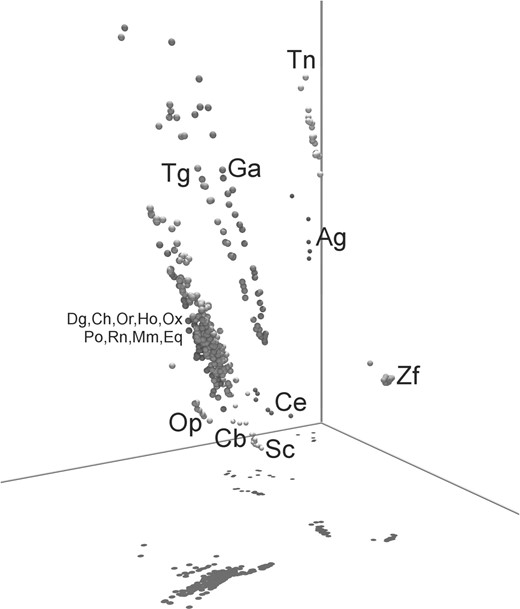
图6显示了18个核基因组序列长度的3D MDS图n个=8,显示了涉及物种的模式的清晰空间组织,尤其是哺乳动物,除了负鼠外,它们都是高度聚集的。我们还注意到,鸟类{Ga,Tg}以及鱼类{Zf,Tn}彼此距离很近,非脊椎动物物种与脊椎动物相距很远。
图6。
18个物种核基因组的3D MDS图n个=8。阴影点表示物种,所有标记都是为了可读。
图7描述了18个物种中的15个物种如何在初级水平(连续线,标记为“1”)和次级水平(虚线,标记“2”)上彼此最相关。只有相关性≥75%的链接才可见。图中还显示了五个物种簇:{Ho、Ch、Or}、{Cb、Ce}、}Mn、Rn}、[Ga、Tg}和{Eq、Dg、Op、Po、Ox}。
图7。
两个最相关物种的图n个=8(相关性≥75%)。灰色矩形:染色体,链接第页:相似性连接第页在两个物种之间。
3.3线粒体基因组相关性分析
线粒体基因组的DNA序列计数在13000-86000nt之间,而核基因组的DNA顺序计数在12000-35000000nt之间。这意味着线粒体数据比核基因组小得多。
使用可变长度序列为18个物种的线粒体染色体生成18组直方图后n个,对于的每个值n个我们应用Kendallτ相关方法生成相应的18×18相似矩阵S公司米托.
图8揭示了对18个物种的线粒体染色体进行3D MDS的结果n个=8。它还显示了涉及线粒体染色体的模式的清晰空间组织,特别是哺乳动物、灵长类和鸟类。
图8。
18个物种线粒体基因组的3D MDS图n个=8。阴影点代表物种,所有标记都是为了便于阅读。
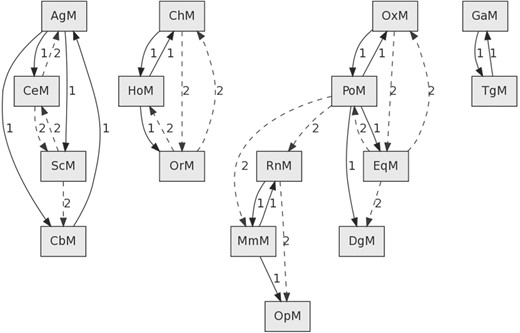
图9描述了18条线粒体染色体中的一些是如何在一级(连续线)和二级(虚线)水平上彼此联系最紧密的。仅显示相关性≥30%的链接n个=8。我们可以观察到图中有五个主要的染色体簇:从左到右的无脊椎动物、灵长类、{Rn、Mm、Op}、{Ox、Po、Eq、Dg}和{Ga、Tg}。
图9。
两条最相关的线粒体染色体图n个=8(相关性≥30%)。灰色矩形:染色体,链接第页:相似性的连接第页在两条染色体之间。
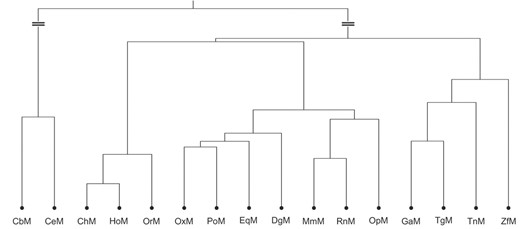
丹麦图10描述了使用“内部联合”聚类方法对线粒体基因组进行的层次聚类,为了清晰起见,对最顶部的聚类进行了压缩。只有两个或两个以上的物种组合在一起,才能在图10(即10种哺乳动物、2种鸟类、2种鱼类和2种线虫)。
图10。
基于线粒体基因组的Dendogramn个=8(压缩了最顶部的群集)。
4讨论
我们描述了一种新的DNA分析方法,该方法以输入的全基因组染色体序列为基础,然后使用无比对序列技术从直方图相关性中提取高级信息。此信息用于生成与染色体或物种相关的几种表格和/或图形输出。
在本研究中,一个重要参数是单词长度n个,用于序列处理和直方图构造步骤。这个n个=1例仅仅是{T,C,a,G}的计数,但当n个从2到8,相应的3DMDS图揭示了与染色体相关的越来越明显的空间和结构模式,如图所示图11对于n个= 8.
在这个3D渲染中(使用“地板上的阴影”),我们可以观察到单个染色体和许多空间分组:一个包括所有哺乳动物的大分组(Op稍微分开);Ga和Tg分组;与Ce和Cb分组。鱼类物种Zf和Tn在空间上彼此相距较远。值得注意的是,大多数物种都存在空间结构:除{Cb、Ce、Sc、Zf}外,其余物种都显示出染色体的“线性组织”,物种之间的“线”大多平行。在大多数物种中,有性染色体与相应物种的“线”距离更远,但这也发生在其他非有性染色体上。
我们无法立即解释这种显著的结构,但它可能与染色体和基因组中较高水平的信息有关。从染色体DNA中出现的这种明显的新结构知识似乎是我们研究方法所独有的,并为研究(Seitz,2007年).
我们还使用18个物种的核染色体和线粒体染色体进行了一项研究,在染色体、基因组和物种水平上得出了以下结果:
核和线粒体染色体的3D MDS图显示,基于全染色体DNA数据的无比对测序和数据挖掘,而不是基因或其他小序列,出现了种内和种间空间模式。
核染色体和线粒体染色体的有向图表明,染色体关系可以从直方图相关性以及基因组/物种关系中推导出来。
基于染色体的dendogram显示,直方图相关性可以用于计算描述染色体和物种层次聚类的图表。
在的树状图中图5,我们观察到人类、黑猩猩和猩猩的染色体聚集成三个主要组:成为最后一个最“不同”的人。对于16+20+17+19+22集群分离,目前尚无直接解释。
使用分割成染色体的DNA碱基序列,这项研究还显示了获得高级染色体信息的可能性,就像大多数种间相关染色体一样(图4和7).
看看丹麦图10考虑到线粒体基因组中存储的少量信息,似乎可以将其视为一种物种的特征。此dendogram描述了一个假设的系统发育树,在定性方面与Wildman公司等。(2007),墨菲等。(2007年),赵和布尔克(2009),Prasad和Allard(2008年),埃伯斯贝格尔等。(2007),邓恩等。(2008)和希利尔等。(2004年). 值得注意的是,上述作者描述的生成系统发育树的过程使用了DNA碱基序列的一部分,而不是像本研究中提出的那样的“转换”版本。还应该提到的是,使用的是整个DNA染色体序列,而不仅仅是像基因或其他部分序列这样的部分。
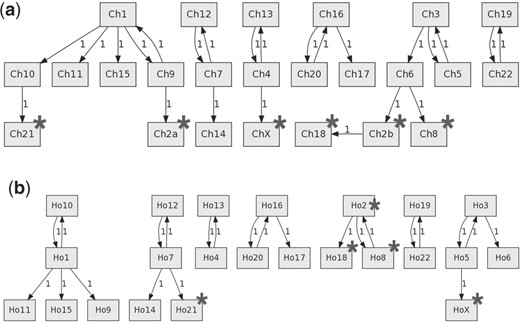
使用所述的直方图相关方法,还可以生成显示最具种内相关染色体并突出差异的输出。图12黑猩猩和人类的结构差异用星号标记。
图12。
最相关的核染色体图n个黑猩猩和人类为8。灰色矩形:染色体,连接:两条染色体之间相似度最高的连接。星号标志着染色体关系的主要差异。(一)黑猩猩(n个=8,相关性≥90%)。(b条)人类(n个=8,相关性≥90%)。
在图12我们可以观察到,染色体Ch21/Ho21与不同的染色体组相连(黑猩猩为1+9+10+11+15,人类为7+12+14),以及ChX/HoX(黑猩猩4+13,人类为3+5+6)等。
所有描述的结果都有助于我们认识到,核基因组和线粒体基因组包含允许染色体分析和其他高级分析的结构信息,以及物种相关研究(例如进化/比较基因组学和系统发育树构建)。
4.1未决问题和未来工作
在这项研究中,我们使用了不完整的染色体信息,如UCSC基因组生物信息学网站所述。对于中提到的许多物种表1,有相当数量的DNA序列数据尚未附加到染色体上,或者与特定染色体相关,其位置尚未定义。这种信息的不确定性是不可取的,并且容易导致误导性的结果,而这些结果不是由所采用的数学和计算工具引起的。
对于数据处理,对于DNA序列长度,我们使用n个= 1,…, 8. 虽然值较大n个是允许的,应该注意的是直方图箱的总数为米= 4n个而且,有一个很大的米,对于较小的染色体,大多数直方图仓可能变为零。例如,对于所考虑的最小核染色体(Sc1,具有231k碱基)n个=8那么米=65 536,每个直方图箱平均有3.5个样本,但对于Sc1n个=10,每个直方图箱的平均样本数降至0.2(即4/5箱等于零)。从收集的经验证据来看n个是6、7和8。进一步的研究应该解决并澄清这个问题。
Kendallτ秩相关方法已被证明足以生成相关矩阵S公司,但其他相关方法也进行了测试。这一问题将是进一步研究和评估的主题。
最后,当完整的基因组数据可用时,应重复该研究,并将其扩展到更多物种,最终实现更大的“生物多样性”。一旦有更多的DNA物种数据可用或更新,这个问题就会得到解决。
致谢
我们感谢以下组织允许访问基因组数据:
作者感谢审稿人的评论和改进本文的机会。
利益冲突:未声明。
参考文献
, . , 现代多维标度理论及其应用
, 2005
2美国
施普林格
, . 关于直方图之间距离的测量
, 图案识别
, 2002
,体积。 35
(第1355
-1370
) . 计算Kendallτ的快速算法
, 计算。斯达。
, 2005
,卷。 20
(第51
-62
) , . , 多维缩放
, 2001
2美国
查普曼和霍尔/CRC
,等人广泛的系统发育取样提高了动物生命树的分辨率
, 性质
, 2008
,卷。 452
(第745
-750
) 等绘制人类遗传祖先
, 分子生物学。埃沃。
, 2007
,卷。 24
(第2266
-2276
) , . 用多端程序解决凝聚层次聚类中的非唯一性
, J.分类。
, 2008
,卷。 25
(第43
-65
) 等鸡基因组的序列和比较分析为脊椎动物进化提供了独特的视角。国际鸡基因组测序协会
, 性质
, 2004
,卷。 432
(第695
-716
) . 秩相关的一种新度量
, 生物计量学
, 1938
,卷。 30
(第81
-89
) , . 多维缩放
, 圣哲大学社会科学定量应用系列论文
, 1978
伦敦
Sage出版物
, . 直方图比较的扩散距离
, 2006年IEEE计算机学会计算机视觉和模式识别会议记录
, 2006
纽约
(第246
-253
) 等DNA的分数动力学
, 通用非线性科学数字模拟。
, 2010
,卷。 16
(第2963
-2969
) 等, . 生命之树网络项目。林奈三百年:无脊椎动物分类学进展
, 动物分类学
, 2007
,体积。 1668
(第1
-766
) 等利用基因组数据揭示胎盘哺乳动物系统发育的根源
, 基因组研究。
, 2007
,卷。 17
(第413
-421
) . 遗传学:什么是基因?
, 性质
, 2006
,卷。 441
(第398
-401
) , . 利用大型比较序列数据集确认哺乳动物的系统发育
, 分子生物学。进化。
, 2008
,卷。 25
(第1795
-1808
) , . , 生物系统学:原理与应用
, 2009
2美国伊萨卡
康奈尔大学出版社
. . 蛋白质-DNA相互作用的分析
, 生化工程生物技术进展。
, 2007
柏林/海德堡
施普林格
. 近似性分析:具有未知距离函数的多维标度
, 心理测量学
, 1962
,卷。 27
(第219
-246
) 等大规模基因组数据集的多维标度
, BMC生物信息学
, 2008
,体积。 9
第页。 179
, . 无对齐序列比较-综述
, 生物信息学
, 2003
,卷。 19
(第513
-523
) ,等人多维直方图的距离度量
, 计算。视觉。图表。图像处理。
, 1985
,卷。 32
(第328
-336
) 等基因组学、生物地理学和胎盘哺乳动物的多样性
, 程序。美国国家科学院。科学。美国
, 2007
,卷。 104
(第14395
-14400
) , . 哺乳动物系统发育中基因组重排的恢复
, 基因组研究。
, 2009
,卷。 19
(第934
-942
)
作者注释
©作者2011。牛津大学出版社出版。保留所有权利。有关权限,请发送电子邮件至:journals.permissions@oup.com