wwPDB 2023新闻
目录
12/18/2023
使用ORCiD改善存款人体验
 OneDep中位于的ORCiD登录按钮用红色圈出
OneDep中位于的ORCiD登录按钮用红色圈出自2023年3月,储户可以使用他们的ORCiD访问OneDep系统。此身份验证方法使联系人作者能够在不共享密码的情况下访问会话。将ORCiD与OneDep一起使用会返回一个汇总表,其中包含为联系人作者提供ORCiD的所有条目。
wwPDB改进了摘要表:
- 两个新的列显示了实验方法和保留到期日期
- EM和NMR的EMDB和BMRB登录代码显示在条目ID旁边
- 所有列现在都可排序
虽然仍然可以使用存款ID和密码登录OneDep,但我们鼓励存款人使用ORCiD来改善用户体验。
问题或反馈?联系人deposit-help@mail.wwpdb.org.
12/12/2023
具有新配体的PDB条目现在仅以PDBx/mmCIF和PDBML文件格式分发
使用PDB三字符化学成分IDPDB已经开始为OneDep系统中的CCD ID发布五个字符的字母数字登录代码。为了避免与当前的四字符PDB ID混淆,不使用四字符代码。由于传统PDB文件格式的限制,包含新的五字符ID代码的PDB条目以PDBx/mmCIF和PDBML格式分发(请参阅以前的公告)。旧版PDB文件格式不支持包含这些扩展ID的PDB条目(请参阅以前的公告).
此外,wwPDB还保留了一组CCD ID:01-99、DRG、INH、LIG,这些ID将永远不会在PDB中使用。这些保留代码可用于结构测定期间的新配体,以便在沉积时识别为新配体并在生物化期间添加到CCD中。
wwPDB要求用户和软件开发人员审查他们的代码,取消当前对PDB和CCD ID长度的任何限制,并允许使用PDBx/mmCIF格式的文件。https://github.com/wwPDB/extended-wwPDB-identifier-examples具有扩展PDB和/或CCD ID的示例文件可通过GitHub获得协助修改代码要了解PDBx/mmCIF,请访问https://mmcif.wwpdb.org/.
如需更多信息,请联系我们info@wwpdb.org.
11/22/2023
PDB档案中FTP文件下载协议的弃用
多年来,用于文件下载的FTP协议越来越不受欢迎,取而代之的是HTTP/S。HTTP/S有许多优点,包括速度、无状态、安全性(HTTPS)和更好的支持。重要的是,在过去2-3年中,主要的web浏览器(Chrome和Firefox)已经放弃了对FTP协议的支持,这实际上已经停止了对非技术用户的FTP协议。
鉴于互联网上的大多数文件下载活动已经转移到HTTP/S,wwPDB计划在2024年11月1日废除FTP下载协议(参见之前的公告).
将继续维护对RSYNC协议的支持,该协议提供了额外的功能。
如前所述,wwPDB支持特定于协议的DNS名称:
请联系info@wwpdb.org有任何问题。
11/14/2023
多肽残基的主干注释和标准化现已上线
wwPDB推出了更新的化学成分词典(CCD)数据文件,其中包括标准化的原子命名和对肽残基中蛋白质骨架和末端原子的附加注释。包含这些更新的CCD的条目已相应更新。这提高了PDB数据的可查找性和互操作性,并为使用更新的肽残基注释开辟了新的机会。
如前所述,新的数据项被添加到CCD文件中,用于肽链组分,以标记形成主链、N或C末端基团的原子。三个新的CCD数据项被添加到CCD类别_chem_comp_atom中,作为pdbx_backbone_flag、pdbx_N-末端_flag和pdbx_C-末端_flag,分别标记主干、N-末端和C-末端原子。
此外,我们对CCD文件中肽骨架原子的原子命名进行了标准化,以遵循标准惯例。这遵循我们的文档确保羧基、氨基和侧链连接碳(C-α)的原子命名遵循标准原子命名。这样就可以清楚地识别整个档案中肽残基的主链原子。
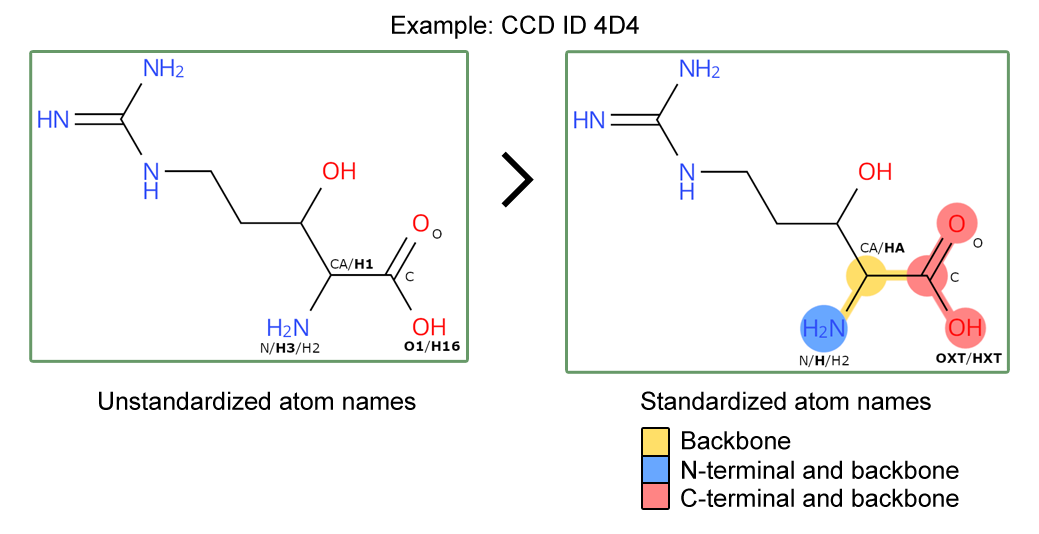 更新后的肽CCD将具有标准化的原子名称和主链/N和C末端注释。
更新后的肽CCD将具有标准化的原子名称和主链/N和C末端注释。问题或反馈?联系人deposit-help@mail.wwpdb.org.
肽残基化学成分词典修复项目是蛋白质化学修饰(PCMs)和翻译后修饰(PTMs)修复项目的一部分,该项目是一个wwPDB合作项目,主要由PDBe公司在EMBL-EBI公司由BBSRC拨款BB/V018779/1资助。
10/18/2023
蛋白质学会海报奖
wwPDB基金会在2023年蛋白质学会年度研讨会(7月13日至16日,马萨诸塞州波士顿)上为优秀学生海报颁奖。
 泰勒·M·拉弗拉姆
泰勒·M·拉弗拉姆 凯文·拉米雷斯
凯文·拉米雷斯泰勒·M·拉弗拉姆对于
靶向锚蛋白重复序列的小分子抑制剂的特异性研究
Taylor M.Laflamem(1)、Emma I.Kane(1),Dipti Kanabar(2)、Tejashri Chavan(2),Aaron Muth(2)和Donald E.Spratt(1)
1) 克拉克大学;2) 圣约翰大学
凯文·拉米雷斯对于
检测SARS-CoV-2病毒的宿主蛋白诱饵荧光传感器
凯文·拉米雷斯(1、2)、大卫·布扎达(3)、阿尔扬·贝恩斯(1、2中)、穆拉德·萨奇(1、二)、尤金尼奥·森蒂斯·巴斯克斯(3),帕特里夏·利旺(1、2-)和维克托·穆尼奥斯(1,2-)
1) 生物工程系,2)加州大学默塞德分校细胞和生物分子机器CREST中心,3)西班牙圣地亚哥大学
非常感谢蛋白质协会的组织者和海报奖评委使这些奖项成为可能。
wwPDB基金会成立于2010年,旨在筹集资金支持世界卫生大会的推广活动。基金会筹集资金,帮助支持PDB50活动、研讨会和教育出版物。该基金会被特许为501(c)(3)实体,专门用于科学、文学、慈善和教育目的。
wwPDB基金会感谢我们的行业赞助商:Discengine、OpenEye Scientific、Roivant Sciences、Rigaku和ThermoFisher Science也可提供个人赞助。
考虑支持PDB未来50年的开放、合作和教育精神向wwPDB基金会捐款.
10/11/2023
IUCr颁发海报奖
wwPDB基金会于8月22日至29日在澳大利亚墨尔本举行的第26届国际晶体学联合会大会上为杰出的学生海报颁奖。
 阿基拉·皮拉皮提亚
阿基拉·皮拉皮提亚 莉莉亚娜·格雷罗
莉莉亚娜·格雷罗阿基拉·皮拉皮提亚对于
肠致病性大肠杆菌毒素EspC的晶体结构揭示了对抗腹泻感染的方法
阿基拉·皮拉皮提亚(Akila Pilapitiya)、莉莲·霍尔(Lilian Hor)、杰森·帕克斯曼(Jason Paxman)、贝戈尼亚·赫拉斯(Begoña Heras)
拉筹伯大学
莉莉亚娜·格雷罗对于
Druging the Undrugable:通过结构生物学揭示磷酸酶的构象景观和配体性
Liliana Guerrero(1)、Ali Ebrahim(1)和Blake T.Riley(1),Minyoung Kim(1、2)、Qingqiu Huang(3)、Aaron D.Finke(3)和Daniel A.Keedy(1、4)
1) 纽约市立大学高级科学研究中心结构生物学倡议2)普林斯顿大学分子生物学系3)康奈尔大学高能同步辐射源(CHESS)4)纽约市立学院化学与生物化学系
非常感谢IUCr组织者和海报奖评委为这些奖项的颁发做出了贡献。
wwPDB基金会成立于2010年,旨在筹集资金支持世界卫生大会的推广活动。基金会筹集资金,帮助支持PDB50活动、研讨会和教育出版物。该基金会被特许为501(c)(3)实体,专门用于科学、文学、慈善和教育目的。
wwPDB基金会感谢我们的行业赞助商:Discngine、OpenEye Scientific、Roivant Scientific、Rigaku和ThermoFisher Scientific也可提供个人赞助。
考虑支持PDB未来50年的开放、合作和教育精神向wwPDB基金会捐款.
10/11/2023
非标准坐标系中晶体结构的修复
总共268个结构条目2022年12月至2023年9月期间,以非标准坐标系或空间组设置存放于PDB的文件已重新修订和发布。这个更新利用现代图形和细化软件,对沉积的实验数据进行改进的模型可视化和验证。
每个重新修订的条目都将原子x、y、z坐标转换为标准晶体学框架。非晶体对称矩阵(如果存在的话)也已被转换以对更新的坐标进行操作。
坐标转换是从REMARK或SCALE记录中提取的,或者从主要引文中发布的转换中提取的。
已仔细检查所有转换后的结构坐标,以确保晶体填料的完整性,并在可行的情况下,根据沉积的结构因子数据进行验证。
原始保存的坐标/矩阵与中以前的主要版本一样,仍然可以访问/下载PDB的版本化存档.
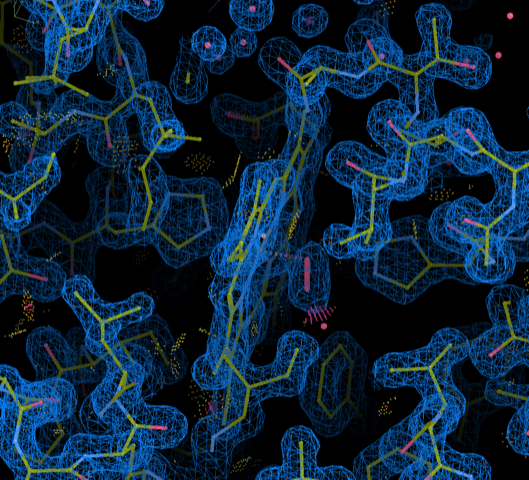 坐标框架转换的肌红蛋白1MBO坐标与使用COOT可视化的2Fo-Fc图。
坐标框架转换的肌红蛋白1MBO坐标与使用COOT可视化的2Fo-Fc图。
10/03/2023
电子显微镜元数据在PDBx/mmCIF文件格式中的分布
2023年10月初,电子显微镜数据库(EMDB)将开始以PDBx/mmCIF文件格式以及现有的XML格式文件分发电子显微镜(EM)元数据。在档案中添加PDBx/mmCIF文件将在元数据服务方式上实现EMDB和PDB之间的对等(EM实验数据vs 3D结构),同时提高两个档案之间的一致性和互操作性。2023年10月之前发布的EMDB条目将在稍后添加PDBx/mmCIF文件。EM元数据文件中的PDBx/mmCIF类别和项目的描述可以在PDBx/mmCIF字典中找到(https://mmcif.wwpdb.org/dictionaries/mmcif_pdbx_v50.dic/Groups/em_group.html). 对于EMDB条目EMD-xxxxx,PDBx/mmCIF文件名将为EMD-xxxxx.cif.gz,它将位于wwPDB存档库中名为metadata(EMD-xxxxxx/metadata)的条目子文件夹中:
10/01/2023
宣布凯尔·莫里斯为EMDB的新团队负责人
wwPDB很高兴地宣布,凯尔·莫里斯(Kyle Morris)已加入EMDB团队,担任新的团队负责人。Kyle在低温电子显微镜领域拥有10年的经验,并将从他的科学研究和低温电子显微镜设备中的工作人员角色中汲取第一手知识。Kyle在eBIC期间开发和协调培训计划的经验将补充EMDB如何继续与社区接触。他对EMDB的愿景包括加强其核心功能,开发与补充数据库的连接,以及为未来低温电子显微镜技术(包括机器学习应用程序)准备档案。
09/12/2023
即将推出:带有新配体的PDB条目仅以PDBx/mmCIF和PDBML文件格式分发
按照目前的增长速度,我们预计到2023年底将用完三个字符的化学成分ID。在此之后,wwPDB将发布OneDep系统中CCD ID的五字符字母数字登录码。为了避免与当前的四字符PDB ID混淆,将不使用四字符代码。由于传统PDB文件格式的限制,包含新的五字符ID代码的PDB条目将仅以PDBx/mmCIF和PDBML格式分发(请参阅以前的公告).
此外,wwPDB还保留了一组CCD ID:01-99、DRG、INH、LIG,这些ID将永远不会在PDB中使用。这些保留代码可用于结构测定期间的新配体,以便在沉积时识别为新配体并在生物化期间添加到CCD中。
wwPDB要求用户和软件开发人员审查代码,以消除当前对CCD ID长度的任何限制,并允许使用PDBx/mmCIF格式的文件。具有扩展CCD ID的示例文件可通过GitHub获得协助代码修订。有关PDBx/mmCIF字典和文件格式的信息,请访问mmcif.wwpdb.org.
如需更多信息,请联系我们info@wwpdb.org.
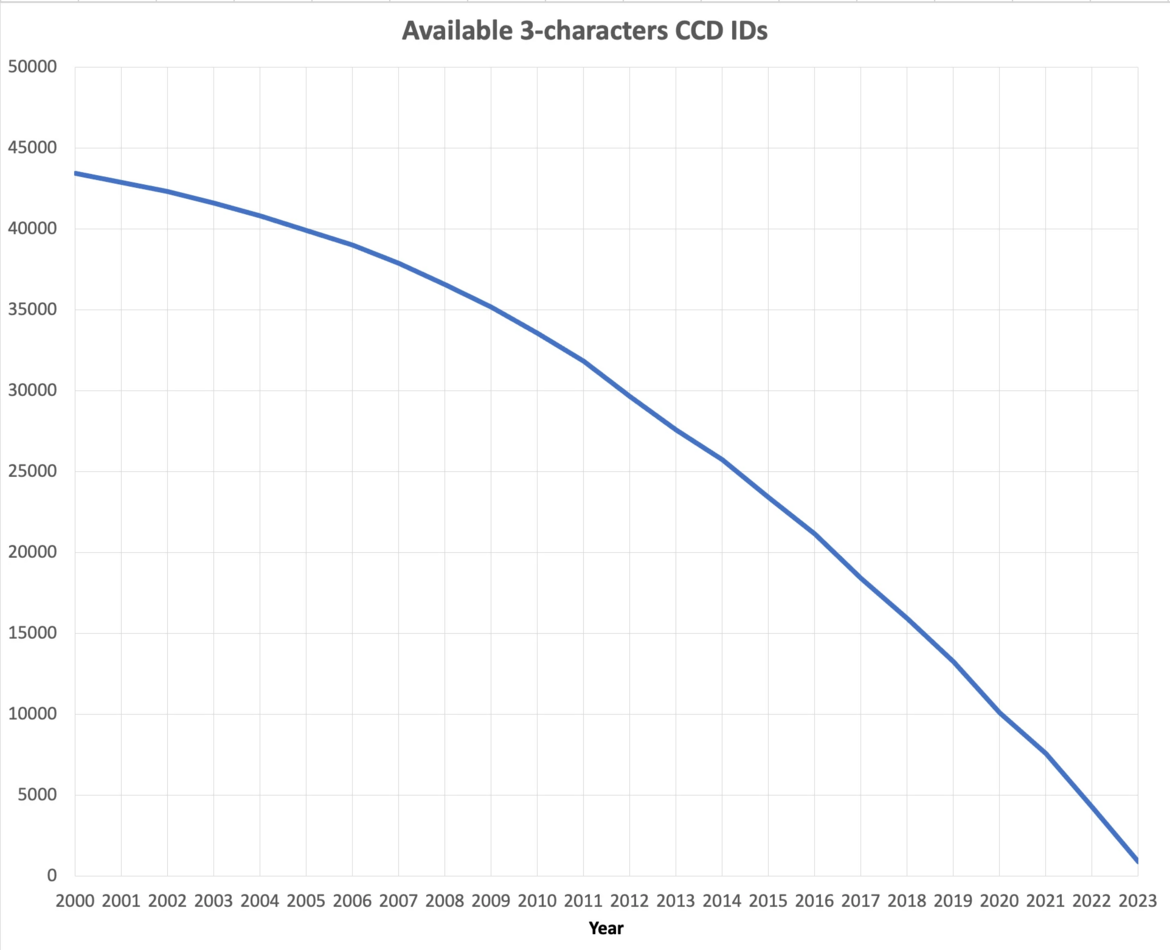 截至2023年9月可用的3字符CCD ID的数量。
截至2023年9月可用的3字符CCD ID的数量。
08/16/2023
IUCr上的wwPDB活动
 wwPDB正在庆祝成立20周年。
wwPDB正在庆祝成立20周年。8月22日至29日,在澳大利亚墨尔本举行的国际结晶学联合会(IUCr)第26届大会和大会上,与来自全球的wwPDB成员会面。
参观展厅内的wwPDB 26号展位,并获得一份特别礼物,以纪念wwPDB成立20周年。了解最新的wwPDB新闻,包括肽残基的最新注释和标准化,如何使用ORCiD存取存款等等。
其他wwPDB活动包括:
- 8月23日星期三,在A090会议上:生物分子SAS和集成方法:标准和验证,Brinda Vallat(RCSB PDB)将谈到PDB-Dev:集成结构归档原型系统
- 8月26日星期六:Stephen K.Burley导演将发表主题演讲蛋白质数据库:从两次大流行到全球大流行再到mRNA疫苗和霍乱病毒
- 8月27日星期日,在A009/A010:结构生物学初级序列预测模型和结构生物学深度学习与人工智能预测模型的使用和比较中,Dennis Piehl(RCSB PDB)将介绍从RCSB蛋白质数据库的人工智能/机器学习中探索实验确定的结构和计算的结构模型
- 8月29日星期二,A014会议:数据库和数据管理,Genji Kurisu(PDBj)将出席日本蛋白质数据库:3D大分子结构数据的亚洲中心和Christine Zardecki(RCSB PDB)将出席RCSB蛋白质数据库:维持活的数字数据资源,实现科学研究和生物医学教育的突破
这个wwPDB基金会将赞助两项奖金,每笔250美元,将颁发给学生生物大分子领域(包括本科生、研究生(硕士)和博士)在国会。
考虑用一个向wwPDB基金会捐款. ThewwPDB基金会成立于2010年,旨在筹集资金支持世界和平发展委员会的推广活动,包括2023年全年颁发的海报奖。
 参观wwPDB展台,领取wwPDC毛巾,庆祝合作20周年
参观wwPDB展台,领取wwPDC毛巾,庆祝合作20周年
07/25/2023
肽残基的最新注释和标准化
2023年10月,wwPDB将推出更新的化学成分词典(CCD)数据文件,其中包含标准化的原子命名以及对肽残基中蛋白质骨架和末端原子的附加注释。包含这些更新的CCD的条目也将相应更新。这将提高PDB数据的可查找性和互操作性,并为使用更新的肽残基注释开辟新的机会。
作为补救过程的一部分,我们将在CCD文件中添加新的数据项,用于肽链组分,以标记形成主链、N-或C-末端基团的原子。三个新的CCD数据项将添加到CCD类别_chem_comp_atom中,分别作为pdbx_backbone_flag、pdbx_n-terminal_flags和pdbx_c-terminal _flag.标记主干、n端和c端原子。
此外,我们将按照标准惯例对CCD文件中肽骨架原子的原子命名进行标准化。这将遵循下面链接的文档中概述的一组规则,确保羧基、氨基和侧链连接碳(C-alpha)的原子命名遵循标准原子命名。这将允许在整个档案中清楚地识别肽残基的主链原子。
有关这项工作的详细信息可从wwPDB网站获得,包括PDBx/mmCIF字典扩展和示例文件(github;肽残留物化学成分词典修复文档).
 更新后的肽CCD将具有标准化的原子名称和主链/N和C末端注释。
更新后的肽CCD将具有标准化的原子名称和主链/N和C末端注释。我们鼓励用于优化或可视化PDB数据的软件包的开发人员审查此信息。
问题或反馈?联系人deposit-help@mail.wwpdb.org.
肽残基化学成分词典修复项目是蛋白质化学修饰(PCM)和翻译后修饰(PTM)修复项目的一部分,该项目是一个wwPDB合作项目,主要由PDBe在EMBL-EBI开展,由BBSRC拨款编号BB/V018779/1资助。
07/09/2023
庆祝wwPDB成立20周年
 2023年,全球蛋白质数据库庆祝成立20周年
2023年,全球蛋白质数据库庆祝成立20周年2003年7月全球蛋白质数据库作为RCSB蛋白质数据库之间的合作伙伴启动(RCSB PDB公司,美国),欧洲蛋白质数据库(PDBe公司),日本蛋白质数据库(PDBj公司)在原子级、生物大分子的三维(3D)结构的基本蛋白质数据库核心档案的管理中,通过大分子晶体学(MX)、核磁共振(NMR)光谱或三维冷冻电子显微镜(3DEM;1,2)实验确定。
自那时起,wwPDB合作范围扩大,包括作为合作伙伴的其他核心档案馆:BioMagResBank(BMRB公司美国)生物大分子和代谢物核磁共振波谱研究得出的光谱和定量数据核心档案;和电子显微镜数据库(EMDB公司,UK)来自3DEM和电子冷冻断层扫描的大分子复合物和亚细胞结构的3D体积和相关信息的核心档案。wwPDB合作伙伴坚持可查找性、可访问性、互操作性和可重用性的公平原则(2),并确保在最宽松的条件下,可以免费访问所有存档数据,并且对使用没有限制Creative Commons CC0 1.0通用许可.
在wwPDB庆祝其运营二十周年之际,wwPDB很高兴欢迎中国蛋白质数据库(PDBc)作为准成员加入该组织。PDBc位于上海的国家蛋白质科学设施,该设施与中国科学院上海高级研究院、上海理工大学SIAIS和iHuman研究院有关联。描述PDBc加入wwPDB过程的公告已提交给Acta Cryst D(PDF格式, 3). PDBc已经接受了wwPDB合作伙伴的培训和支持,预计PDBc将处理在中华人民共和国工作的结构生物学家向所有三个wwPDB核心档案馆提交的大部分(可能全部)证词。
自成立以来,PDB一直是一个国际档案馆,wwPDB的建立确保了这些核心档案馆中包含的宝贵数据将继续被存储、管理和免费提供,以造福世界各地的科学家。
- (1) 海伦·M·伯曼、金·亨里克、中村春树。(2003)宣布全球蛋白质数据库。自然结构生物10: 980. 数字对象标识:10.1038/nsb1203-980
- (2) wwPDB联合会(2019)蛋白质数据库:3D大分子结构数据的单一全球档案,核酸研究47:D520–D528 doi:10.1093/nar/gky949年
- (3) 徐文清(Wenqing Xu)、萨米尔·维兰卡(Sameer Velankar)、阿丹·帕特沃德汉(Ardan Patwardhan)、杰弗里·霍赫(Jeffrey C.Hoch)、斯蒂芬·伯里(Stephen K.Burley)、根吉·库里苏(Genji Kurisu)(2023)宣布启动中国蛋白质数据库(PDBc),成为全球蛋白质数据库(wwPDB)伙伴关系(2023《水晶学报》。D79 792-795 doi:顶部
06/13/2023
wwPDB NMR约束修复符合标准NMR-STAR和NEF格式
核磁共振数据文件在NEF公司和NMR-STAR格式[乌尔里奇,2019年]用于PDB条目,这些条目具有多种不同软件特定格式的NMR数据。该修复项目在单个NMR-STAR/NEF文件中为PDB条目提供统一的NMR数据。wwPDB正在推进NMR-STAR/NEF格式的核磁共振数据(限制、化学位移和可能的峰值列表)的单文件上传,并将在未来逐步取消OneDep中软件特定格式的上传。在过渡期间,OneDep在沉积时将某些约束格式转换为NMR-STAR/NEF,并为用户提供核磁共振抑制验证在wwPDB验证报告中,NMR条目具有指定的化学位移和以下支持格式的合理限制:
琥珀色、生物医学、CHARMM、CNS、青灰色、DYNAMO/TALOS/PALES、GROMACS、ISD、ROSETTA、SYBYL和XPLOR-NIH
根据约束的复杂性以及是否存放了谱峰列表,第一个卷展栏包括NMR条目,这些条目具有支持格式的简单约束,但没有谱峰列表。下一个卷展栏将包括具有光谱峰值列表的条目。符合以下标准的条目是补救范围:
- 指定的化学位移和限制均已交给PDB
- 必须提供有效的拓扑文件或特定注释,以解释特定格式的约束文件(即琥珀色、CHARMM、GROMACS)
- 核磁共振数据中描述的所有原子(指定的化学位移和限制)与模型的原子一致
- 模型和约束匹配之间的序列对齐,允许端子序列扩展
NMR-STAR格式是此NMR约束修复项目中用于处理各种约束的主格式。以“Auth_asym_ID”、“Auth_seq_ID”“Auth_comp_ID”和“Auth_atom_ID”术语开头的数据项,即“_Gen_dist_constraint.Auth_seq_ID_1”,分别指向mmCIF中的等效数据项,“_atom_site.Auth_asym_ID”、“_atom_site.Auth_seq_ID”,“_a汤姆网站.Auth_comp_ID”和“_atom_网站.Auth_atom_ID”。使用“_Assembly”和“_Entity”类别合并完整的原子名称映射信息。结构确定软件使用但NMR-STAR本体未涵盖的NMR约束,如化学平面度、平衡键角、非晶体学对称性等,作为JSON数据存储在“_Other_constraint_list.text_data”标记下,以确保补救过程中不会丢失信息。此外,还提供了有关补救约束的统计数据。NEF数据文件是作为从主NMR-STAR数据文件的尽力转换而提供的。
现有NMR条目的NEF和NMR-STAR格式的新统一NMR数据文件以相同的方式分发到PDB档案的“NMR_data”目录中,用于附带单个NMR数据文档的条目(https://files.wwpdb.org/pub/pdb/data/strustructures/divided/nmr_data/)和BMRB存档(https://bmrb.io/ftp/pub/bmrb/nmr_pdb_integrated_data/coordinates_restraints_chemshifts/remediated_constraints). PDB存档目录“nmr_chemical_shifts”和“nmr_restricts”中的现有数据文件保持不变。
要检索NMR数据,用户应首先在..获取统一的NMR数据文件/nmr_data目录。只有在没有统一的核磁共振数据的情况下,才能使用传统的化学位移和限制。
对于具有传统分离NMR数据文件的新沉积NMR条目,OneDep现在将指定的化学位移和限制统一到一个数据文件中,该文件可以从OneDep上传摘要页面访问。提交后,存款人无法回滚到传统格式。
重新计算wwPDB验证报告,为目标条目提供NMR约束验证。在结构计算期间,不同的软件可以对核磁共振约束进行不同的解释和加权。NMR约束验证软件采用NMR-VTF推荐的更通用方法,独立验证每个模型中每个约束的满意度。当实际结构确定软件采用不同的方法或在结构计算期间分配较少的权重时,这有时会导致大量违规。
05/31/2023
从2023年9月开始,从wwPDB下载PDB存档的DNS名称更改
2022年,wwPDB引入了DNS名称对于PDB存档下载的编程访问:
- 资金转移定价:网址://ftp.wwpdb.org
- HTTPS协议:https://files.wwpdb.org(替换https://ftp.wwpdb.org)
- RSYNC公司:rsync://rsync.wwpdb.org(替换rsync://wwpdb.org)
这个PDB存档下载文档中包含详细信息。
自2023年9月起,wwPDB将开始强制使用这些更新的DNS名称。DNS名称与协议不匹配的URL(例如。,https://ftp.wwpdb.org, ftp://files.wwpdb.org网站)届时将不再工作。
鼓励以编程方式下载PDB存档数据的用户尽快切换到新的DNS名称。对于单个文件下载,首选HTTPS协议(超过FTP)。
请联系info@wwpdb.org有任何问题。
05/16/2023
ls-lR索引文件将于2023年7月12日删除
随着PDB归档的不断增长,列出所有目录内容的文件的大小(当前https://files.wwpdb.org/pub/pdb/ls-lR)将成为长期维护的挑战。2023年7月12日00:00 UTC,wwPDB将从PDB存档中删除以下文件:
- ../pdb/ls-lR
- ../pdb/data/strustructures/ls-lR
- ../pdb/数据/结构/模型/ls-lR
- ../pdb/data/strustructures/models/current/ls-lR
- ../pdb/数据/结构/模型/过时/ls-lR
我们强烈鼓励用户利用文件之前宣布的包含相同数据的(https://files.wwpdb.org/pub/pdb/holdings网站/).
这些库存数据文件提供了存档中数据的快速概览。添加了两个新的实验数据清单文件。这些文件是可扩展的JSON格式,可以在新的/pdb/holdings/archive树下找到。
提供的库存清单包括:
- current_file_holdings.json.gz:发布的PDB条目列表以及PDB核心档案中每个条目的文件类型(例如坐标数据、实验数据、验证报告)。
- refdata_id_list.json.gz:发布的化学参考条目列表,其内容类型(例如,化学成分、BIRD),以及参考文件的最新修改日期。
- released_structures_last_modified_dates.json.gz:PDBx/mmCIF文件最新修改日期的已发布PDB条目列表。
- releasedthential_datalastmodifieddates.json.gz:具有最新修改日期的已发布实验数据文件列表
- obsolete_structures_last_modified_dates.json.gz:PDBx/mmCIF文件最新修改日期的过时PDB条目列表。
- obsoleteexperimental_datalastmodifieddates.json.gz:包含最新修改日期的废弃实验数据文件列表。
- all_removed_entries.json.gz:过时PDB条目的列表,包括条目作者、条目标题、发布日期、过时日期和替代PDB ID(如果有)的信息。
- unreleased_entries.json.gz:保留PDB条目的列表、条目状态、沉积日期和预发布序列信息(如果可用)。
鼓励用户使用这些库存文件。例如,可以使用执行PDB存档更新检查当前文件保存.json.gz或发布的结构最后修改的日期.json.gz在/pub/pdb/holdings/中。
请联系info@wwpdb.org有任何问题。
2023年5月30日更新
05/05/2023
使用新的pdb_extract功能准备沉积
pdb_extract将结构确定程序生成的输出文件中的坐标数据、作者提供的元数据和数据处理信息合并到一个完整的PDBx/mmCIF文件中,该文件可用于与OneDep进行轻松沉积。使用pdb_extract在线表单或易于安装命令行界面经过重新设计(Python)。
坐标数据
上传的坐标文件(PDBx/mmCIF或PDB)将根据PDBx/mmCIF字典进行检查。旧的PDB格式文件将转换为符合OneDep的PDBx/mmCIF数据文件。
元数据
鼓励存款人使用PDBj CIF编辑器轻松编辑模板文件,以包含相应的元数据(序列、结晶条件等)。特定于方法的模板已预加载到PDBj CIF编辑器中:X射线,3DEM公司、和核磁共振。单击左上角菜单(浅灰色窗口小部件图标)以PDBx/mmCIF格式保存编辑的元数据文件。将完成的文件上传到pdb_extract中,以准备提交的单个或多个相关结构。
结构确定输出文件
上传数据处理过程中产生的日志文件,pdb_extract将解析相关衍射元数据。支持来自各种独立软件包以及CCP4和autoPROC管道的日志文件,包括:
- 无目标的
- DIALS(刻度盘)
- d*TREK公司
- HKL-2000
- HKL-3000
- 毫无意义
- 斯卡拉
- 电子秤包装
- XDS公司
- 下2
- X刻度
04/02/2023
从PDB存档中删除ls-lR索引文件
随着PDB存档的不断增长,列出所有目录内容的文件的大小(当前https://files.wwpdb.org/pub/pdb/ls-lR)将成为长期维护的挑战。wwPDB计划于2023年7月12日00:00 UTC从PDB存档中删除此文件。我们强烈鼓励用户使用之前宣布的包含相同数据的文件(https://files.wwpdb.org/pub/pdb/holdings网站/).
这些库存数据文件提供了存档中数据的快速概览。这些文件是可扩展的JSON格式,可以在新的/pdb/holdings/archive树下找到。
提供的库存清单包括:
- all_removed_entries.json.gz:过时PDB条目的列表,包括条目作者、条目标题、发布日期、过时日期和替代PDB ID(如果有)的信息。
- current_file_holdings.json.gz:发布的PDB条目列表以及PDB核心档案中每个条目的文件类型(例如坐标数据、实验数据、验证报告)。
- obsolete_structures_last_modified_dates.json.gz:过时的PDB条目列表,包含有关PDBx/mmCIF文件最新修改日期的信息。
- refdata_id_list.json.gz:发布的化学参考条目列表,其内容类型(例如,化学成分、BIRD),以及参考文件的最新修改日期。
- released_structures_last_modified_dates.json.gz:PDBx/mmCIF文件最新修改日期的已发布PDB条目列表。
- unreleased_entries.json.gz:保留PDB条目的列表、条目状态、沉积日期和预发布序列信息(如果可用)。
鼓励用户使用这些库存文件。例如,可以使用执行PDB存档更新检查当前文件保存.json.gz或发布的结构最后修改的日期.json.gz在/pub/pdb/holdings/中。
请联系info@wwpdb.org有任何问题。
03/26/2023
使用ORCiD访问存款
我们很高兴地宣布,联系人作者现在可以使用ORCiD公司验证OneDep访问权限。此身份验证方法允许每个联系人作者登录OneDep,而无需共享密码即可查看和访问其所有证词。
仍然可以使用存款ID和密码登录OneDep,但只能访问特定存款。
将ORCiD与OneDep一起使用会返回一个条目摘要表,其中为联系人作者提供了ORCiD。用户可以进一步访问每个条目的存款界面,而无需使用存款ID或密码再次登录。
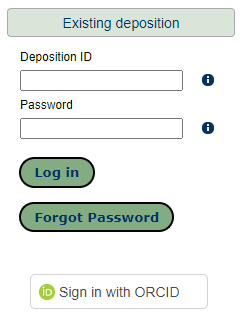 ORCiD登录按钮位于现有登录字段下方。
ORCiD登录按钮位于现有登录字段下方。 使用ORCiD登录后,此OneDep面板将显示所有可用的沉积。
使用ORCiD登录后,此OneDep面板将显示所有可用的沉积。首次与OneDep联系的作者需要验证其电子邮件地址,然后才能创建新的证词,类似于在不登录ORCiD的情况下创建新证词。
请注意,在“管理>联系信息”OneDep页面中添加联系人作者ORCiD将授予此作者访问当前证词的权限。
为OneDep联系人作者提供ORCiD自2018年起强制执行.
03/09/2023
向Olga Kennard博士致敬
wwPDB财团向奥尔加·肯纳德博士致敬听到她去世的不幸消息她在晶体数据库开发方面的开创性工作为现代分子结构数据归档以及随后利用这些数据的科学突破奠定了基础。
奥尔加以建立CCDC(剑桥晶体数据中心)维护剑桥结构数据库(CSD)中的小分子。CSD最初由奥尔加于1965年成立基于其研究小组的活动,已成为世界上小分子有机和金属有机晶体结构的知识库。奥尔加收集了这些数据,以便研究晶体是如何形成的,她的调查是“晶体工程”发展的基础。现在,该数据库包含了来自X射线和中子衍射分析的一百多万个结构,这一精确的三维结构数据库已成为世界各地科学家的重要资源。
随着人们对解决生物分子结构的兴趣和突破的增加,由BNL(Brookhaven National Laboratory)的Walter Hamilton创建了PDB(蛋白质数据库)。奥尔加与沃尔特合作,支持PDB档案的建立,档案最初由BNL和CCDC联合运营(参见1971年PDB公告自然新生物学). 在BNL进行数据处理的同时,CCDC负责组织数据存档,Olga和CCDC在数据存档方面的经验非常有益。如今,使用CCDC软件对PDB中存档的生物结构中包含的小分子进行验证,CCDC软件结合了CSD中嵌入的知识。
 从左至右:海伦·M·伯曼(Helen M.Berman)、珍妮特·桑顿(Janet Thornton)、肖莎娜·沃达克(Shoshana Wodak)和奥尔加·肯纳德(Olga Kennard)于1996年在耶路撒冷举行的PDB-SwissProt研讨会上。
从左至右:海伦·M·伯曼(Helen M.Berman)、珍妮特·桑顿(Janet Thornton)、肖莎娜·沃达克(Shoshana Wodak)和奥尔加·肯纳德(Olga Kennard)于1996年在耶路撒冷举行的PDB-SwissProt研讨会上。奥尔加是一个非常正直和有干劲的人,在计算机真正发展之前的一个时代,她看到了交叉数据分析的价值,从而得出了控制小分子相互作用的原理。很少有科学家能声称他们的工作促成了数千篇论文和调查。奥尔加建立和维持CSD的远见卓识和决心意味着,她是众多其他科学家所支持的巨人之一。
另请参见在CCDC庆祝1924-2023年剑桥结构数据库创始人Olga Kennard OBE FRS博士
03/07/2023
带有扩展CCD或PDB ID的PDB条目将仅以PDBx/mmCIF格式分发
wwPDB与PDBx/mmCIF工作组已制定计划,在未来延长PDB和化学成分词典(CCD)条目的登录代码(ID)长度。旧版PDB文件格式不支持包含这些扩展ID的PDB条目。(请参阅以前的公告)
CCD ID扩展
CCD条目目前由唯一的三字符字母数字ID标识。按照目前的增长速度,我们预计在2024年之前三个字符的ID将用完。在此之后,wwPDB将发布CCD ID的五字符字母数字登录码在OneDep系统中。为了避免与当前的四个字符的PDB ID混淆,将不使用四个字符的代码。由于传统PDB文件格式的限制,包含新的五字符ID代码的PDB条目将仅以PDBx/mmCIF格式分发。
此外,wwPDB还保留了一组CCD ID:01-99、DRG、INH、LIG,这些ID将永远不会在PDB中使用。这些保留代码可用于结构测定期间的新配体,以便在沉积时识别为新配体并在生物化期间添加到CCD中。
PDB ID扩展
wwPDB将把PDB ID长度扩展到以“PDB”为前缀的八个字符,例如PDB_00001abc。每个PDB条目都有一个相应的数字对象标识符(DOI),通常需要将手稿提交给期刊,并由结构作者在出版物中进行描述。自2021年8月以来,所有新条目和重新发布条目的PDBx/mmCIF格式原子坐标文件中都包含了扩展的PDB ID和相应的PDB DOI。
例如,使用4个字符的PDB ID 1abc发布的PDB条目将具有扩展PDB ID(PDB_00001abc)和相应的PDB DOI(10.2210/pdb1abc/PDB),如_database_2 PDBx/mmCIF类别中所列。
回路_
_数据库2.database_id
_数据库2.database_code
_数据库2.pdbx数据库访问
_数据库_2.pdbx_DOI
PDB 1abc PDB_00001abc 10.2210/pdb1abc/PDB
例如,在使用所有4个字符的ID之后,发出带有8个字符的PDB ID PDB_00099xyz的PDB条目:
回路_
_数据库2.database_id
_数据库2.database_code
_数据库2.pdbx数据库访问
_数据库_2.pdbx_DOI
PDB PDB_00099xyz PDB_0099xyz 10.2210/PDB_00099hyz/PDB
在使用了所有四个字符的PDB ID之后,新存放的PDB条目将只发布扩展的PDB标识码,并且PDB条目仅以PDBx/mmCIF格式分发。具有四个字符PDB ID的PDB条目将保持不变。
资源
wwPDB要求用户和软件开发人员审查他们的代码,取消当前对PDB和CCD ID长度的任何限制,并允许使用PDBx/mmCIF格式的文件。带有扩展PDB和/或CCD ID的示例文件可通过github获得,以帮助代码修订,请参阅https://github.com/wwPDB/extended-wwPDB-identifier-examples。要了解PDBx/mmCIF,请访问https://mmcif.wwpdb.org/.
欲了解更多信息,请联系我们info@wwpdb.org.
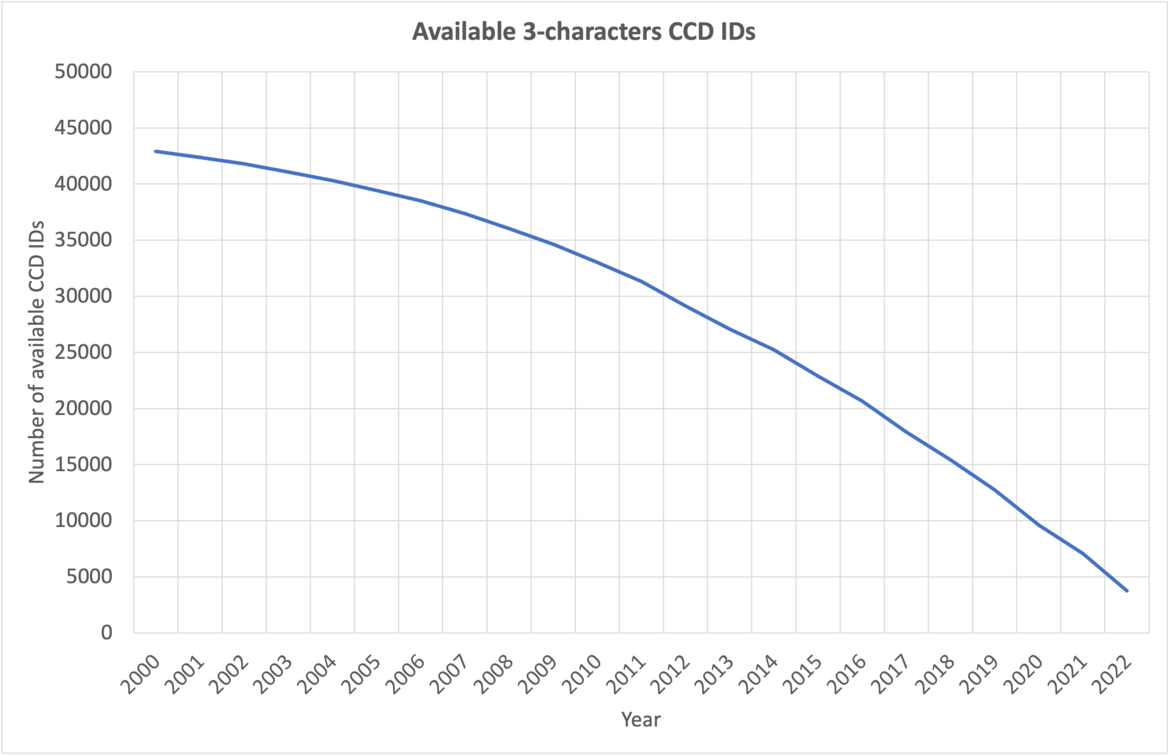 每年可用的3字符CCD ID的数量。
每年可用的3字符CCD ID的数量。
02/14/2023
小角度散布新闻
一个旨在测试和基准测试从PDB坐标建模SAS剖面的不同方法的项目成果已发布:
圆形方法提供了对生物分子小角度散射数据再现性的详细评估,并产生了基准一致性曲线
Trewella,J.、Vachette,P.、Bierma,J.,Blanchet,C.、Brookes,E.、Chakravarthy,S.、Chatzimagas,L.、Cleveland,T.E.、Coweeson,N.、Crossett,B.、Duff,A.P.、Franke,D.、Gabel,F.、Gillilan,R.E.、Graewert,M.、Grishaev,A.、Guss,J.M.、Hammel,M.,Hopkins,J.和Huang,Q.、Hura,G.L.、Irving,T.C.、Jeffries,C.、Jeong,C.、。,Kirby,N.、Krueger,S.、Martel,A.、Matsui,T.、Li,N.,Perez,J.、Porcar,L.、Prange,T.,Rajkovic,I.、Rocco,M.、Rosenberg,D.J.、Ryan,T.M.、Seifert,S.,Sekiguchi,H.、Svergun,D.、Teixera,S.和Thureau,A.、Weiss,T.M.、Whitten,A.E.、Wood,K.和Zuo,X。
(2022)《水晶学报》。D78:1315-1336数字对象标识代码:10月107日/2059798322009184
总共收集并集中分析了五种蛋白质(核糖核酸酶A、溶菌酶、木聚糖酶、尿酸氧化酶和木糖异构酶)的171个SAXS和76个SANS测量值。在此过程中,开发了新的数据比较和合并方法。为此工作生成的数据已存放在SAS生物数据库作为共识数据以及贡献的单个数据集。
此外,还出版了一章,描述了为制定2017年生物分子SAS出版指南所做的工作、SASBDB的建立以及基准项目的演变和成果:
第一章-小角度散射生物分子结构的数据质量保证、模型验证和数据共享
吉尔·特瑞维拉
(2023)酶学方法678:1-22数字对象标识代码:2016年10月10日/bs.mie.2022.11.002
这些出版物反映了wwPDB小角度散射任务组(SAStf)他于2012年首次与主席吉尔·特雷维拉会面。SAStf在这项重要工作的进展中发挥了重要作用,这项工作使得生物分子SAS越来越被接受为主流结构生物学技术。
2023年6月2日
PDB NextGen档案原型现已提供
PDB的下一代归档存储库原型现已可用。名为“NextGen”的归档文件托管PDBx/mmCIF和PDBML格式的结构模型文件文件-下一代.wwpdb.org。此丰富的PDB存档提供了元数据中外部数据库资源的注释,以及PDB主存档中结构模型文件中提供的内容文件.wwpdb.org.
该原型提供了来自外部资源(如UniProt、SCOP2和Pfam)的原子级、残基级和链级序列注释。该映射信息来源于结构与功能、分类和序列集成(SIFTS)项目(https://www.ebi.ac.uk/pdbe/docs/sifts/)是由EMBL-EBI的PDBe和UniProt团队开发和维护的一项服务。每个段的序列映射在_pdbx_sifts_unp_segments和_pdbx_sifts_xref_db_segions类别中提供,残渣级的序列映射为_pdbx_ifts_xfref_db,原子级的序列映像为_atom_site。
PDB NextGen Repository当前每月在本月第一个星期三UTC 00:00更新一次,以后可能会更改。您可以在以下位置访问这些NextGen文件:
数据的结构基于条目ID,带有两个字母的散列代码,即“倒数第三个字符”和“倒数第一个字符的倒数第二个字符”。一旦PDB ID代码扩展到具有PDB_前缀的四个字符以上,该哈希代码将保持一致。
以下是一些示例:
访问条目pdb_00008alyhttps://files-nextgen.wwpdb.org/pdb_nextgen/data/entries/divided/al/pdb_00008aly/该位置提供PDBx/mmCIF和PDBML。对于条目pdb_00008aly:- pdb_00008aly_xyz-encrich.cif.gz
-
pdb_00008aly_xyz-no-atom-enrich.xml.gz
请联系info@wwpdb.org有任何问题。
01/31/2023
增强的启动模型集合
引入了一个新的PDBx/mmCIF类别,即_PDBx_initial_refinement_model,以改进收集到的有关X射线、3DEM和NMR方法起始模型的信息。
将区分实验推导模型和计算模型。如果公开可用,将捕获获得起始模型的资源的来源(例如PDB、AlphaFoldDB、RoseTTAFold等)及其登录代码/标识符。
有关完整定义,请参见pdbx初始定义模型。示例如下:
_pdbx初始定义模型id 1
_pdbx初始定义模型实体列表1
_pdbx_initial_refinement_model.type“实验模型”
_pdbx初始定义模型源名称PDB
_pdbx初始定义模型附加代码3LTQ
wwPDB强烈建议所有PDB用户和软件开发人员审查他们的代码,并将此定义用于未来的应用程序。
01/30/2023
结构预测器:将ModelCIF用于计算结构模型
型号CIF(github)是一个由计算结构生物学家开发的数据信息框架,用于描述由计算方法衍生的大分子结构模型。它为这些蛋白质模型的沉积、存档和公开传播提供了可扩展的数据表示,从而能够向全球用户提供可查找、可访问、可互操作和可重用(FAIR)数据。
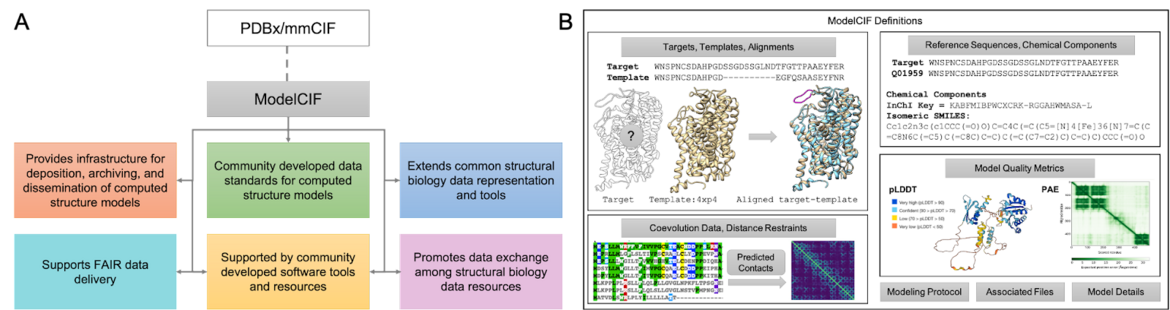 A.PDBx/mmCIF的CIF模型扩展概述。B.ModelCIF数据规范的示意图。ModelCIF包括基于模板和无模板建模中使用的输入数据的定义;大分子序列和小分子成分的参考信息;本地和全球CSM质量指标;以及有关建模协议、CSM分类(从头算、同源性等)和相关文件描述的元数据信息。
A.PDBx/mmCIF的CIF模型扩展概述。B.ModelCIF数据规范的示意图。ModelCIF包括基于模板和无模板建模中使用的输入数据的定义;大分子序列和小分子成分的参考信息;本地和全球CSM质量指标;以及有关建模协议、CSM分类(从头算、同源性等)和相关文件描述的元数据信息。ModelCIF是蛋白质数据库交换/大分子结晶信息框架的扩展(PDBx/毫米到岸价),它是表示实验确定的大分子三维(3D)结构和相关元数据的全球数据标准。PDBx/mmCIF框架及其扩展(例如ModelCIF)由wwPDB与相关社区利益相关者(例如wwPDB ModelCIF工作组.
这种用于表示计算结构模型(CSM)的语义丰富且可扩展的数据框架加快了科学发现的步伐。此外,该数据标准的使用促进了结构生物学数据资源之间的互操作,ModelCIF目前由模型存档,AlphaFold数据库、和MODBASE(国防数据库)存储库。最近向提交了一份手稿生物Rxiv描述ModelCIF的体系结构、内容和治理,以及维护和扩展数据标准的工具和过程[1]。
访问型号CIF GitHub有关此数据信息框架的更多信息。
[1}Vallat B,Tauriello G,Bienert S,Haas J,Webb BM,et al.模型CIF:计算结构模型的PDBx/mmCIF数据表示的扩展。生物Rxiv数字对象标识:10.1101/2022.12.06.518550.
01/10/2023
PDB达到新里程碑:200000多个条目
 存款人:下载这张图片,写下存款结构的数量,并在照片中标记我们
存款人:下载这张图片,写下存款结构的数量,并在照片中标记我们随着本周的更新,PDB档案包含了创纪录的200069个条目。存档已通过2019年150000座建筑物和2014年10万座建筑物.
该中心公共档案馆成立于1971年,由于世界各地结构生物学家的努力,已达到这一关键里程碑,他们提供了实验确定的蛋白质和核酸结构数据。
wwPDB数据中心支持在线访问生物大分子的三维结构,帮助研究人员了解生物医学、农业和生态学的许多方面,从蛋白质合成到健康、疾病再到生物能源。自2014年档案馆发布第10万个结构以来,已经达到了许多里程碑。PDB数据对理解SARS-CoV-2具有重要意义,并为AI/ML预测蛋白质结构技术的发展奠定了基础。整个2021年庆祝了PDB成立50周年.
如今,归档文件相当大,包含超过3000000个与这些PDB条目相关的文件,这些条目需要超过1086 GB的存储空间。PDB结构包含超过18亿个非氢原子。
函数遵循形式
20世纪50年代,科学家首次在原子水平上直接观察蛋白质和DNA的结构。通过X射线晶体学对这些早期三维结构的测定,开创了生物学的新纪元——这是由形式和生物功能之间的密切联系驱动的。由于科学界迅速认识到存档和共享这些数据的价值,蛋白质数据库(PDB)于1971年与位于美国和英国的数据中心进行国际合作,成为生物学领域第一个开放存取的数字资源。
PDB中沉积的第一个结构是肌红蛋白和血红蛋白,这两种氧结合分子的结构由化学诺贝尔奖获得者约翰·肯德鲁和马克斯·佩鲁茨阐明。随着本周的定期更新,PDB欢迎266个新结构进入档案。这些结构与其他对药物发现、生物信息学和教育至关重要的结构结合在一起。
PDB增长迅速,自2011年以来规模增长约160%(每6-8年规模翻倍)。2022年,平均每周向科学界发布275个新结构。研究人员、学生和教育工作者每年都会访问数亿次该资源,目的是探索不同蛋白质之间的相互关系,以阐明基本的生物机制并发现新的药物。
二十年的合作
自成立以来,PDB一直是一个社区驱动的企业,逐渐发展成为生物研究的关键任务国际资源。wwPDB合作伙伴关系于2003年7月与PDBe、PDBj和RCSB PDB建立。如今,合作伙伴包括BMRB(2006年加入)和EMDB(2021年加入)。
wwPDB确保这些有价值的PDB数据得到安全存储、专业管理,并免费提供给全球科学家和教育工作者。wwPDB数据中心与社区专家密切合作,以定义沉积和注释策略,解决数据表示问题,并实施社区验证标准。此外,wwPDB致力于提高结构生物学在越来越广泛的受众中的形象。
提交给档案馆的每个结构在发布之前都由wwPDB工作人员仔细管理。新的沉积物通过附加值注释进行检查和增强,并与其他重要的生物数据相关联,以确保具有广泛背景和兴趣的用户能够发现和解释PDB结构。
wwPDB热切期待着接下来的10万个结构以及这些新数据将带来的宝贵知识。