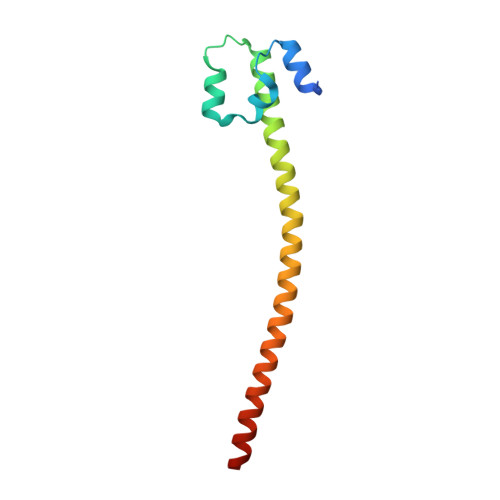
CNC家族转录因子Nrf2转录调控的结构基础。
公共医学 : 36454022 内政部: https://doi.org/10.1093/nar/gkac1102 相关结构的主要引文: 7X5E型 , 7x5英尺 , 7x5克 PubMed摘要: 一些碱性亮氨酸拉链(bZIP)转录因子在其DNA结合域中具有辅助基序,例如CNC家族的CNC基序或小Maf(sMaf)蛋白的EHR基序。 CNC家族蛋白与sMaf蛋白异源二聚体化,以识别CNC-sMaf结合DNA元件(CsMBE),与sMaf-同二聚体竞争,但CNC基序的功能作用仍不明确。 在本研究中,我们报道了与MafG和CsMBE复合物中调节抗应激转录反应的CNC家族蛋白Nrf2/NFE2L2的晶体结构。 CNC基序限制了碱性区关键Arg残基的构象,这些残基与DNA骨架磷酸盐形成广泛接触。 因此,Nrf2-MafG异二聚体对CsMBE的亲和力约为经典bZIP蛋白(如AP-1蛋白)的200倍。 CNC-sMaf异二聚体的高DNA亲和力可能使其能够在目标基因上与sMaf同二聚体竞争,而不会受到其他具有类似序列特异性的低亲和力bZIP蛋白的干扰。