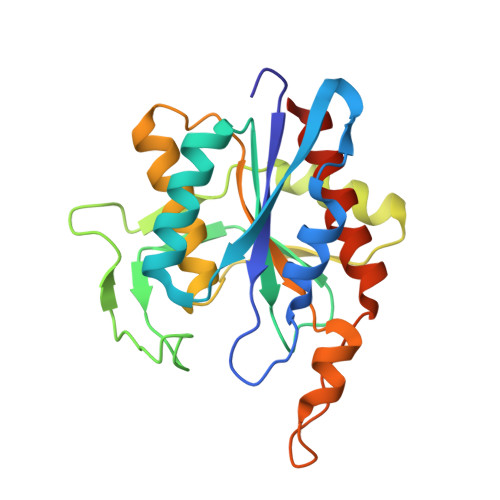
耐辐射球菌吡咯烷酮羧酸肽酶I的晶体结构揭示了L-焦谷氨酸的识别机制。
公共医学 : 30950401 内政部: https://doi.org/10.1107/S2059798319000676 相关结构的主要引文: 5Z47型 , 5Z48型 PubMed摘要: 吡咯烷酮羧酸肽酶(PCP)催化从肽和蛋白质的N端去除一种不寻常的氨基酸L-焦谷氨酸(pG)。 它对原核生物和真核生物中不同肽的功能调节都有意义。 然而,PCP酶的pG-识别机制在很大程度上仍然未知。 这里,来自耐辐射球菌(PCPdr)的PCP I的晶体结构以无pG和结合pG的形式报告,分辨率分别为1.73和1.55º。 PCPdr中的四个原体形成四聚体结构。 负责识别pG残基的残基主要由活性位点附近的柔性环(环a)贡献。 这些残基在所有已知PCP I中都是保守的,包括哺乳动物的PCP I。 环A的Phe9和Phe12与pG的吡咯烷酮环形成堆叠相互作用,而Asn18与pG OE形成氢键。非保留残基的主链Leu71与pG NH和OE形成两个氢键。因此,pG通过范德瓦尔斯和极性相互作用在酶的S1底物亚基中识别, 为肽的pG残基提供特异性。 与先前报道的PCP I结构相比,PCPdr四聚体是一种封闭构象,具有不可接近的活性位点。 结构表明,底物可以通过环A的无序化来访问活性位点。这种无序化也可以通过从S1亚基释放结合的pG产物来阻止产物抑制,从而使酶与新鲜底物结合。