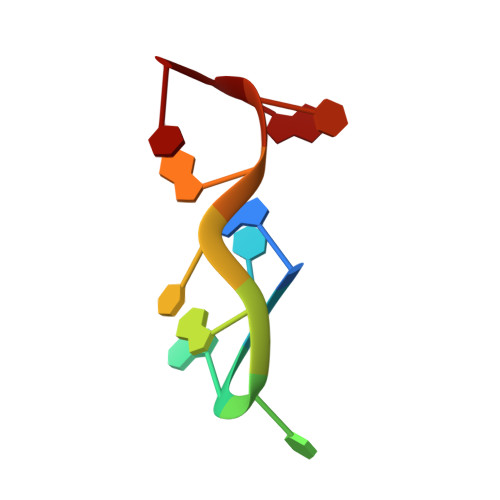
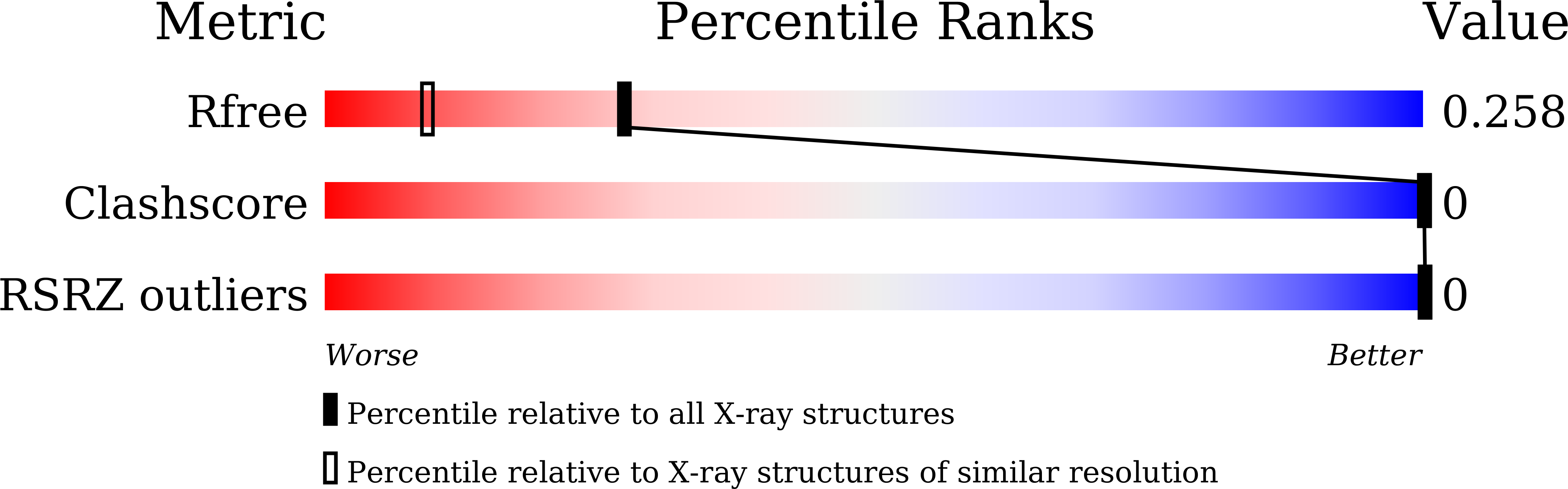d的新型双折叠结构(GCATGCATGC):一种可能的三重重复序列模型
公共医学 : 26457435 内政部: https://doi.org/10.107/S1399004715013930 相关结构的主要引文: 4ZKK号 PubMed摘要: 十脱氧核糖核苷酸d(GCATGCATGC)的结构以1.8º的分辨率呈现。 十聚体采用了一种新型的双折叠结构,其中主链的前进方向在两个胸腺嘧啶残基处发生变化。 串内堆积相互作用(包括核糖部分的内基O原子与相邻嘌呤碱之间的相互作用)、氢键和钴离子相互作用稳定了单链的双折叠结构。 两条这样的双折叠链在晶体中聚在一起形成二聚体。 品牌间Watson-Crick氢键形成四个碱基对。 十聚体结构的这一部分与之前报道的其他寡核苷酸结构中观察到的结构相似,被称为“bi-loop”。 双折叠单链结构和二聚双环结构都是构建三重重复DNA序列模型的起点,这与许多人类疾病有关。