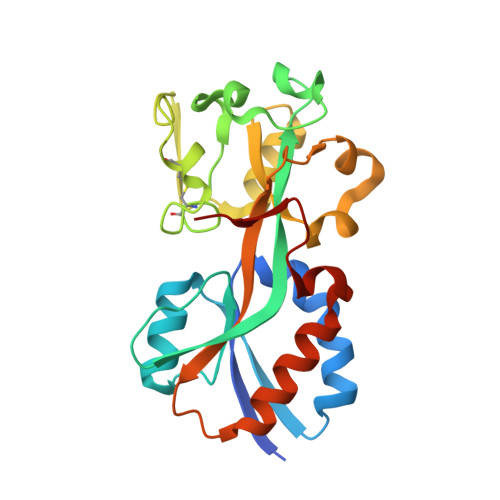
牙龈卟啉单胞菌OxyR调节域的结构解释了OxyR调控子在大肠杆菌和牙龈假单胞菌中表达的差异。
公共医学 : 24100327 内政部: https://doi.org/10.107/S090744913019471 相关结构的主要引文: 3HO7公司 , 3T22型 , 3英国国际 PubMed摘要: OxyR通过细胞氧化还原状态的可逆还原半胱氨酸二硫化物生物传感器转录调节大肠杆菌氧化应激反应基因。 这些半胱氨酸中氧化还原变化引起的结构变化通过构象传递到二聚体-亚单位界面,从而改变二聚体和四聚体与DNA的相互作用。 与大肠杆菌OxyR调节域结构相反,牙龈卟啉单胞菌OxyR调控域的晶体结构在半胱氨酸二硫键氧化还原状态的变化中二聚体构型的差异最小。 牙龈假单胞菌OxyR调节域二聚体的这种锁定结构与大肠杆菌OxyR调控域二聚物的氧化(激活)形式极为相似。 它与在牙龈卟啉单胞菌中观察到的一些氧化应激基因的组成型激活相关,并可归因于牙龈卟啉单胞菌OxyR中相对于大肠杆菌OxyR的单一氨基酸插入。 对全长牙龈卟啉单胞菌、大肠杆菌和脑膜炎奈瑟菌OxyR DNA复合物的建模预测了每种复合物的还原和氧化形式的不同DNA结合模式。