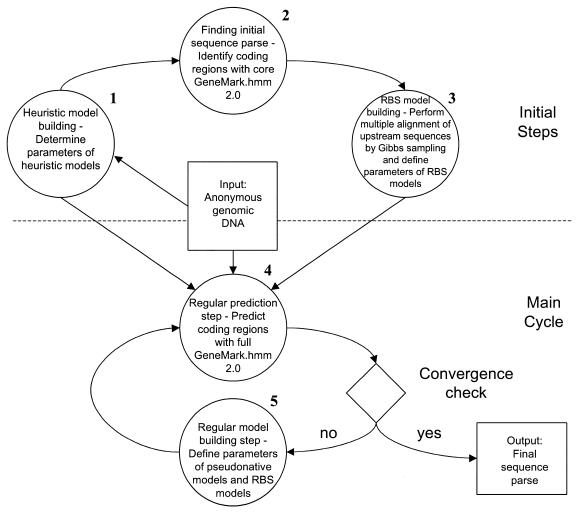
与其他项目的比较 比较GeneMarkS与Glimmer和ORPHEUS的性能 我们不得不下载并运行这两个程序。 Glimmer 2.02是 按照分发文件中的说明运行。 注释 就基因检测而言,Glimmer 2.02运行 使用默认参数检测到的基因明显多于注释的基因 两者都适用 枯草杆菌 和 大肠杆菌 。 虽然其中一些预测可能检测到未注释的真实基因 在GenBank中,假设百分比 GenBank中遗漏的真实基因数量如此之大。然而,我们没有 将Glimmer的默认参数更改为 设计决策( 7 ). 得到 Glimmer 2.02使用无核糖体结合的精确基因预测 能量计算。 此功能需要16S rRNA的事先数据 给定物种的序列。 提供16S rRNA序列 每个物种。 核糖体结合自由能的选择 在Glimmer文档中评论为“不完全 测试。” 然而,我们观察到Glimmer结果总是 启用此功能时得到改进; 因此,这个选项是 打开。
在运行ORPHEUS之前,非冗余蛋白质数据库是 通过合并更新的SWISS-PROT、TrEMBL和PIR数据库创建 借助NRDB2软件(W.Gish,未出版材料)。 这个 ORPHEUS的默认参数设置阻止查找<105的基因 nt。然而,在 大肠杆菌 测试 集合以及在短集合中 枯草杆菌 基因: 最短为114nt长。
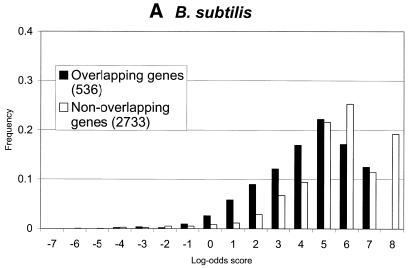
就整体而言 枯草杆菌 基因组闪光2.02 检测到98.1%的注释基因,而GeneMarkS 检测到96.7%,ORPHEUS检测到85%(表 4 ). 反过来,GeneMarkS准确地发现了83.2%的 ORPHEUS的基因精确预测了73.9%的 基因和Glimmer占62.4%。 请注意,GeneMarkS位于 中间步骤(步骤4.1,图。 1 ) 得出了与Glimmer几乎相同的结果 检测到的基因数量(表 三 ). 在这里 步骤GeneMark.hmm 2.0使用启发式马尔可夫模型并检测 98.1%的注释 枯草杆菌 基因。 尽管如此,GeneMark.hmm 2.0在这一步做出了4316个基因预测 Glimmer 2.02做出的5075个预测。
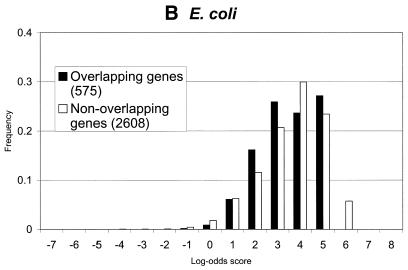
195台经实验验证 大肠杆菌 基因, GeneMarkS和Glimmer 2.02都检测到了所有的基因(表 4 ). GeneMarkS准确鉴定了94.4%的 基因启动,而ORPHEUS和Glimmer 2.02精确预测 基因起始位置分别为75.9%和71.3%。
对于短集 枯草杆菌 基因(as 如表所示 4 )预测人 GeneMarkS似乎比Glimmer的更准确。
就基因检测而言,我们又进行了一次 利用基因组进行比较测试 大肠杆菌 和 枯草杆菌 .我们已经指出了过渡 从对大量基因的不太精确的预测到 随着GeneMarkS迭代,对较少数量的基因进行更精确的预测 进度。 这种变化是由于 从更敏感而不太具体的启发式进行训练的过程 模型更具体,敏感性略低 模型。 本质上,这种转换给了GeneMarkS更多的功能 在失去某些能力的情况下找到典型班级的基因 寻找非典型基因。
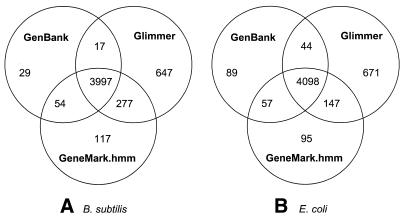
为了结合启发式模型和伪原生模型的强度, 我们在另一次运行GeneMark.hmm时同时使用了这些模型 2.0如上所述(见表 2 ). 这个 通过此程序设置获得的结果 枯草杆菌 和 大肠杆菌 基因组也如图所示 4 其中比较了GeneMark.hmm 2.0预测, 在基因检测方面,用GenBank注释这两个细菌基因组 以及Glimmer 2.02的预测。
图4。
显示组的维恩图 GenBank注释与检测到的基因集之间的关系 由GeneMark.hmm 2.0和Glimmer 2.02为 枯草杆菌 基因组 (A) 和 大肠杆菌 基因组(B)。
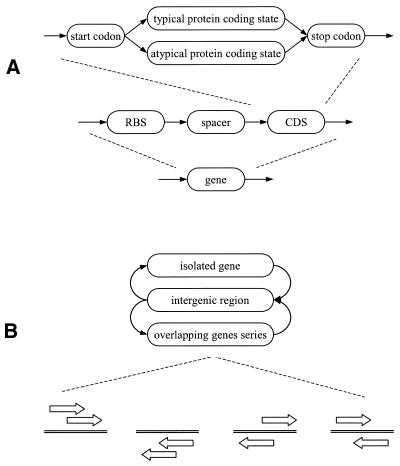
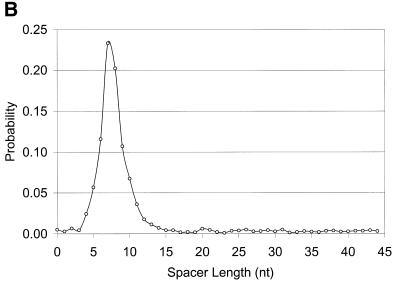
基序的功能和进化变异性 在上游序列中 GeneMarkS为研究序列模式提供了新的机会 位于基因启动上游的区域。 在许多原核生物中 基因组,上游序列携带一个功能位点(即 RBS位点)具有两个核苷酸频率基序(图。 三 A) 以及垫片长度分布(图。 三 B) 。
有趣的是,GeneMarkS导出了上游信号的模型 可能因研究中的基因组和环境而异 程序参数的。 如下所示,上游序列集 可能是不均匀的。 通常,如果 上游序列集有助于 序列基序,通过Gibbs采样的未映射多重比对 不太适合派生模式。 然而,可能有 是一些有趣的模式配置,其中方法仍然 作品。 下面我们分别讨论分析的细节 细菌种类和两类古细菌 和克里纳恰奥塔。
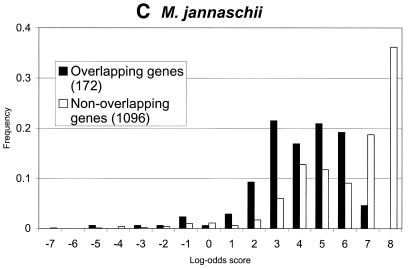
在所研究的细菌基因组中,25或 50 nt长的上游序列导致提取 RBS图案,类似于为 枯草杆菌 基因组 (图。 三 ). 然而,仍有几个 担忧。 一种是RBS序列在 一个基因组。 为了探索这种可能性,我们进行了额外的 对几个数据集的分析。 我们选择了一组基因启动子 被同一条链上的前一个基因重叠。 请注意 4nt的链基因重叠是最常见的基因重叠。 我们扫描并表征了上游序列中的六聚体 根据RBS主题和背景得出的log-odds分数 模型。 背景模型是编码的普通马尔可夫模型 基因重叠情况下的区域。 对于非重叠启动 这是一个非编码序列的普通马尔可夫模型。 通过识别 假定苏格兰皇家银行在50 nt上游地区内得分最高, 我们对 枯草杆菌 , 大肠杆菌 和 M.jannaschii先生 基因组。 RBS站点的分数分布与前一项重叠 编码区与非重叠区相比没有显著差异 (图。 5 ). 间隔棒长度分布 重叠RBS的确有三个周期(未显示数据), 整个间隔棒长度分布完全缺乏 (图。 三 B) 。
图5。
log-odds的分布 GeneMarkS检测到的RBS站点的分数,以重叠的形式显示 和非重叠基因( A类 ) 枯草杆菌, ( B类 ) 大肠杆菌 和( C类 ) M.jannaschii先生 可以看出, 重叠的基因可能位于操纵子内部, 经常拥有强大的RBS站点。 尽管如此,核糖体的大多数强位点 结合先于非重叠基因(独立基因和 引导操纵子的基因)。 这种趋势在 古菌基因组病例 M.jannaschii先生 比 在中 大肠杆菌 和 枯草杆菌 基因组。
作为此分析的扩展,我们导出了集合的RBS基序 与重叠基因启动子相关的上游序列。 在几个 这些基序与衍生基序之间的显著差异 观察到非重叠启动。 有趣的是,对于 结核分枝杆菌 基因组RBS基序 前一个基因与4nt重叠的基因更加明显 而非重叠基因衍生的RBS基序。 请注意 G+C丰富度 结核分枝杆菌 基因组 使RBS模式难以检测( 30 , 39 )。
古生物基因组的GeneMarkS分析结果如下 符合转录和翻译的概念 古生菌的机械是真核生物和细菌的复杂混合物 特性( 44 ). 转录 古生菌的形成机制与真核生物有很多相似之处。 尤其, 基本起始因子TFIIB和TFIID以及八个“小”亚基 RNA聚合酶显示出与真核生物对应物的同源性。 此外,真核生物TATA-box结合蛋白被证明具有 几种古生物中的同源蛋白质。 另一方面, 细菌和古生菌的翻译起始机制 长期以来一直被认为是功能上的不同组件 类似,以细菌型多顺反子的加工为中心 信使核糖核酸( 45 ). 两者之间的重要区别 翻译起始的古代和真核机制 真核生物中是否缺少蛋白质的古代同源物 mRNA CAP识别。
GeneMarkS应用于几个古生物基因组 提取TATA盒或RBS型图案作为模型 位于上游序列的保守位置。 这个特别的 结果显然与第一个 操纵子和与操纵子内部基因相关的分离基因。 RBS和启动子位点在比对过程中的竞争 可以通过简单限制上游长度来排除 序列。 特别是,长度必须至少为50 nt to 检测TATA盒相关模式。 对于25 nt长的上游序列 非重叠基因的多重比对过程通常会产生 RBS类型模式。 许多古生物都观察到了这一结果 物种,但不是所有物种。
在克里特岛,如 A.pernix公司 和 嗜气芽孢杆菌 我们发现了 TATA盒是为 非重叠基因的上游序列。 这一结果得到了证实 实验观察到的对无领导抄本的强烈偏见 在里面 嗜气芽孢杆菌 (M.Slupska、A.King、S.Fitz-Gibon、, J.Besemer,M.Borodovsky,J.Miller,出版中)。 另一方面, 重叠基因样本的上游序列集, 据推测,操纵子中的那些内部分子表现出了这个主题 与16S rRNA的一部分互补。请注意,对于重叠 的基因 A.pernix公司 ,GeneMarkS预测几乎相等 启动密码子ATG和GTG的频率,一个有趣的偏差不是 在任何其他物种中观察到。 另一种Crenarchaeote的基因组序列, 硫矿硫化叶菌 ,之前进行了分析 并检测到上游序列中发现的基序的二重性 翻译启动的两种不同机制的存在 ( 46 )。
类似地,在嗜热亚麻子的上游序列中 富氏A.fulgidus GeneMarkS检测到这两种转录 和翻译起始相关的主题(图 6 和 7 ). 尤其是占主导地位的保守派 50 nt长上游序列中的基序似乎不是RBS 由于其与16S rRNA和 位置周围的定位–30(图。 8 )。 这个主题有一个共识[G/a,G/a, A、 A,A,A],可以被解释为真核生物类型 启动子基序。 这一结果与获得的结果形成了鲜明对比 用于嗜热性欧亚茶素 M.jannaschii先生 谁的 根据GeneMarkS的测定,上游基序对 定位在距离基因起点更短的地方(图。 8 )并被公认为RBS图案 与16S rRNA序列互补。
图6。
序列标志代表 GeneMarkS检测到的上游序列基序 富列杜斯。 这个 一致序列相当表明存在真核样启动子 元素,而不是原核生物中常见的RBS信号。 地点 与此模式匹配的在 富氏A.fulgidus , 尽管对上游序列子集的进一步分析揭示了 第二个图案(见图。 7 )互补的 至 富氏A.fulgidus 16S rRNA。
图7。
序列标志代表 在该基因上游序列子集中观察到的RBS基序 富氏A.fulgidus 基因组。 该子集由50个组成 nt长上游序列与3′端重叠 前一个基因。 这个主题的共识是互补的 到的一部分 富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus 16S rRNA。
图8。
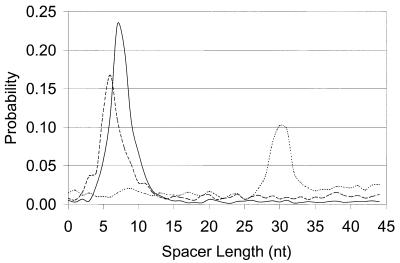
间隔棒长度分布 对于两个具有强RBS模式的物种, 枯草杆菌 和 大肠杆菌 (分别为实线和虚线), 和一个具有强真核启动子样模式的物种, 富氏A.fulgidus (虚线)。 类促进者模式 属于 富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus富足A.fulgidus 位于上游更远处 比RBS模式的启动密码子 枯草杆菌 和 大肠杆菌 。
在搜索中的RBS图案 富氏A.fulgidus 基因组序列, 我们对25 nt长的上游序列进行了吉布斯比对 重叠的基因更可能存在于操纵子内部。 这个 富氏A.fulgidus 基因组包含相对较大的 允许进一步减少集合的基因重叠数 仅使用与 他们上游的邻居正好减少了4 nt。事实上,吉布斯采样 这组上游序列的比对检测到一个带有 共识对3′端的补充 的 富氏A.fulgidus 16S rRNA(图。 7 ). 这一发现表明,无领导的成绩单 在Crenarcheota中观察到的一些广角海龙物种中也可能存在, 而含铅成绩单的比例似乎要高得多。
有趣的是,在最近的NCBI注释中,发现了欧亚海龙类物种 热等离子体火山岩 ( ftp://ncbi.nlm.nih.gov/genemos/细菌/热质_火山/ ), GeneMarkS程序使用了上游序列的默认长度 作为50 nt。我们对运行上游长度为25 nt的GeneMarkS的测试 序列长度导致了非常接近的结果,98.4%的 基因开始预测在同一位置。
在细菌基因组中,我们观察到吉布斯取样 非重叠基因的50nt上游序列比对 汇聚成带有RBS图案的路线。 这个观察结果 提示启动子相关的基序在 度比RBS基序。
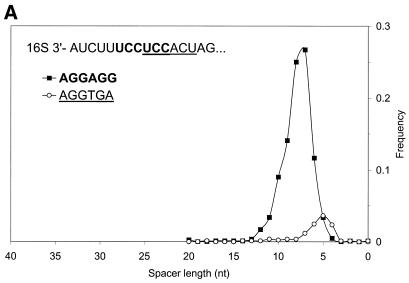
GeneMarkS在某些情况下产生的结果显示出非同质性 带有RBS基序的序列集。 我们举例说明 的结果 枯草杆菌 和 热自养M 基因组 在图中 9 A和B。如果 枯草杆菌 二 六聚体AGGAGG和AGGTGA可以叠加在Gibbs中 上游序列的采样多重比对。 两种六聚体 是对重叠部分的补充 枯草杆菌 16秒 rRNA。 六聚体对内部位置有不同的偏好 与基因启动有关的mRNA(图。 9 A) ●●●●。 顺便提一下,这些偏好使得16S rRNA的结合 将核糖体定位在一个或另一个六聚体上 与翻译起始地点的距离。 这个解释 实验突变的结果也支持了观测数据的 翻译起始效率研究( 47 )。
图9。
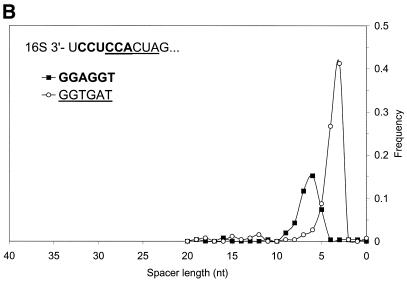
( A类 )分发 在 枯草杆菌 基因组 针对两种不同类型的可能的RBS六聚体:AGGAGG和AGGTGA。 多重排列允许这些六聚体叠加。 在 实际上游序列中,这些六聚体往往占据不同的位置 相对于起始密码子的位置。 可能涉及此偏好 翻译起始时核糖体的精确定位 16S rRNA与mRNA结合的位点 平均在距离基因起点较远的地方观察到 而不是稀有的六聚体。 ( B类 )间隔棒分布 观察到的长度 热自养M 基因组 用于两种不同类型的RBS六聚体:GGAGGT和GGTGAT。 属性 其中的六聚体与在 枯草杆菌 基因组(A),除了更频繁的 现在发现六聚体与基因的平均距离更近 开始比罕见的六聚体。
注意,将上游序列集拆分为两个(或更多) 同质子集暗示了一个事实,即整个基因集 一个特定物种可以分为两个(或更多)同质物种 类,即典型和非典型基因类。 还有,更进一步 分析没有提供任何明显的证据表明 六聚体与基因类型相关(数据未显示)。
对于古生物基因组 热自养M GeneMarkS 分析导致了对集合非均匀性的类似观察 如图所示 9 B.两个 六聚体,GGAGGT和GGTGAT,可以叠加在Gibbs中 采样多重对齐。 这两种六聚体都是对 的重叠部分 热自养M 16秒 rRNA。 有趣的是,与 枯草杆菌 案例, GGTGAT六聚体通常位于较短的距离 从基因开始。