背景 雌激素参与许多脊椎动物的发育和生理过程。 人类和动物研究揭示了雌激素受体(ER)在男女性发育和行为、生殖功能、神经内分泌和心血管系统的调节以及骨代谢中的作用[ 1 ]. 乳腺肿瘤的分子特征和流行病学研究也表明雌激素和雌激素受体在乳腺癌的发生、发展和治疗中具有重要作用[ 2 , 三 ].
已知两种ER亚型,ERα和ERβ,可介导雌激素信号; 它们作为配体依赖的转录因子发挥作用[ 4 ]. 雌激素穿过细胞膜后,与受体结合,导致受体活化。 ER与 顺式 -靶基因的调节元件直接与先前描述的保守雌激素反应元件(ERE;5'-GGTCANNNTGACC-3',其中N是任何核苷酸)结合,或间接与AP-1和Sp1转录因子复合物及其各自的结合位点结合[ 5 - 9 ]. 辅激活物和辅阻遏物与雌激素受体形成复合物并参与调节雌激素反应[ 10 ]. 靶基因调控元件的ER和转录复合物的周期性周转也提供了一种额外的调控机制[ 11 - 13 ]. 共调节因子、相关转录因子复合物、受体亚型和剪接变异体的组织特异性分布是观察到的雌激素多效性作用的潜在机制[ 14 ]. 在分子水平上,ER激活的结果似乎是靶基因转录活性和表达谱的改变。 许多基因,包括三叶因子1/pS2、组织蛋白酶D、细胞周期蛋白D1、c-Myc和孕酮受体的基因,都受到ERα的正向调节[ 15 - 20 ]. 雌激素受体的转录抑制已有文献记载,但尚未得到很好的研究或理解。
已经进行了微阵列实验,特别是在乳腺癌细胞系中,以研究雌激素治疗后基因表达谱的改变[ 21 - 27 ]. 然而,利用这些反应基因的初始清单,包括跨细胞系反应的整体保存,许多关键问题仍有待解决, 体内 与乳腺肿瘤的相关性,以及 顺式 -调节元件定位、分子特征和ER与假定靶基因之间相互作用的确认。 在本研究中,我们采用组合方法,通过使用高密度DNA微阵列来发现和表征ERα靶基因,以获得ERα阳性(EPα + )乳腺肿瘤细胞。 这包括检查ER-介导和翻译依赖性调节的药物治疗,整合额外的 在体外 雌激素应答数据和人类乳腺肿瘤样本基因表达数据用于候选基因验证和相关鉴定 体内 靶点、计算结合位点建模和启动子分析以绘制推测的ER结合位点,染色质免疫沉淀(ChIP)以表征ER和候选靶基因调控元件之间的相互作用。 在这里,我们展示了我们的发现,并讨论了它们为ER介导的转录调控网络的全基因组结构及其在细胞系、乳腺肿瘤和进化中的保护提供的见解。
结果 雌激素反应的全球基因表达谱 高密度DNA微阵列是一种强大的工具,可以同时确定数千个基因的转录谱,特别适合于转录因子功能的研究。 以前的研究确定MCF-7激素治疗后基因表达谱的变化[ 21 - 25 , 27 ]和ZR-75-1[ 26 ]急诊室 + 乳腺癌细胞株产生了许多新的雌激素反应基因,并证明了这种基因组技术在研究雌激素生物学中的实用性。 然而,这些早期的研究只包括在有限的时间点进行抗雌激素和环己酰亚胺(CHX)治疗,或仅在少数假定应答基因的验证分析中进行。 因此,为了更全面地绘制由ER调节的转录调控网络,并在额外的ER中生成数据 + 乳腺肿瘤细胞背景下的跨细胞系分析,我们治疗雌激素依赖性T-47D ER + 含有17β-雌二醇(E2)和E2与纯抗雌激素ICI 182780(ICI)或蛋白合成抑制剂CHX联合的乳腺癌细胞系,并进行了高分辨率的时间进程基因表达分析(见图 第1页 治疗和时间点)使用斑点寡核苷酸(60-mers)微阵列,其中包含约19000个人类基因的探针。 本研究中使用的E2(1 nM)和ICI(10 nM)浓度分别足以驱动和抑制激素反应性细胞增殖。 T-47D细胞在核型异常和核受体共调节因子表达水平上与以前使用的细胞系MCF-7和ZR-75-1不同,但具有在更生理水平上表达ER的优势[ 28 , 29 ]. 在激素治疗后的前8小时(0-8小时),每小时采集一次样本,在接下来的16小时(10-24小时),每两小时采集一次样本,总共调查了16个时间点(图 第1页 ).
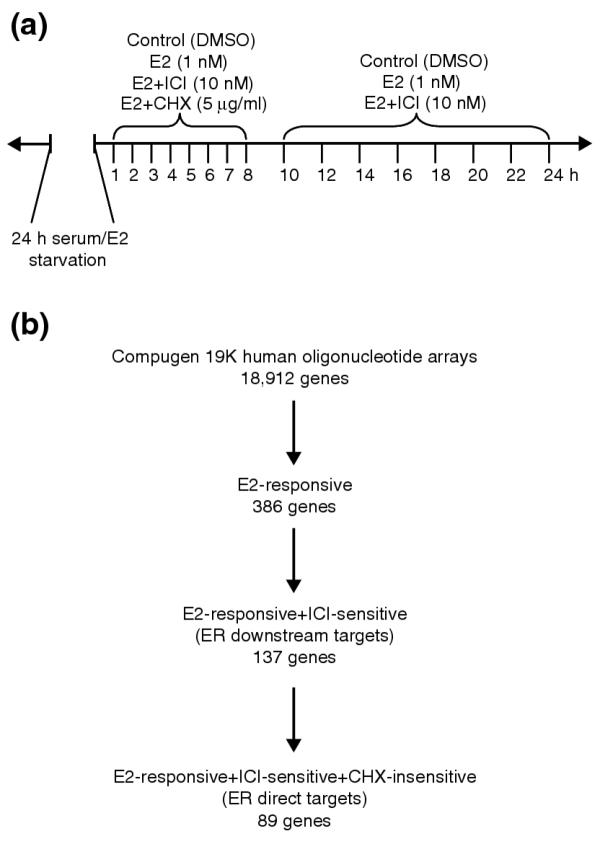
图1。
T-47D细胞雌激素反应时程分析的实验设计和微阵列数据选择。 (a) 在用二甲亚砜(DMSO;载体对照)、17β-雌二醇(E2)和E2与ICI 182780(ICI)和环己酰亚胺(CHX)联合治疗之前,细胞需要24小时的血清和雌激素。 在指定的16个时间点采集样本。 (b) 确定直接ER目标的程序。 雌激素反应基因的数据选择基于 第页 -价值截止点( 第页 ≤0.052)和响应幅度(三个时间点在同一方向上至少变化1.2倍)。 ICI敏感性和CHX不敏感性也通过统计方法测定, 第页 ≤0.058和 第页 分别≥0.24。 截止值由组织蛋白酶D和黄体酮受体的表达谱决定,这两种受体都已知受ER调节。
通过使用双尾配对对E2处理的样本与模拟处理的对照组中的表达比率进行统计分析,确定雌激素应答基因 t吨 -使用测试 第页 -价值截止。 此外,在三个或更多时间点内,基因在同一方向上的变化至少为1.2倍。 我们选择数据选择标准的依据是观察到的已知激素应答和ER靶基因的表达水平和特征,如孕酮受体、组织蛋白酶D和锡酸钙2。 E2反应性选择反应基因( 第页 <0.052)并过滤ICI灵敏度( 第页 <0.057)和CHX不敏感( 第页 >0.24)通过比较E2处理样品与E2+ICI和E2+CHX样品,分别分离出推测的ER下游靶点和直接靶点。 图 1亿 总结了选择过程的统计信息。 通过Eisen聚类图显示雌激素反应基因的表达谱,并通过层次聚类对基因进行排序[ 30 ].
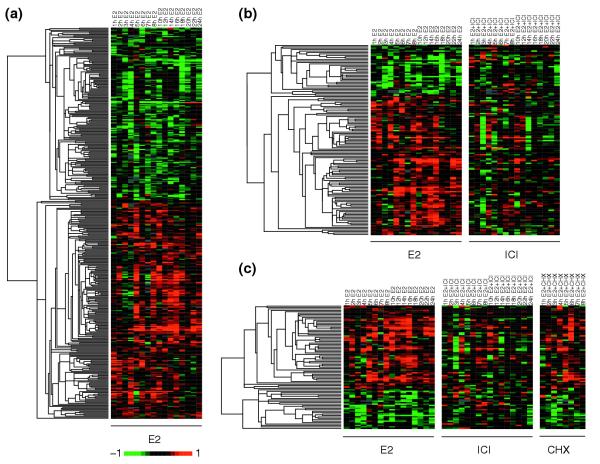
雌激素反应基因(图 2a个 ),表达谱分为两组:激素治疗上调的基因(图 2a个 ,红色)和那些被下调的(图 2a个 ,绿色)。 在应答基因中,58.5%(226/386)在雌激素治疗后上调。 图 第2页 显示了由雌激素受体(ER)特异性调节的137个基因的表达谱(定义为对雌激素有反应并被ICI治疗阻断,图 第2页 ,右侧面板)。 这些基因以与雌激素反应基因相似的方式聚集。 一项值得注意的发现是,由E2反应和ICI敏感性决定的ER调节基因仅占所有雌激素反应基因的35.5%(137/386),这表明在介导激素暴露的转录反应中可能存在ER依赖性信号转导机制。 有趣的是,ICI对E2-上调基因的影响似乎大于下调基因,因为这些上调基因占ICI敏感亚群的71.5%(98/137)。 89个主要反应基因构成了推测的ER直接靶点,定义为对激素治疗有反应,对ICI敏感,但不受CHX治疗的影响(图 2厘米 ,右侧面板)。 从这些观察结果来看,参与乳腺癌细胞启动激素反应的直接靶基因数量可能仅占人类基因组中基因的0.47%(89/18912)。 表中列出了推测的ER靶基因 1 E2应答基因的完整列表见附加数据文件4。 先前显示为ER靶基因的基因以粗体显示(见表 1 ). 我们注意到,我们列表中的五个直接靶基因与唯一一项已发表的微阵列研究中的结果一致,该研究将抗雌激素和CHX治疗作为实验设计的一部分[ 26 ]. 这两个数据集之间的差异可能是由于细胞系、实验设计、阵列平台和用于选择靶基因的过滤参数的差异。 例如,Soulez和Parker的研究[ 26 ]使用仅在两个时间点(6小时和24小时)测试的ZR-75-1细胞。 此外,该研究中使用的Affymetrix HuGeneFL阵列包含5600个基因的探针,而我们的阵列上有近19000个基因,代表了具有已知局限性的早期阵列技术。 同样,我们的直接靶基因列表中缺少已知的靶基因TFF1/pS2和细胞周期蛋白D1可能是由于T-47D细胞中转录辅因子和基因组异常的差异。 c的缺失- MYC公司 我们名单上的原癌基因可能是因为我们研究中使用的阵列中没有探针。
图2。
雌激素反应基因的表达谱。 (a) 通过层次聚类和Eisen TreeView软件对386个E2反应基因进行可视化。 列表示按时间顺序排列的时间点,每行表示特定基因的表达谱。 按照惯例,上调用红色信号表示,下调用绿色信号表示。 变化的幅度与信号的亮度成正比。 每个面板代表列标题中所述的治疗条件。 (b) ICI和 (c) CHX处理分别鉴定137个ER调节基因和89个主要反应基因。
表1。
加入号码 符号 基因名称 时间点数量* 基因本体论
(a) 推测ER靶基因上调(89个中的59个)
AL049265号 mRNA; cDNA DKFZp564F053 14 生物工艺未知[00000004]
NM_003246号 泰铢1 凝血酶敏感蛋白1 13 细胞粘附[0007155]
NM_016339 链接-GEFII 连接鸟嘌呤核苷酸交换因子II 13 神经发生[0007399]
U79299型 OLFM1型 Olfactomedin 1 13 信号转导[0007165]
AK026062号 DNAJC1公司 DnaJ(Hsp40)同系物,C亚家族,成员1 12 蛋白质折叠[0006457]
M62403型
IGFBP4型 †
胰岛素样生长因子结合蛋白4 12 信号转导[0007165]
NM_001089号
澳大利亚广播公司3 †
ATP-装订盒,子家族A(ABC1),成员3 12 ATP-装订盒(ABC)运输机[0004009]
NM_003714号 STC2系统 锡酸钙2 12 细胞间信号[0007267]
AF075060型 假想蛋白DKFZp761P0423 11 生物工艺未知[00000004]
非洲271070 SLC38A1型 溶质载体系列38,成员1 11 氨基酸转运[0006865]
AK024639公司 假设蛋白FLJ20986 11 阳离子运输[0006812]
AL080199号 mRNA; cDNA DKFZp434E082 11 生物工艺未知[00000004]
NM_014365号
H(H) 11
蛋白激酶H11 11 转化监管,启动[0006446]
AF245389型
绿色1 †‡
GREB1蛋白 10 高亲和力锌离子转运[0006830]
NM_002894号
RBBP8型 †
视网膜母细胞瘤结合蛋白8 10 DNA修复[0006281]
NM_005067号 SIAH2号机组 缺席7名同系物2( 果蝇属 ) 10 泛素依赖性蛋白质降解[0006511]
16752美元
CXCL12系列 †
趋化因子(C-X-C基序)配体12(基质细胞衍生因子1) 10 免疫应答[0006955]
AF086500型 FZD8型 卷发同源物8( 果蝇属 ) 9 生物工艺未知[00000004]
2012年6月17日 JDP1 含J结构域的蛋白质1 9 生理过程[0007582]
AF182416型 NIF3L1 NIF3 NGG1相互作用因子3样1(S.pombe) 9 DNA甲基化[0006306]
AK023772号 假设蛋白质FLJ13710 9 开发过程[0007275]
AK024361号 假设蛋白质FLJ14299 9 转录调控[0006355]
AK025812 cDNA:FLJ22159 fis,克隆HRC00251,mRNA序列 9 生物工艺未知[00000004]
纳米_001037 SCN1B型 钠通道,电压门控,I型,β多肽 9 钠运输[0006814]
NM_003287号 TPD52L1型 肿瘤蛋白D52样1 9 信号传感器[0004871]
NM_003646号 DGKZ公司 二酰甘油激酶,zeta 104 kDa 9 信号转导[0007165]
NM_014333号 IGSF4型 免疫球蛋白超家族,成员4 9 毒力[0009406]
NM_016300 ARPP-21协议 环腺苷酸调节的磷酸蛋白,21 kD 9 生物工艺未知[00000004]
AF200341型 HPYR1型
幽门螺杆菌 响应1
8 生物工艺未知[00000004]
AK023199号 cDNA FLJ13137 fis,克隆NT2RP3003150,mRNA序列 8 生物工艺未知[00000004]
AK025571 假设蛋白质FLJ21918 8 RNA处理[0006396]
D00265号 中国青年科学院 细胞色素c,体细胞 8 电子传输[0006118]
NM_000926号
PGR公司 †
孕酮受体 8 信号转导[0007165]
NM_001634号 AMD1公司 S-腺苷蛋氨酸脱羧酶1 8 多胺生物合成[0006596]
NM_002184号
il6吨 ‡
白细胞介素6信号转导子(gp130,抑癌素M受体) 8 信号转导[0007165]
NM_003489
自然保护区1 †
核受体相互作用蛋白1 8 转录调控[0006355]
NM_004878号 PTGES公司 前列腺素E合成酶 8 前列腺素代谢[0006693]
NM_012111 C14或3 14号染色体开放阅读框3 8 蛋白质折叠[0006457]
NM_015878号 OAZIN公司 鸟氨酸脱羧酶抗酶抑制剂 8 多胺生物合成[0006596]
AK023680 PPP1R15B型 蛋白磷酸酶1,调节(抑制剂)亚单位15B 7 生物工艺未知[00000004]
NM_001909号
CTSD公司 †
组织蛋白酶D(溶酶体天冬氨酰蛋白酶) 7 蛋白质分解和肽解[0006508]
NM_003774号 GALNT4公司 UDP-N-乙酰-α-D-半乳糖胺:多肽N-乙酰半乳糖氨基转移酶4 7 生物工艺未知[00000004]
NM_014810 CAP350型 中心体相关蛋白350 7 非选择性囊泡转运[0006899]
NM_016391 热休克蛋白111 假设蛋白HSPC111 7 主绞线伸长[0006272]
AK021773号 cDNA FLJ11711 fis,克隆HEMBA1005152,mRNA序列 6 生物工艺未知[00000004]
AK025766公司 BRI3BP公司 BRI3结合蛋白 6 生物工艺未知[00000004]
L23401型 人类重复区mRNA 6 生物工艺未知[00000004]
NM_000427号 LOR公司 洛里克林 6 单元格形状和单元格大小控制[0007148]
纳米_001932 MPP3(MPP3) 膜蛋白,棕榈酰化3(MAGUK p55亚家族成员3) 6 信号转导[0007165]
NM_002227 日本1号机组 Janus激酶1(一种蛋白酪氨酸激酶) 6 蛋白质磷酸化[0006468]
NM_006392号 NOL5A(无5A) 核蛋白5A(56 kDa,含KKE/D重复序列) 6 转录[0006350]
纳米_006796 AFG3L2型 AFG3 ATP酶家族基因3-样2(酵母) 6 生物工艺未知[00000004]
NM_013324 CISH公司 细胞因子诱导的含SH2蛋白 6 JAK-STAT级联[0007259]
NM_016233号 PADI3型 肽基精氨酸脱氨酶,III型 6 蛋白质修饰[0006464]
NM_020120号 UGCGL1公司 UDP-葡萄糖神经酰胺葡萄糖基转移酶样1 6 蛋白质修饰[0006464]
AB037842号
KIAA1421号机组 ‡
KIAA1421蛋白 5 RNA依赖的DNA复制[0006278]
NM_001116号 ADCY9公司 腺苷酸环化酶9 5 信号转导[0007165]
NM_014121 PRO0233蛋白质 5 双品牌断口修复[0006303]
纳米_018053 假想蛋白FLJ10307 5 细胞死亡[0008219]
(b) 推测ER靶基因下调(89个中的30个)
NM_012342号
国家气象局 †
推测跨膜蛋白 13 酪氨酸合成黑色素[0006583]
AF039944型 国家发改委1 N-myc下游调控基因1 11 生物工艺未知[00000004]
AL049471号 mRNA; cDNA DKFZp586N012 11 生物工艺未知[00000004]
AK024964号 美国国家足球协会 核因子I/A 9 DNA复制[0006260]
M16006型 SERPINE1系列 丝氨酸(或半胱氨酸)蛋白酶抑制剂,E族,成员1 9 急性相反应[0006953]
NM_004438号 EPHA4型 EphA4 9 信号转导[0007165]
NM_006449 CDC42EP3系统 CDC42效应蛋白(Rho-GTPase结合)3 9 信号转导[0007165]
AK026298号 NMES1型 正常食管粘膜特异性1 8 生物工艺未知[00000004]
D16875号 人HepG2 3'区cDNA,克隆hmd1f06,mRNA序列 8 生物工艺未知[00000004]
NM_002237号 KCNG1公司 钾电压门控通道,G亚家族,成员1 8 钾运输[0006813]
纳米_003032 SIAT1公司 唾液酰基转移酶1(β-半乳糖苷α-2,6-唾液酰基转移酶) 8 蛋白质修饰[0006464]
NM_006605 RFPL2 食指蛋白样2 8 蛋白质结合[0005515]
AK025922号 假想蛋白FLJ22269 7 开发过程[0007275]
NM_000504号
F类 10
凝血因子X 7 蛋白质分解和肽解[0006508]
NM_001139号 ALOX12B型 花生四烯酸12-脂氧合酶,12R型 7 表皮分化【0008544】
NM_004354号 CCNG2公司 细胞周期蛋白G2 7 细胞周期检查点[0000075]
NM_006137 CD7(CD7) CD7抗原(p41) 7 体液防御机制[0006959]
NM_007273
REA公司 ‡
雌激素受体活性抑制剂 7 细胞增殖阴性对照[0008285]
NM_014583号 LMCD1型 LIM和富含半胱氨酸的结构域1 7 转录因子[0003700]
NM_017572 MKNK2型 MAP激酶相互作用丝氨酸/苏氨酸激酶2 7 蛋白质磷酸化[0006468]
第49356页 人信使核糖核酸(S100C相关基因) 6 细胞周期[0007049]
NM_001878号 CRABP2型 细胞维甲酸结合蛋白2 6 信号转导[0007165]
NM_004388号 全面禁试条约 壳二糖酶,二-N-乙酰- 6 碳水化合物代谢[0005975]
NM_006622号 SNK公司 血清诱导激酶 6 蛋白质磷酸化[0006468]
AK022072号 cDNA FLJ12010 fis,克隆HEMBB1001635,mRNA序列 5 生物工艺未知[00000004]
AL137529号 假想蛋白FLJ23751 5 脂质代谢[0006629]
NM_000430号 PAFAH1B1型 血小板活化因子乙酰水解酶,亚型Ib,α亚单位45 kDa 5 信号转导[0007165]
NM_013332号 高强度气体2 低氧诱导蛋白2 5 生物工艺未知[00000004]
NM_014770 CENTG1标准 半人马座,伽马1 5 细胞生长和/或维持[0008151]
NM_001719
骨形态发生蛋白7 †
骨形态发生蛋白7(成骨蛋白1) 4 细胞生长和/或维持[0008151]
为了控制ICI和CHX的混杂直接作用,我们也在不依赖E2的情况下,仅用ICI处理细胞2、8、12或24小时,或仅用CHX处理细胞2或8小时。 CHX治疗通过诱导CHX、E2或E2+CHX治疗后可检测到的E2样效应,部分掩盖了89个符合假定直接靶基因选择标准的基因中4个(4.5%)的反应(见附加数据文件5和6)。 由于这个结果不排除这些基因作为直接靶点,我们将它们列入直接靶点列表,但在表中注意到了这一警告 1 在32个E2应答基因中,CHX单独处理与E2处理的效果相反(8.3%;32/386)。 然而,在激素和药物同时存在(E2+CHX)的情况下,CHX不会拮抗这些基因的E2反应,因此不会影响假定直接靶点的选择。 仅ICI处理就在九个(2.3%;9/386)E2应答基因中引发了意料之外的E2样反应(即E2、ICI或E2+ICI处理后的相同反应)。 由于这9个基因不符合ICI拮抗的选择标准,因此推测的直接靶基因也没有受到影响。 因此,ICI和CHX的独立效应并没有实质性改变我们分析中的最终基因列表或结论。
T-47D和MCF-7雌激素反应谱的比较 许多乳腺癌细胞系已用于 在体外 雌激素反应的研究,但大多数数据,包括已发表的微阵列研究,都来自使用MCF-7细胞的实验。 因此,我们有兴趣比较我们研究中使用的MCF-7细胞和T-47D细胞对激素治疗反应的表达谱。 为了进行分析,我们获得了一个公开的MCF-7激素和三苯氧胺反应数据集[ 31 ]来自斯坦福微阵列数据库[ 32 ].
使用构建166中的Unigene簇ID作为两个数据集之间的通用标识符,我们从137个T-47D ER调节基因中的104个中提取了表达数据(图 3a年 )也存在于MCF-7数据集中。 对于MCF-7数据中具有多个条目的基因,选择具有最完整数据或与T-47D结果类似表达谱的条目进行分析。 总的来说,MCF-7实验的结果对应于T-47D细胞中获得的响应基因的大多数(64%;66/103)表达谱,如激素治疗后MCF-7细胞中可用数据点的相同方向变化所定义(见图 3a年 ). 使用MCF-7 E2反应和三苯氧胺治疗敏感性数据的严格选择标准(见材料和方法),我们在MCF-7和T-47D数据集中发现了24个可被定义为ER调节的基因。 值得注意的是,这两项研究之间高度一致,24个基因中有23个(96%)的基因对雌激素的表达反应一致(图 第3页 ). 相反,尽管使用了相似的实验系统和相同的阵列平台,但来自无关干细胞研究的微阵列数据集几乎没有一致性[ 33 ]. 这项研究中确定的基因与Frasor及其同事报告的雌激素反应基因之间的许多重叠进一步证实了这些发现(数据未显示)[ 23 ]. 我们使用不同的实验设计和阵列平台观察到的两个ER研究之间的相似性表明,这两个ER + 细胞系具有共同的雌激素反应途径。
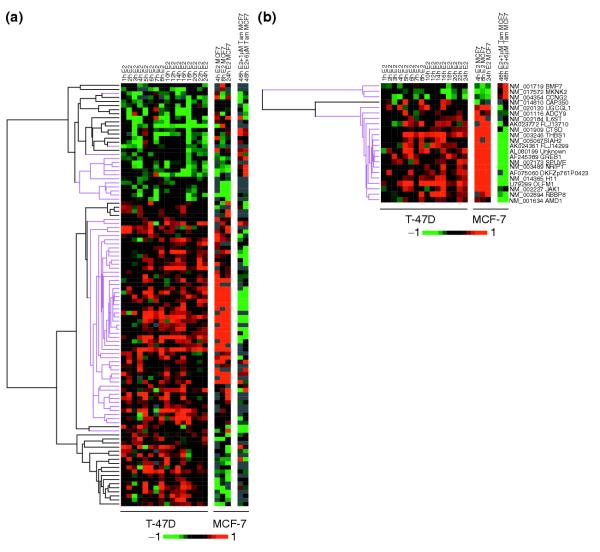
图3。
对T-47D和MCF-7乳腺癌细胞系雌激素反应基因表达谱的比较分析显示出类似的反应。 (a) T47-D细胞中103个对E2敏感且对ICI治疗敏感的基因的表达数据显示,64%(66/103)的基因(以洋红突出显示)对E2的反应一致。 (b) 如果选择MCF-7数据作为E2反应和三苯氧胺(Tam)敏感基因(参见材料和方法),则两个数据集之间有24个基因重叠,E2和抗雌激素ICI或三苯氧乙烯的反应高度一致(洋红色),接近96%(23/24)。
乳腺肿瘤中ER调节基因的差异表达 我们希望解决的一个关键问题是 在体外 细胞系中的观察反映了生物学意义 体内 为了解决这个问题,我们探索了在我们的 在体外 乳腺肿瘤标本中ER状态相关表达谱的分析。 我们假设ER靶基因在乳腺肿瘤中的差异表达与ER状态相关。 例如,在MCF-7细胞中,pS2/TFF1和cyclin D1均被雌激素上调,但在ER中表达水平较高 + 肿瘤[ 34 , 35 ].
许多乳腺癌微阵列研究表明,ER状态仍然是最重要的预后标志物和肿瘤分类器。 来自六项乳腺癌微阵列研究的表达数据(L.D.M.、B.M.F.Mow、L.A.V.和E.T.L.,未发表的工作和[ 36 - 40 ])被挖掘出差异表达的基因( 第页 -值<0.01错误发现率,ER + 与ER相比 - )与ER状态相关的人类乳腺肿瘤样本。 在T-47D研究中确定的137个ER调节基因(E2反应,ICI敏感)中,至少有一项乳腺癌研究中有44个基因存在差异表达(图 4 ). 在3812个符合选择标准的ER状态相关基因中,44个ER调节基因仅占约1%(44/3811)( 第页 <0.01(在一项或多项研究中),表明雌激素反应途径仅代表乳腺肿瘤中ER状态相关转录组的一小部分。 这与Meltzer及其同事之前的观察结果类似[ 22 , 39 ]. 然而,与我们研究中使用的微阵列中这些ER调节基因的频率相比,ER状态相关基因中的ER调节基因似乎显著丰富(1.15%,44/3811 vs 0.72%,137/18912, 第页 =0.006(通过X平方分析)。
图4。
细胞系研究中确定的雌激素反应基因在ER中也有差异表达 + 乳腺肿瘤与ER的比较 - 肿瘤。 在所调查的六项乳腺肿瘤研究(L.D.M.、B.M.F.Mow、L.A.V.和E.T.L.,未发表的工作和[36-40])中,将雌激素应答基因表达谱与这些基因的复合表达率进行比较。 差异表达基因定义如下 第页 ER之间<0.01 + 和ER - 肿瘤样本。 ER中反应类似的基因 + 肿瘤和 在体外 以下E2处理以洋红色突出显示。
为了比较反应基因和差异表达基因的表达谱,我们绘制了每个基因的平均相对表达率(ER + /急诊室 - )乳腺癌研究的所有样本(图 4 ). T-47D细胞中的雌激素反应基因与乳腺肿瘤中差异表达的基因之间存在惊人的一致性(70.5%;31/44)。 例如,激素治疗上调了基因(图 4 ,左面板,红色)也在ER中过度表达 + 乳腺肿瘤(图 4 ,右侧面板,红色)。 我们注意到雌激素治疗后有一组(29.5%;13/44)基因表现出相反的反应 在体外 与肿瘤中ER状态相关表达相比。 然而,这13个在细胞系和肿瘤数据之间不一致的基因在两个细胞系(T47-D和MCF-7)中是一致的。 这表明一些下游通路的调控依赖于环境,这在原发性肿瘤和实验性细胞系之间可能是不同的。 综上所述,我们注意到 在体外 经验证的雌激素反应基因在ER中也有差异表达 + 因此可能具有直接的生物学和临床意义。
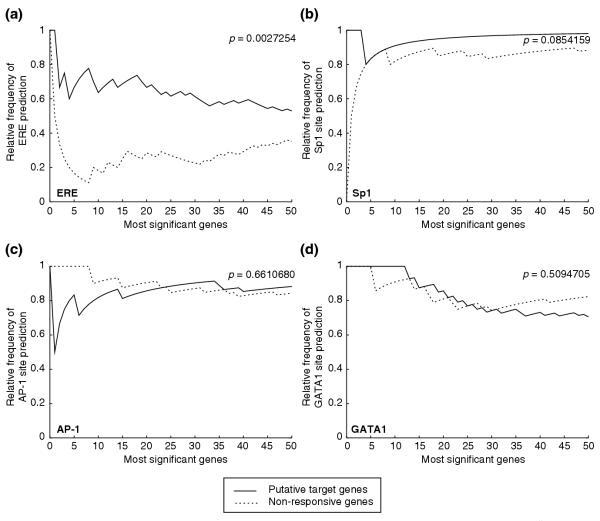
ER结合位点的计算建模和预测 先前的研究已经确定DNA中的一致ERE和AP-1-或Sp1-结合位点可能是的靶基序。 这表明89个直接反应基因应该在转录控制区内富集这些结合基序。 为了进一步探索这一点,我们通过计算提取了候选基因转录起始位点(TSS,见材料和方法部分)侧翼(-3000至+500)的序列,该位点被定义为NCBI RefSeq数据库中参考转录物的最大5'核苷酸,并查询其潜在的ER-binding位点。 之所以选择起始位点两侧序列的大小和位置,是因为大多数具有特征的ER-binding位点都已映射到已知靶基因中的这些区域[ 5 ].
对于结合位点预测,我们使用了前面描述的ERE模型[ 41 ]TRANSFAC数据库中的AP-1和Sp1结合位点位置权重矩阵[ 42 ]. 我们还将GATA1转录因子的结合位点作为阴性对照,因为它不参与ER结合。 在检测模型训练数据中已知结合位点时,所有被调查位点的模型灵敏度设置为ERE模型的83%灵敏度的既定最佳设置。 图 5 显示了ERE的性能(图 5a级 ),AP-1(图 5亿 ),Sp1(图 5厘米 )和GATA1(图 5天 )结合位点模型。 这个 年 -每个图的轴表示结合位点预测的相对频率,由具有预测结合位点的基因占查询基因总数的比例决定; 这个 x个 -轴表示每个基因组中被调查的最重要基因的数量,按统计显著性排序(参见材料和方法)。 由于短结合位点基序在人类基因组中普遍存在,我们询问与无反应基因相比,在3.5 kb的反应基因上游窗口中是否存在此类反应元件的富集。 每个基序的丰富性由假定靶基因结合位点预测相对频率的差异表示(图 三 ,实线)和非应答基因(图 三 ,支离破碎的线条)。 对于ERE预测,我们观察到10个最重要的初级反应基因中的推定位点比最无反应的对照组富集了三倍(图 3a年 )25个和50个最重要的基因分别富集了两倍和大约70%。 总的来说,ERE位点在推测的ER直接靶基因中的富集具有统计学意义( 第页 = 0.0027). 目标基因中假定的Sp1位点的富集程度较低,但未达到统计显著性(10个最重要的目标基因富集12.5%; 第页 = 0.085). 这是预期的,因为Sp1位点在人类基因组中非常常见,并且在一般转录调控中具有额外功能。 我们没有观察到AP-1位点的任何富集( 第页 =0.66)或阴性对照GATA1位点( 第页 = 0.51). 这些发现表明,ERE是在全基因组范围内调节ER靶基因特异性调节的主要反应元件。 我们还推测,虽然已知Sp1和AP-1结合位点可促进某些靶基因中的内质网功能,但它们并不是一种常见的内质网膜靶基因 顺式 -人类基因组中的调节元件,至少不足以区分目标基因和非响应基因。
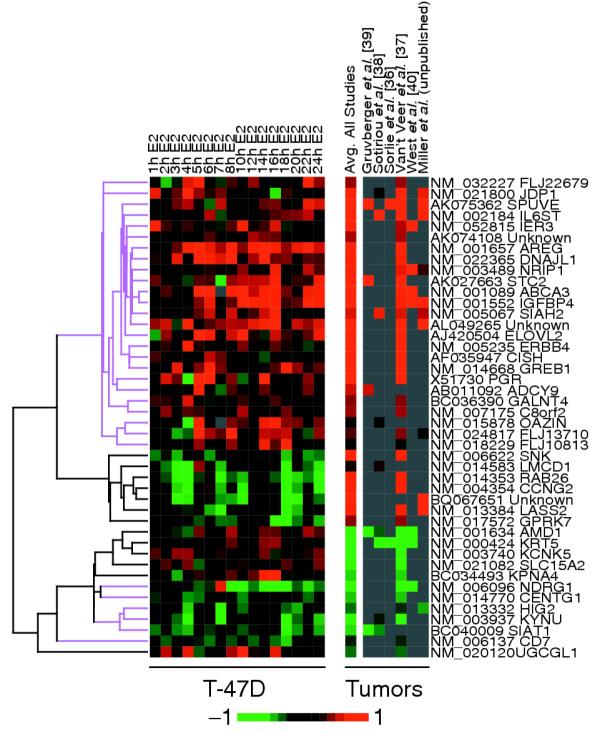
图5。
ERE样序列富集在假定靶基因的扩展启动子区域。 (a) 使用先前发布的模型和优化的灵敏度设置进行ERE预测。 根据统计显著性排序,对最重要的推测ER靶基因和非响应基因的扩展启动子区域(-3000到+500,两条链)进行预测模型测试。 这个 年 -轴表示结合位点预测的相对频率,由具有预测位点的基因数除以总基因数决定。 查询的最重要基因的数量显示在 x个 -轴。 推测靶基因中ERE预测的频率明显更高( 第页 =0.0027)。 结合位点预测也使用位置权重矩阵来描述 (b) Sp1、, (c) AP-1和 (d) 已知Sp1和AP-1都参与调节某些靶基因的ER结合。 GATA1位点被列为阴性对照。 这些位点在假定的靶基因中没有显著富集(参见 第页 -图中的值)。
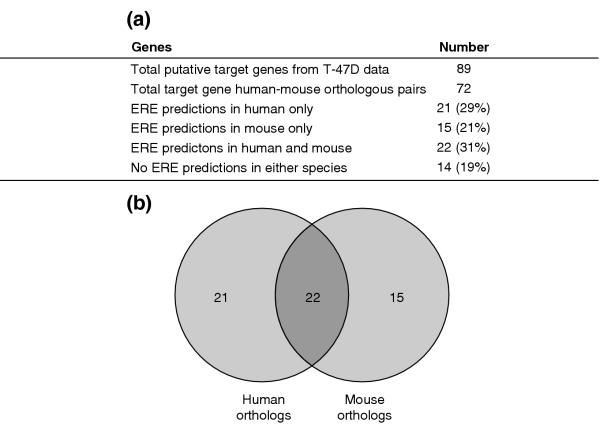
为了确定预测的ERE的保守性和潜在功能,我们还检测了89个假定的人类ER靶基因的小鼠直系同源基因5'上游区域中的相同3.5 kb窗口。 从小鼠基因组数据库中提取了72对人-鼠同源基因对[ 43 ]以及为潜在ERE划定和分析的调节区域,如人类序列所述(见材料和方法)。 然后,我们比较了这两种生物体的ERE预测,以了解以下特征:当两种生物体中预测出多个ERE时,核心ERE半位点(GGTCANNTGACC)在两个最相似的序列之间的保存,不包括侧翼嘌呤碱; 预测ERE的5'和3'端侧翼的20个碱基(总共40个碱基)的保守性; 以及结合位点序列和TSS之间的距离。
我们的分析统计总结如图所示 第6页 在鼠-人同源对中,81%(58/72)至少有一个ERE预测,22(31%;22/72)个基因对在两种生物体中都有ERE预测。 然而,在人类直接靶基因中,29%(21/72)的小鼠同源基因上游没有ERE。 相反,21%(15/72)患有ERE的小鼠基因在其人类同源基因上游没有ERE。 在这两种生物体中具有ERE预测的22个基因对中(见图中的维恩图 6b条 ),只有四个具有核心ERE序列的完美保守性(表 2 ). 这四个完全保守的ERE对在其侧翼序列中也具有最高的保守性(平均同一性=74%),而在结合位点的相对位置上差异最小(平均差异Δ d日 =469个碱基)。 事实上,如果预测的ERE为 绿色1 (人类, NM_014668号 ; 鼠标, NM_015764号 )其相对位置相差1.7kb,被排除在分析之外。 对于核心序列中有一个或多个碱基偏差的ERE鼠-人对,侧翼序列和预测ERE的相对位置几乎没有守恒性(见表 2 ). 这些发现表明,尽管ERE基序在进化过程中是保守的,但在靶基因的5'调控区发现的特定ERE很少保守。 他们还表明,人类和啮齿动物在内质网功能的分子机制和靶基因库方面存在潜在差异。 有鉴于此,我们根据动物研究结果对人类ER功能的推断可能需要重新评估和额外验证。
图6。
人类和小鼠直系亲属的比较。 (a) 来自人类和小鼠同源假定靶基因对中预测ERE的比较分析的统计数据。 (b) 维恩图显示,在提取用于分析的72对同源对中,只有22对在人类和小鼠序列中进行了ERE预测。
表2。
基因符号 人类 鼠标 %身份* 相对位置*(Δ d日 )
参考序列ID 预测的数量 参考序列ID 预测的数量 核心ERE 侧翼区域
ALOX12B型 NM_001139号 三 纳米_009659 4 100 87 40
自然保护区1 NM_003489 2 173440纳米 4 100 72 70
绿色1 NM_014668号 2 NM_015764号 2 100 72 1,720
ACP33型 NM_016630 三 NM_138584 4 100 66 46
10层 NM_000504号 2 NM_007972号 6 90 49 2,003
SERPINE1系列 NM_000602号 4 NM_008871 三 90 23 41
CTSD公司 NM_001909号 三 NM_009983号 1 90 26 1,310
OAZIN公司 NM_015878号 1 NM_018745号 1 90 19 2,264
PADI3型 NM_016233号 4 NM_011060 2 90 21 4,153
未知 NM_017770 2 NM_019423 1 90 28 3,828
JDP1 纳米_021800 1 NM_013888号 1 90 30 1,089
NIF3L1 NM_021824 1 NM_022988号 2 90 28 955
SCN1B型 纳米_001037 2 NM_011322 1 80 36 2,716
STC2系统 NM_003714号 1 NM_011491号 2 80 26 2638个
LOR公司 NM_000427号 2 NM_008508 2 70 32 4,253
ADCY9公司 NM_001116号 4 NM_009624号 1 70 28 3,042
AHCYL1型 NM_006621号 1 NM_145542 2 70 32 2,190
CISH公司 NM_013324 1 NM_009895号 三 70 19 1,624
热休克蛋白111 NM_016391 1 NM_178605 1 70 19 1,302
FLJ22269型 NM_032219 2 NM_172883 1 70 47 3,391
DNAJC1公司 NM_022365号 1 NM_007869 1 60 30 1,672
CDC42EP3系统 NM_006449 1 纳米_026514 1 50 25 451
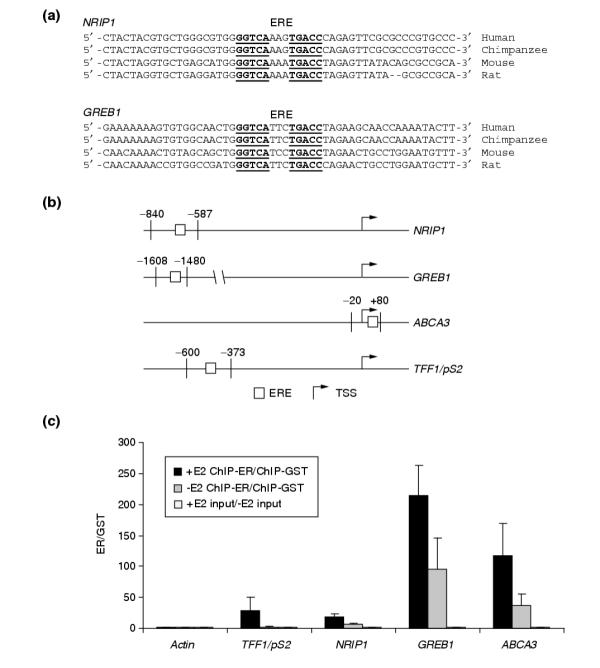
染色质免疫沉淀法验证直接内质网靶基因 基因组学和信息学方法使我们能够识别符合ER靶基因传统定义的基因(例如,对E2敏感、对ICI敏感和对CHX不敏感),这些基因在ER中是保守的 + 乳腺癌细胞系和肿瘤样本,并在启动子区域编码推测的ER结合位点。 在这些分析之后,两个基因出现在直接靶基因列表的顶部。 一个是核受体相互作用蛋白1(NRIP1),也称为受体相互作用蛋白质140(RIP140),最初被鉴定为ER-结合蛋白和受体活性的共同调节蛋白[ 44 , 45 ]. 随后发现它能结合和调节其他核受体的转录活性[ 46 , 47 ]. 先前在MCF-7和ZR75-1细胞中的微阵列实验表明 自然保护区1 雌激素治疗后转录水平升高,其在抗雌激素和CHX存在下的表达动力学与其他主要反应基因一致[ 23 , 24 , 26 ]. 在本研究中,我们还确定了 自然保护区1 作为一个被E2上调的ER靶基因,对ICI治疗敏感,对T-47D细胞中的CHX不敏感。 此外,我们在TSS上游约700个碱基处检测到一个保守的完美ERE,这表明存在一个潜在的ER-结合位点,并由激活的受体直接调控。 乳腺癌中雌激素调节的另一个直接靶基因1( 绿色1 )-在MCF-7细胞中雌激素反应基因的消减杂交筛选中鉴定。 它没有已知的功能,似乎与序列数据库中的任何其他基因没有显著同源性[ 48 ]. 在TSS上游约1.6 kb处发现了完美的ERE 绿色1 并且预测的ERE在小鼠中也很保守。 考虑到这两者 自然保护区1 和 绿色1 我们比较了人类、黑猩猩、小鼠和大鼠基因组序列的5'上游区域,以了解预测的调控元件是否在其他鼠和灵长类物种中得到了保护。 对于所有被调查的地区,我们发现核心ERE已经被完全保存(图 第7页 ). 此外,预测ERE两侧的序列也高度保守,表明这些区域具有功能性。
图7。
ER以雌激素依赖的方式结合编码保守和非保守预测反应元件的启动子区域。 (a) 在上游发现ERE(带下划线) 自然保护区1 和 绿色1 编码区在人类、黑猩猩、小鼠和大鼠基因组中是保守的。 (b) 预测保守基因侧翼的PCR引物( 自然保护区1 和 绿色1 )和非服务( 澳大利亚广播公司3 和 TFF1/pS2 )ERE被设计用于检测ChIP测定后的ER结合。 指出了引物和ERE相对于TSS的相对位置。 (c) 雌激素治疗增强了ER和预测ERE之间的相互作用。 MCF-7细胞要么用载体二甲基亚砜(-E2,灰色条)模拟处理,要么用雌二醇(+E2,黑色条)处理,然后进行ChIP实验。 黑色和灰色条表示抗ER ChIP实验中结合位点的富集程度高于抗GST ChIP实验。 在激素处理的细胞中观察到所有ERE的富集,而模拟处理的细胞显示较少或很少富集。 肌动蛋白外显子3控制区或任何输入对照(开条)均未富集。
为了确定预测ERE作为ER-结合位点的作用,我们使用抗ER抗体进行染色质免疫沉淀(ChIPs)。 除了两个保守的ERE外,我们还包括来自 TFF1/pS2 (阳性对照)和ATP结合盒,亚家族A,成员3( 澳大利亚广播公司3 ),一个与其他ABC转运体相关的基因,被认为参与细胞脂质转运,是本研究和先前研究确定的推测ER直接靶基因[ 26 ]. 正向和反向引物(图 7亿 )ERE侧翼设计用于在实时PCR实验中特异检测和量化与ER共沉淀的基因组DNA片段。 在激素治疗后,与抗GST抗体对照或所有测试引物对的核裂解物的输入基因组DNA相比,我们没有观察到抗ER沉淀物中阴性对照肌动蛋白外显子3区域的显著富集(图 第7页c ). 相反,使用预测ERE两侧的引物对ChIP产物进行半定量PCR分析(参见材料和方法),结果显示,在没有雌激素和激素治疗后,ER与这些位点结合(参见图 第7页c ). 此外,雌激素治疗后,这种结合似乎增强了,这表明激活的受体在介导观察到的这些基因的转录调控中发挥了作用。 保守ERE的功能 自然保护区1 和 绿色1 最近在一项关于人类和小鼠基因组中近一致ERE的研究中也有报道[ 49 ].
讨论 我们对E2应答基因进行了全基因组分析。 通过使用基因组学、信息学和实验生物学的迭代验证策略,我们在我们的微阵列上表示的18912个基因中(0.5%),识别并表征了89个ER直接靶基因的核心集。 这组来自T-47D细胞实验的直接靶基因在另一种细胞系MCF-7中表现出非常相似的行为,也与能够区分人类乳腺癌ER状态的基因重叠。 总之,这些结果表明了ER转录控制的共同潜在机制,并定义了ER转录网络的特定遗传成分,这些成分在模型实验和临床条件中是一致的。
这些结果使我们有勇气破译ER转录控制的规则和信息框架。 ICI药物的抗雌激素治疗和CHX细胞的预培养使我们能够确定可能是ER直接靶点的基因。 我们提取了扩展的启动子区域(-3000 bp到+500 bp),并使用ERE、AP-1和Sp1结合位点模型确定了潜在的ER-结合位点[ 41 , 42 ]. 由于转录因子结合元件在基因组中频繁出现,因此仅在ER反应基因中发现ERE、AP-1或Sp1位点是极不可能的。 相反,我们询问是否发现与雌激素受体相关的反应元件的概率显著高于雌激素受体无反应基因。 我们的结果如图所示 5 ,清楚地表明ERE富含推测的直接雌激素反应基因( 第页 =0.0027),但AP1、Sp1或GATA1(我们用作阴性对照反应元件)的结合位点不是。 因此,ERE似乎是主要的ER转录控制元件。 此外,尽管有明确的实验表明AP-1和Sp1位点具有介导内质网反应的能力[ 7 - 9 , 47 , 50 - 54 ],我们的结果表明,在全基因组范围内,它们的使用并不是特定ER转录控制的常见机制。
先前的研究发现 铝 基因组中经常出现的重复序列[ 55 ]. 包含此内容 铝 然而,在我们的分析中,ERE显著降低了在直接ER反应基因中发现的ERE的富集( 第页 = 0.06). 这表明,尽管这些ERE在实验上具有功能,但它们对特定的ER转录盒几乎没有影响,在系统中只起到“噪音”的作用。 这已被几个国家的负面ChIP数据证实 铝 -ERE站点(数据未显示)。 这些观察结果突出了功能相关反应元件全基因组分析中潜在的混杂因素。
我们使用TSS周围的3.5 kb窗口来搜索相关的ERE,捕获了大多数已知的ERE[ 5 ]并代表了对5个监管区域的自由调查。 尽管如此,我们发现只有约50%的靶基因在其启动子中编码ERE样序列(包括ERE半位点)。 这一窗口之外的雌激素受体结合位点可能参与调节雌激素受体的具体活动。 为了支持这一点,Bourdeu及其同事最近描述了一些人类基因5'区域DNA上游10kb(相对于TSS)和下游5kb内ERE的识别和验证[ 49 ]表明在我们研究的区域之外存在功能增强因子。 此外,TSS或额外5’外显子的注释错误可能导致已知基因中TSS测定的错误率高达8%,预测基因中的错误率为80%(Y.J.Ruan、E.T.L.和C.L.Wei,未发表的工作)。 未来的研究需要将这些信息纳入ERE分析。
鉴于此 生物信息学 ERE的鉴定并不能保证其在ER反应中的功能,我们选择了三个新的推测的直接ER靶基因,这些基因是根据我们严格的标准鉴定出来的,以供进一步验证。 自然保护区1 , 绿色1 、和 澳大利亚广播公司3 是否所有基因在至少两个细胞系中都有ER反应; 它们在TSS周围有一个可识别的ERE,被ICI阻断而不被CHX抑制,它们的表达可以识别乳腺癌中的ER状态。 通过使用ChIP,我们确认了雌激素刺激后,所有三种雌激素的雌激素受体直接靶向(图 7 ). 因此,我们通过共识进行排名的过程(即通过满足的标准数量,根据成为ER直接目标的可能性进行排名)似乎是确定ER实际直接目标的合理方法。
这些靶基因表明ER在调节细胞内信号通路方面可能发挥潜在作用,这些信号通路可能对乳腺和肿瘤生物学过程产生影响。 自然保护区1 最初被鉴定为一种ER结合共调节蛋白,随后发现通过核受体结合基序LXXLL与其他核受体相互作用。 Kerley和同事[ 56 ]表明了这一点 自然保护区1 转录物和蛋白质水平也被上调- 反式 维甲酸治疗并提示 自然保护区1 可以促进核受体家族成员之间的串扰。 因此 自然保护区1 被激活的ER不仅可以调节雌激素反应,还可以影响其他核受体的转录活性以及细胞对其相应配体的反应。 那个 自然保护区1 ER中转录水平升高 + 与ER相比 - 乳腺肿瘤表明,其他核激素受体的下游功能可能受到ER转录级联元件的协调调节(见图 4 ).
澳大利亚广播公司3 编码ABC转运体的一个成员,利用ATP水解来驱动底物跨细胞膜运输; 虽然其底物未知,但ABCA3似乎与参与脂质转运的其他ABC转运蛋白有关。 ABCA3蛋白水平在肺组织中最高,ABCA3似乎定位于富含磷脂酰胆碱的肺泡上皮细胞的板层体[ 57 ]. 这些观察结果提供了ER激活与乳腺上皮细胞分化和转化过程中磷脂水平变化之间的潜在联系。 绿色1 然而,它是一个功能未知的基因,与任何其他已知基因无关。 其在ER中过度表达 + 乳腺肿瘤及其进化保护表明该基因在ER信号传导和乳腺肿瘤生物学中起着重要作用。 值得注意的是,本研究中确定的21%(19/89)的假定靶基因没有已知的生物功能(见表 1 ).
评估中使用的一种策略 顺式 -基因组中的调控元件一直在绘制TSS上游非编码区的保守片段。 使用上述三个基因( 自然保护区1 , 澳大利亚广播公司3 和 绿色1 )作为内质网监管的严格测试直接目标和经过充分研究的内质网直接目标, TFF1/pS2 ,我们评估了人类和小鼠同源物之间验证的上游ERE的进化保守性。 有趣的是,我们发现高度保守的ERE(包括侧翼区域)仅适用于 自然保护区1 和 绿色1 . 澳大利亚广播公司3 和 TFF1/pS2 两者在人类基因中都有上游功能性ERE,但在小鼠同源基因中没有(图 7亿 ).
然后,我们将这一进化保守性搜索扩展到其余89个假定的人类内质网直接靶基因。 令人惊讶的是,我们发现在大多数鼠-人同源对中,ERE核心序列、侧翼区域和相对于TSS的位置并不保守:在检测的72对可测试的同源对中只有4对(6%)显示人和鼠基因之间的ERE序列保守。 鉴于84.7%的人口,这一点值得注意[ 58 ]编码区域内老鼠和人类序列之间的一致性。 综上所述,我们的结果表明转录控制的进化通过 顺式 -调控机制必须具有不同的突变率或机制,并且可能经历了与施加在编码序列上的选择压力不同的选择压力。 此外,小鼠和人类雌激素反应基因ERE的低水平保守表明了两个后果:首先,核心生理雌激素效应,如性别分化/乳腺发育,可能由一小部分高度保守和类似调节的雌激素反应基因介导; 其次,小鼠和人类的下游雌激素效应可能存在显著差异。
我们通过显示MCF-7细胞中雌激素诱导的表达谱和这些基因在ER中的行为之间的显著相似性,提出了许多雌激素反应基因与乳腺肿瘤生物学的相关性 + 六种乳腺癌微阵列研究数据库中的肿瘤[ 36 - 40 ]. 不出所料,我们观察到,与定义雌激素受体的基因总数相比,直接雌激素反应基因的数量很少 + 其他人也注意到,乳腺肿瘤表明雌激素反应途径仅占受体阳性分子信号的一部分[ 22 ]. 然而,综上所述,ER似乎 + 原发性乳腺癌的状态可以通过激活ER的协同效应来解释。有趣的是,与肿瘤共识相比,这些差异表达的基因(约30%)在细胞系中的表达方向相反。 我们推测,这可能是由于内质网辅因子的持续不同特征,或者是由于肿瘤相关基质细胞中与癌细胞相反的强烈表达特征。 然而,那些在细胞系中对雌激素有反应并且在雌激素受体中差异表达的基因 + 肿瘤是最有希望进行进一步功能分析的候选肿瘤。
总之,我们提出了一种发现和表征内质网靶基因、反应元件和内质网激活下游转录调控网络的综合策略。 通过这种方法,我们发现了一组通用的基因,这些基因描述了内质网最直接的影响,并在多个基因中起作用 在体外 和 体内 系统。 经检查,这个核心直接靶基因列表并不能预测由ER控制的统一生物过程。相反,基因功能可以预测多效性细胞反应。 通过进一步 生物信息学 通过对启动子区域的分析,我们观察到 顺式 -人类和小鼠间直接雌激素反应基因的调节区。 这提出了一个有趣的可能性,即控制转录调控结构的进化过程将不同于影响基因功能域的进化过程。 此外,我们预测小鼠的雌激素反应与人类的明显不同,但一小部分雌激素受体直接靶基因在其体内高度保守 顺式 -调控区域将成为进化上重要的核心内质网功能(如性别分化)的关键效应器。
结论 人类乳腺癌细胞中的雌激素反应似乎由一组相对较小的保守的ER调节核心基因介导。 检查 顺式 -该核心组中假定靶基因的调控区域显示ERE序列基序丰富,但没有其他已知的ER-结合位点。 在人类直接靶基因中预测的所有ERE中,只有少数(6%)在小鼠直系同源基因中保守,尽管保守和非保守预测的ERE在人类细胞系中都与ER结合。 综上所述,这些发现表明了激素暴露对物种特异性机制和效应的潜在影响。
材料和方法 细胞培养、处理和RNA提取 T-47D和MCF-7细胞保存在37°C添加10%胎牛血清(FCS)(Hyclone)的DMEM/F12(1:1)培养基(Invitrogen)中,并用5%CO缓冲 2 对于雌激素治疗,用PBS清洗细胞,并在无酚DMEM/F12培养基中预先培养24小时,培养基中添加0.5%碳过滤FCS(Hyclone)。对于时间过程实验,用1 nM 17β-雌二醇(E2;Sigma-Aldrich)或1 nM E2+10 nM ICI 182,780(Tocris Cookson)处理T-47D细胞 在指定的时间内。 为了确定主要反应,在开始雌激素治疗之前,用5μg/ml环己酰亚胺(CHX;Sigma-Aldrich)处理细胞30分钟。 在规定的时间和相同的浓度下,也使用ICI(2、8、12和24小时)和CHX(2和8小时)单独进行对照处理。 为了提取RNA,用PBS清洗细胞,在Trizol(Invitrogen)中溶解,并按照制造商的规定通过额外的酚-氯仿提取步骤收集样品。
基因表达谱的微阵列分析 通过在聚赖氨酸涂层的玻璃载玻片上发现由Sigma-Genosys制造的Compugen 19K人类寡核苷酸库,生成微阵列。 每个样本总RNA和人类通用参考RNA(Stratagene)的25微克分别用Cy5-共轭dUTP和Cy3-共轭dUTP-(PerkinElmer)标记,并使用Patrick O.Brown实验室制定的协议与阵列杂交[ 59 ]. 使用GenePix4000B扫描仪和GenePix Pro软件(Axon Instruments)获取并处理阵列图像和数据。 用两两法测定差异表达基因 t吨 -在每个时间点对匹配的处理过的样本和模拟处理过的对照进行测试,并在多个时间点进行折叠差截止,如所述,使用艾森聚类和TreeView程序进行聚类和可视化[ 30 ]. 推定目标基因的基因本体论来源于Compugen的注释。
乳腺癌和细胞系微阵列数据的Meta分析 对包含已发表和未发表乳腺癌表达数据的数据库进行查询,以获取其表达谱区分ER的基因 + 和ER - 肿瘤。 通过计算每个基因的错误发现率,独立分析每个数据集的差异表达基因[ 60 ]并设置 第页 -值过滤器小于或等于0.01。 然后将ER状态相关基因与 在体外 通过UniGene簇ID(构建166)的雌激素反应基因。 使用每个具有统计学意义的研究的对数转换平均以平均值为中心的表达值进行可视化。 原始 在体外 MCF-7雌激素反应数据[ 31 ]从斯坦福微阵列数据库下载[ 32 ]. 这些数据直接与T-47D结果进行比较,或根据以下选择标准选择用于ER调节:首先,在三个时间点中的两个时间点,在同一方向上至少有1.15倍的变化,并且在任何时间点都没有冲突(相反方向)的数据; 第二,在两种治疗条件中的其中一种条件下,与三苯氧胺(Tam)联合治疗48小时后,变化方向相反,两种治疗中没有相互矛盾的数据。 1.15倍的截止值不同于T-47D数据的1.2倍变化,用于捕获该数据集中已知的E2应答基因。
启动子序列提取与ER结合位点检测 LocusLink和RefSeq[ 61 ]美国国家生物技术信息中心(NCBI)的数据库用于识别人类和小鼠基因,并精确定位其在基因组中的位置。 选择这些注释是因为它们的全面性、注释基因的数量以及它们与NCBI contig数据库当前状态的一致性。 使用TSS(定义为参考转录物中最多的5'核苷酸)和转录物的3'末端作为参考点,我们提取起始位点上游3kb和下游500个碱基用于结合位点分析。 NCBI人类基因组序列构建33和小鼠基因组序列构建30用于转录比对和基因组序列提取。 由于某些参考序列5'端的信息不完整,在LocusLink和RefSeq中注释的TSS位置可能仅近似于真实起始点,但我们认为用于我们分析的相对较大(3.5 kb)区域允许TSS位置的波动。 人类-小鼠同源基因测定基于小鼠基因组数据库中的注释[ 43 ]. 所用的四个结合位点位置权重矩阵(PWM)模型要么是在早期研究中推导出来的[ 41 ]或从TRANSFAC(6.0版)转录因子结合位点数据库下载[ 42 ]. 检测参数是根据Dragon ERE Finder的优化设置设置的[ 41 ]以83%的灵敏度检测训练数据,并对其他PWM进行相应设置,使其具有类似的灵敏度。 通过蒙特卡罗模拟,在定义的基因集和随机生成的基因集的预测之间,确定假定目标基因和非响应基因之间结合位点富集的统计意义。 进行了一组蒙特卡罗模拟,以评估一组雌激素直接靶基因和非响应基因之间假定ERE明显富集的重要性。 在每次模拟中,我们随机生成两组基因(大小相当于直接靶基因和非响应基因),绘制相应的曲线,并计算两条曲线下面积的差异。 模拟执行了100000000次,模拟中随机区域差异至少与观察到的区域差异一样大的时间分数报告为经验值 第页 -值。 分析中使用的最重要的直接靶基因排名最低 第页 -E2处理样品和对照样品、E2和E2+ICI样品以及E2和E_2+CHX样品的分析值。 无反应基因排名最高 第页 -来自相同分析的值。
染色质免疫沉淀分析 MCF7细胞被剥夺雌激素24小时,并用100nM E2处理45分钟,然后1%甲醛处理以交联转录机制和染色质。 使用ERα(HC-20)或GST抗体(Santa-Cruz Biotechnology)和蛋白A-sepharose珠(Zymed)隔夜进行免疫沉淀。 清洗和提取方案根据之前描述的方法进行了修改[ 62 ]PCR反应在LightCycler(Roche Diagnostics)实时系统中进行。 使用以下引物对沉淀DNA和对照输入DNA进行40个PCR周期: TFF1/pS2 ERE:远期CCATGTGGCACGCTAGTC; 背面为ACAACAGTGGCTCACGGGT。 自然保护区1 ERE:转发,TGCTCCTGGGTCCACGTCT; 反转TCCCCTTCACCCACAACACACA。 绿色1 ERE:正向AGCAGTGAAAAAAGTGGCAACTGGG; 背面为CGACCCACAGAAAATGAAAAGGCAGACAACT。 澳大利亚广播公司3 ERE:转发,CACCTTCCATCTGTCCAAAG; 背面为CAACCCTGGGTTGGGAAC。 肌动蛋白外显子3控制:正向,AGACCTTCAACCCCACACCC; 背面为GTCACGCACGGATTCCCGCT。 在每次运行结束时,还通过熔化曲线分析来分析扩增产物的特异性。 通过建立每个引物集的标准曲线,并使用与输入相似的基因组DNA作为模板,进行相对定量。 通过比较抗ER和对照抗GST产物的相对数量来确定ER结合的富集。
其他数据文件 本文的在线版本提供了以下附加数据文件:时间进程微阵列研究的处理原始数据(附加数据文件 1 )和复制1(附加数据文件 2 )和复制2(附加数据文件 三 )在仅使用ICI和CHX治疗的对照微阵列研究中,文章中描述的所有387个雌激素反应基因的完整列表,以及构建166的UniGene簇号(附加数据文件 4 ),对照实验中确定的ICI和CHX应答基因的表达谱(附加数据文件 5 )和相应的图形图例(附加数据文件 6 ).
补充材料
附加数据文件2 仅用ICI和CHX治疗复制1个对照微阵列研究
附加数据文件3 用单独的ICI和CHX处理复制对照微阵列研究的2
附加数据文件4 文章中描述的所有387个雌激素反应基因的完整列表,UniGene簇编号来自构建166
附加数据文件5 对照实验中确定的ICI和CHX应答基因的表达谱
附加数据文件6 对照实验中确定的ICI和CHX应答基因表达谱的对应图例
致谢 致谢 我们感谢Soek Ying Neo、Suk Woo Nam和Christopher Wong对微阵列技术的帮助,感谢Phillip Long、Joshy George和Radha Krishna Murthy Karuturi对数据分析的有益讨论。 我们还感谢乔治·里德对手稿提出了深刻的评论。 在新加坡基因组研究所进行的研究得到了新加坡科学、技术和研究局(A*STAR)生物医学研究理事会(BMRC)的资助。
参与者信息 Chin-Yo Lin,电子邮件: lincy@gis.a-star.edu.sg。
Anders Ström,电子邮件: anders.strom@cbt.ki.se。
Vinsensius Berlian Vega,电子邮件: vegav@gis.a-star.edu.sg。
说李刚,电子邮件: kongsl@gis.a-star.edu.sg。
艾莉Yeo,电子邮件: yeoal2@gis.a-star.edu.sg。
Jane S Thomsen,电子邮件: jane.thomsen@biosci.ki.se。
Wan Ching Chan,电子邮件: domdom7@charter.net。
Balraj Doray,电子邮件: bd@im.wustl.edu。
Dhinoth K Bangarusamy,电子邮件: bangarusd@gis.a-star.edu.sg。
Adaikalavan Ramasamy,电子邮件: adaikalavan.ramasamy@cancer.org.uk。
Liza A Vergara,电子邮件: vergarala@gis.a-star.edu.sg。
Tang Suisheng,电子邮箱: suisheng@i2r.a-star.edu.sg。
Allen Chong,电子邮件: achong@i2r.a-star.edu.sg。
Vladimir B Bajic,电子邮件: bajicv@i2r.a-star.edu.sg。
Lance D Miller,电子邮件: millerl@gis.a-star.edu.sg。
Jan-Oke Gustafsson,电子邮件: jan-ake.gustafson@mednut.ki.se。
Edison T Liu,电子邮件: liue@gis.a-star.edu.sg。
工具书类
Korach KS、Emmen JM、Walker VR、Hewitt SC、Yates M、Hall JM、Swope DL、Harrell JC、Couse JF。为分析雌激素受体生物活性而开发的动物模型的更新。 类固醇生物化学分子生物学杂志。 2003; 86:387–391. doi:10.1016/S0960-0760(03)00348-0。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Ali S,Coombes钢筋混凝土。 雌激素受体α在人类乳腺癌中的发生及意义。 乳腺生物肿瘤杂志。 2000; 5:271–281. doi:10.1023/A:1009594727358。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Persson I.雌激素与乳腺癌、子宫内膜癌和卵巢癌的病因——流行病学研究的证据和假设。 类固醇生物化学分子生物学杂志。 2000; 74:357–364. doi:10.1016/S0960-0760(00)00113-8。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Nilsson S,Gustafsson J。 雌激素受体的作用。 真核生物基因实验关键评论。 2002; 12:237–257. doi:10.1615/critreveukaryotgeneexpr.v12.i4.10。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
克林格CM。雌激素受体与雌激素反应元件的相互作用。 2001年《核酸研究》; 29:2905–2919. doi:10.1093/nar/29.14.2905。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Kushner PJ、Agard DA、Greene GL、Scanlan TS、Shiau AK、Uht RM、Webb P.到AP-1的雌激素受体途径。 类固醇生物化学分子生物学杂志。 2000; 74:311–317. doi:10.1016/S0960-0760(00)00108-4。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Saville B、Wormke M、Wang F、Nguyen T、Enmark E、Kuiper G、Gustafsson Já、Safe S.配体、细胞和雌激素受体亚型(α/β)依赖于GC-rich(Sp1)启动子元件的激活。 生物化学杂志。 2000; 275:5379–5387. doi:10.1074/jbc.275.8.5379。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Porter W,Saville B,Hoivik D,Safe S.转录因子Sp1和雌激素受体之间的功能协同作用。 摩尔内分泌。 1997; 11:1569–1580. doi:10.1210/me.11.11.1569。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Paech K、Webb P、Kuiper GG、Nilsson S、Gustafsson Já、Kushner PJ、Scanlan TS。AP1位点雌激素受体ERalpha和ERbeta的差异配体激活。 科学。 1997; 277:1508–1510. doi:10.1126/science.277.5331.1508。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
McKenna NJ,O'Malley BW。核受体和协同调节剂对基因表达的组合控制。 单元格。 2002; 108:465–474. doi:10.1016/S0092-8674(02)00641-4。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Shang Y,Hu X,DiRenzo J,Lazar MA,Brown M.雌激素受体调节转录的辅因子动力学和充分性。 单元格。 2000; 103:843–852. doi:10.1016/S0092-8674(00)00188-4。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Reid G、Hubner MR、Metivier R、Brand H、Denger S、Manu D、Beaudouin J、Ellenberg J、Gannon F.Cyclic,蛋白酶体介导的反应性启动子上未连接和配体ERalpha的周转是雌激素信号传导的一个整体特征。 分子细胞。 2003; 11:695–707. doi:10.1016/S1097-2765(03)00090-X。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Metivier R、Penot G、Hubner MR、Reid G、Brand H、Kos M、Gannon F.雌激素受体alpha指导天然靶基因启动子上辅酶因子的有序、周期性和组合招募。 单元格。 2003; 115:751–763. doi:10.1016/S0092-8674(03)00934-6。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Sanchez R,Nguyen D,Rocha W,White JH,Mader S.雌激素受体基因调节机制的多样性。 生物论文。 2002; 24:244–254. doi:10.1002/bies.10066。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Jakowlew SB、Breathnach R、Jeltsch JM、Masiakowski P、Chambon P。雌激素在人类乳腺癌细胞系MCF-7中诱导的pS2 mRNA序列。 《核酸研究》1984; 12:2861–2878. doi:10.1093/nar/12.62861。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Morisset M,Capony F,Rochefort H。MCF7细胞分泌的52-kDa雌激素诱导蛋白是一种溶酶体酸性蛋白酶。 生物化学与生物物理研究委员会。 1986; 138:102–109. doi:10.1016/0006-291x(86)90252-4。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Altucci L、Addeo R、Cicatiello L、Dauvois S、Parker MG、Truss M、Beato M、Sica V、Bresciani F、Weisz A.17beta-Estradiol在G(1)阻滞的人类乳腺癌细胞的有丝分裂刺激期间诱导细胞周期蛋白D1基因转录、p36D1-p34cdk4复合物激活和p105Rb磷酸化。 致癌物。 1996; 12:2315–2324. [ 公共医学 ] [ 谷歌学者 ]
Foster JS,Wimalasena J.雌激素调节乳腺癌细胞中细胞周期素依赖性激酶的活性和视网膜母细胞瘤蛋白磷酸化。 摩尔内分泌。 1996; 10:488–498. doi:10.1210/me.10.5.488。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Dubik D,Dembinski TC,Shiu RP。刺激与雌激素诱导的人类乳腺癌细胞增殖相关的c-myc癌基因表达。 1987年癌症研究; 47:6517–6521. [ 公共医学 ] [ 谷歌学者 ]
Yu WC、Leung BS、Gao YL。 17β-雌二醇对人乳腺癌细胞系CAMA-1中孕酮受体和胸苷摄取的影响。 1981年癌症研究; 41:5004–5009. [ 公共医学 ] [ 谷歌学者 ]
Coser KR、Chesnes J、Hur J、Ray S、Isselbacher KJ、Shioda T。利用DNA微阵列对MCF7/BUS乳腺癌细胞中雌激素诱导和抑制基因的配体敏感性进行全球分析。 美国国家科学院院刊,2003年; 100:13994–13999. doi:10.1073/pnas.2235866100。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Cunliffe HE,Ringner M,Bilke S,Walker RL,Cheung JM,Chen Y,Meltzer PS。乳腺癌对生长调节剂的基因表达反应:模式和与肿瘤表达谱的相关性。 2003年癌症研究; 63:7158–7166. [ 公共医学 ] [ 谷歌学者 ]
Frasor J、Danes JM、Komm B、Chang KC、Lyttle CR、Katzenellenbogen BS。人类乳腺癌细胞中雌激素上调和下调基因表达的分析:深入了解雌激素控制增殖和细胞表型的基因网络和途径。 内分泌学。 2003; 144:4562–4574。 doi:10.1210/en.2003-0567。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Inoue A、Yoshida N、Omoto Y、Oguchi S、Yamori T、Kiyama R、Hayashi S。雌激素反应基因表达谱cDNA微阵列的开发。 分子内分泌杂志。 2002; 29:175–192. doi:10.1677/jme.0.290175。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Lobenhofer EK,Bennett L,Cable PL,Li L,Bushel PR,Afshari CA。17β-雌二醇对DNA复制叉基因的调节。 摩尔内分泌。 2002; 16:1215–1229。 doi:10.1210米/米16.6.1215。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Soulez M,Parker MG.通过表达谱鉴定人类ZR75-1乳腺癌细胞中新的雌激素受体靶基因。 分子内分泌杂志。 2001; 27:259–274. doi:10.1677/jme.0.270259。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Wang DY、Fulthorpe R、Liss SN、Edwards EA。 通过互补脱氧核糖核酸微阵列鉴定雌激素反应基因,并鉴定一个新的早期雌激素诱导基因:EEIG1。 摩尔内分泌。 2004; 18:402–411. doi:10.1210/me.2003-0202。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Keydar I、Chen L、Karby S、Weiss FR、Delarea J、Radu M、Chaitcik S、Brenner HJ。 人乳腺癌起源细胞系的建立和鉴定。 《欧洲癌症杂志》。 1979; 15:659–670. doi:10.1016/0014-2964(79)90139-7。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Magklara A、Brown TJ、Diamandis EP。 显示激肽释放酶2和3差异调节的人类乳腺癌细胞系中雄激素受体和核受体共同调节因子表达的特征。 国际癌症杂志。 2002; 100:507–514. doi:10.1002/ijc.10520。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Eisen MB、Spellman PT、Brown PO、Botstein D.全基因组表达模式的聚类分析和显示。 美国国家科学院院刊,1998年; 95:14863–14868。 doi:10.1073/pnas.95.25.14863。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Finlin BS、Gau CL、Murphy GA、Shao H、Kimel T、Seitz RS、Chiu YF、Botstein D、Brown PO、Der CJ等。RERG是乳腺癌中一种新的ras相关、雌激素调节和生长抑制基因。 生物化学杂志。 2001; 276:42259–42267. doi:10.1074/jbc。 M105888200。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Gollub J、Ball CA、Binkley G、Demeter J、Finkelstein DB、Hebert JM、Hernandez-Boussard T、Jin H、Kaloper M、Matese JC等。斯坦福微阵列数据库:数据访问和质量评估工具。 核酸研究2003; 31:94–96. doi:10.1093/nar/gkg078。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Fortunel NO,Otu HH,Ng HH,Chen J,Mu X,Chevassut T,Li X,Joseph M,Bailey C,Hatzfeld JA,et al.评论“干细胞”:胚胎和成人干细胞的转录图谱”和“干细胞分子特征”。 科学。 2003; 302:393. doi:10.1126/science.1086384。 作者回复393。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Hui R、Cornish AL、McClelland RA、Robertson JF、Blamey RW、Musgrove EA、Nicholson RI、Sutherland RL。细胞周期蛋白D1和雌激素受体信使RNA水平与原发性乳腺癌呈正相关。 1996年临床癌症研究; 2:923–928. [ 公共医学 ] [ 谷歌学者 ]
Wysocki SJ,Hahnel E,Wilkinson SP,Smith V,Hahnel-R.乳腺肿瘤中激素敏感基因的表达。 1990年抗癌研究; 10:185–188。 [ 公共医学 ] [ 谷歌学者 ]
Sorlie T、Perou CM、Tibshirani R、Aas T、Geisler S、Johnsen H、Hastie T、Eisen MB、van de Rijn M、Jeffrey SS等。乳腺癌的基因表达模式区分具有临床意义的肿瘤亚类。 美国国家科学院院刊,2001年; 98:10869–10874. doi:10.1073/pnas.191367098。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
van t Veer LJ、Dai H、van de Vijver MJ、He YD、Hart AA、Mao M、Peterse HL、van der Kooy K、Marton MJ、Witteveen AT等。基因表达谱预测乳腺癌的临床结局。 自然。 2002; 415:530–536. doi:10.1038/415530a。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Sotiriou C、Neo SY、McShane LM、Korn EL、Long PM、Jazaeri A、Martiat P、Fox SB、Harris AL、Liu ET。基于人群研究中基因表达谱的乳腺癌分类和预后。 美国国家科学院院刊,2003年; 100:10393–10398. doi:10.1073/pnas.1732912100。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Gruvberger S、Ringner M、Chen Y、Panavally S、Saal LH、Borg A、Ferno M、Peterson C、Meltzer PS。乳腺癌雌激素受体状态与显著不同的基因表达模式相关。 2001年癌症研究; 61:5979–5984. [ 公共医学 ] [ 谷歌学者 ]
West M,Blanchette C,Dressman H,Huang E,Ishida S,Spang R,Zuzan H,Olson JA,Jr,Marks Jr,Nevins Jr。利用基因表达谱预测人类乳腺癌的临床状态。 美国国家科学院院刊,2001年; 98:11462–11467. doi:10.1073/pnas.201162998。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Bajic VB、Tan SL、Chong A、Tang S、Strom A、Gustafsson JÅ、Lin CY、Liu ET。Dragon ERE Finder版本2:用于准确检测和分析脊椎动物基因组中雌激素反应元件的工具。 核酸研究2003; 31:3605–3607. doi:10.1093/nar/gkg517。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Heinemeyer T,Chen X,Karas H,Kel AE,Kel OV,Liebich I,Meinhardt T,Reuter I,Schacherer F,Wingender E。将TRANSFAC数据库扩展为调控分子机制的专家系统。 《核酸研究》,1999年; 27:318–322. doi:10.1093/nar/27.1318。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Blake JA、Richardson JE、Bult CJ、Kadin JA、Eppig JT。 MGD:小鼠基因组数据库。 核酸研究2003; 31:193–195. doi:10.1093/nar/gkg047。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Cavailles V、Dauvois S、L’Horset F、Lopez G、Hoare S、Kushner PJ、Parker MG。核因子RIP140通过雌激素受体调节转录激活。 EMBO J.1995; 14:3741–3751. doi:10.1002/j.1460-2075.1995.tb00044.x。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Treuter E、Albrektsen T、Johansson L、Leers J、Gustafsson Já。 RIP140在核受体激活中的调节作用。 摩尔内分泌。 1998; 12:864–881. doi:10.1210/me.12.6.864。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Miyata KS、McCaw SE、Meertens LM、Patel HV、Rachubinski RA、Capone JP。受体相互作用蛋白140与过氧化物酶体增殖物激活受体α和肝脏X受体α相互作用并抑制其反式激活。 分子细胞内分泌。 1998; 146:69–76. doi:10.1016/S0303-7207(98)00196-8。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Subramaniam N,Treuter E,Okret S。受体相互作用蛋白RIP140通过糖皮质激素抑制阳性和阴性基因调节。 生物化学杂志。 1999; 274:18121–18127. doi:10.1074/jbc.274.25.18121。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Ghosh MG、Thompson DA、Weigel RJ、PDZK1和GREB1是激素反应性乳腺癌中表达的雌激素调节基因。 癌症研究2000; 60:6367–6375. [ 公共医学 ] [ 谷歌学者 ]
Bourdeau V、Deschenes J、Metivier R、Nagai Y、Nguyen D、Bretschneider N、Gannon F、White JH、Mader S。人类和小鼠高亲和力雌激素反应元件的基因组鉴定。 摩尔内分泌。 2004; 18:1411–1427. doi:10.1210/me.2003-0441。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Petz LN,Ziegler YS,Loven MA,Nardulli AM。雌激素受体α和激活蛋白-1介导MCF-7乳腺癌细胞中孕激素受体基因的雌激素反应。 内分泌学。 2002; 143:4583–4591. doi:10.1210/en.2002-220369。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Castro-Rivera E,Samudio I,Safe S.雌激素对ZR-75乳腺癌细胞周期蛋白D1基因表达的调节涉及多种增强因子。 生物化学杂志。 2001; 276:30853–30861. doi:10.1074/jbc。 M103339200。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Khan S、Abdelrahim M、Samudio I、Safe S.雌激素受体/Sp1复合物是乳腺癌细胞中17β-雌二醇诱导cad基因表达所必需的。 内分泌学。 2003; 144:2325–2335. doi:10.1210/en.2002-0149。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
刘德,张泽,格拉德威尔,滕康泰。雌激素通过保守的激素反应元件刺激雌激素相关受体α基因的表达。 内分泌学。 2003; 144:4894–4904. doi:10.1210/en.2003-0432。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Wang F,Samudio I,Safe S.MCF-7细胞中17β-雌二醇对大鼠肌酸激酶B的转录激活涉及雌激素反应元件和富含GC的位点。 细胞生物化学杂志。 2001; 84:156–172. doi:10.1002/jcb.1276。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Norris J、Fan D、Aleman C、Marks JR、Futreal PA、Wiseman RW、Iglehart JD、Deininger PL、McDonnell DP。 Alu DNA重复序列的一个新亚类的鉴定,该亚类可作为雌激素受体依赖性转录增强因子。 生物化学杂志。 1995; 270:22777–22782. doi:10.1074/jbc.270.39.22777。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Kerley JS、Olsen SL、Freemantle SJ、Spinella MJ。 维甲酸对核受体辅加压素RIP140的转录激活:一种潜在的负反馈调节机制。 生物化学与生物物理研究委员会。 2001; 285:969–975. doi:10.1006/bbrc.2001.5274。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Yamano G、Funahashi H、Kawanami O、Zhao LX、Ban N、Uchida Y、Morohoshi T、Ogawa J、Shioda S、Inagaki N。ABCA3是人体肺泡II型细胞中的一种板层体膜蛋白。 FEBS信函。 2001; 508:221–225。 doi:10.1016/S0014-5793(01)03056-3。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Waterston RH、Lindblad-Toh K、Birney E、Rogers J、Abril JF、Agarwal P、Agarwarla R、Ainscow R、Alexandersson M、An P等。小鼠基因组的初始测序和比较分析。 自然。 2002; 420:520–562之间。 doi:10.1038/nature01262。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
帕特·布朗实验室:实验室协议 http://cmgm.stanford.edu/pbrown/protocols/index.html
Reiner A,Yekutieli D,Benjamini Y。使用错误发现率控制程序识别差异表达基因。 生物信息学。 2003; 19:368–375. doi:10.1093/bioinformatics/btf877。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
普鲁特KD、马格洛特DR.RefSeq和LocusLink:NCBI以基因为中心的资源。 2001年《核酸研究》; 29:137–140. doi:10.1093/nar/29.1.137。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Orlando V,Strutt H,Paro R.体内甲醛交联染色质结构分析。 方法。 1997; 11:205–214. doi:10.1006/meth.1996.0407。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
关联数据 本节收集本文中包含的任何数据引用、数据可用性声明或补充材料。
补充资料
附加数据文件2 仅用ICI和CHX治疗复制1个对照微阵列研究
附加数据文件3 用单独的ICI和CHX处理复制对照微阵列研究的2
附加数据文件4 文章中描述的所有387个雌激素反应基因的完整列表,UniGene簇编号来自构建166
附加数据文件5 对照实验中确定的ICI和CHX应答基因的表达谱
附加数据文件6 对照实验中确定的ICI和CHX应答基因表达谱的对应图例