摘要 DNA甲基化和核小体定位共同产生调节基因表达的染色质结构。 核小体通常使用核酸酶消化绘制,需要大量材料和不同的酶浓度。 我们已经开发了一种方法(NOMe-seq),该方法使用GpC甲基转移酶(M.CviPI)和下一代测序,使用不到100万个细胞生成核小体定位全基因组的高分辨率足迹,同时保留来自同一DNA链的内源性DNA甲基化信息。 通过使用一种新的生物信息学管道,我们发现在启动子不存在的CTCF区域,核小体占有率和DNA甲基化之间存在显著的反相关。 我们进一步表明,启动子处核小体耗竭的程度与表达水平直接相关,可以容纳多个核小体,并提供全基因组证据证明表达的非CpG岛启动子是核小体缺失的。 重要的是,NOMe-seq从同一DNA分子中获得DNA甲基化和核小体定位信息,从而在单分子和单细胞水平上首次实现全基因组DNA甲基化与核小体位置相关性,可用于监测疾病进展和治疗反应。
包括DNA甲基化和核小体定位在内的表观遗传机制共同作用,产生促进、抑制或允许基因表达潜力的特定染色质状态。 活性启动子具有非甲基化DNA,并且在基因转录起始位点(TSS)之前缺少核小体,而非活性启动子则具有密集的核小体并且可以是非甲基化(平衡或抑制)或甲基化(沉默)。 由于染色质结构的多样性,仅通过核小体占有率或DNA甲基化无法预测基因激活潜力。
迈克尔·克拉德(Michael Kladde)及其同事的开创性工作证明了基于甲基转移酶的足迹分析在酵母和哺乳动物细胞中确定核小体定位的能力( Xu等人,1998年 ; Jessen等人2004 , 2006 ; Kilgore等人,2007年 ; Pardo等人,2010年 ). 使用下一代测序,我们描述了一种称为NOMe-seq(核小体占有率和甲基组测序)的全基因组核小体足迹方法,该方法使用GpC甲基转移酶(M.CviPI)( Xu等人,1998年 )根据GpC位点的酶可及性获得核小体定位信息,同时从CpG位点获得内源性DNA甲基化信息。 重要的是,这两条表观遗传信息都是从同一个单独的DNA分子中获得的,揭示了单个染色体上这两个染色质特征之间的关系。 因此,使用单一方法,可以在单个分子水平上生成多个表观遗传修饰的全基因组图。
使用NOMe-seq和全基因组亚硫酸氢钠测序,我们生成了一个完整的图谱,并显示了与特定基因组特征相关的不同核小体/甲基化配置,TSS上游NDR的强度指示了表达水平,可以容纳几个核小体。 通过检测来自两种不同染色质状态(核小体占有率和甲基化定义)的启动子,我们确定了可能处于两种不同等位基因状态的基因,这些基因在X染色体和已知印迹位点上高度富集。 同时测量单个DNA链中的核小体占有率和DNA甲基化是一个重要的工具,可以用来检查在疾病状态下基因组中的染色质结构是如何改变的。
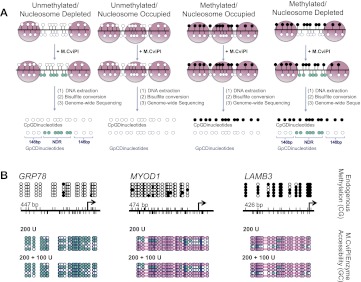
结果 确定各种基因组基因座精确足迹的最佳处理条件 为了生成完整的DNA甲基化和核小体占有信息,用M.CviPI处理细胞核,使不受核小体或紧密结合蛋白保护的GpC二核苷酸甲基化。 继亚硫酸氢盐转化以区分甲基化和非甲基化胞嘧啶残基之后,CpG二核苷酸上下文中包含的胞嘧啶提供内源性甲基化信息,而核小体定位则源自GpC二核苷酸中的胞嘧啶。 当每个单独胞嘧啶的甲基化被计算为甲基化读数除以覆盖该位置的所有读数的分数时,可获得核小体占有率和内源性DNA甲基化信息。 结合CpG和GpC甲基化谱,可以看到四种不同的染色质结构( 图1A ). 我们首先确定了一组反应条件,允许各种染色质构型的精确足迹(即核小体亏损区域的可接近性,而不异常地进入核小体占用区域,定义为146 bp或更大,M.CviPI无法访问)( 图1B ; 补充图S1A)。
图1。
NOMe-seq可以覆盖多种染色质结构。 ( A类 )用M.CviPI处理IMR90细胞核后,提取DNA,进行亚硫酸氢转化,并进行测序。 DNA甲基化状态由CpG二核苷酸获得,核小体占有信息由M.CviPI甲基转移酶无法进入GpC二核苷酸获得。 DNA甲基化和核小体占有率数据的结合可以揭示四种不同的染色质特征:非甲基化和核小体缺失、非甲基化与核小体占用、甲基化与核小体占用以及甲基化和细胞核缺失。 (黑圈)甲基化CpG位点; (茶色圆圈)可进入(甲基化)GpC位点。 ( B )我们发现,200单位的M.CviPI持续7.5分钟,随后增加100单位,准确显示TSS上游的NDR为 热休克蛋白5 (也称为 GRP78级 ),一个活性CGI启动子,同时也显示多梳抑制 MYOD1年 CGI启动子和甲基化修饰的CpG-poor 灯3 正如预期的那样,启动子被核小体占据,M.CviPI无法访问。 大于146 bp的M.CviPI不可及区域被一个粉红色矩形覆盖,表示核小体占据。 PCR扩增子大小: 热休克蛋白5 –447个基点, MYOD公司 –474个基点,以及 灯3 –426个基点。
NOMe-seq揭示了CTCF和转录起始位点的预期核小体占据模式 我们生成了全基因组NOMe-seq文库,并调整了我们的全基因组亚硫酸氢处理管道( Berman等人,2012年 ; Liu等人,2012年 ; 补充材料),以分离基于中央位置含有胞嘧啶的三核苷酸的胞嘧啶。 GCH胞嘧啶通常用于绘制酶可及性(核小体保护或占有),而HCG(其中H=C、T或A)用于内源性甲基化。 由于内源性甲基化和酶甲基化之间的模糊性,GCG被排除在外。 排除GCG不太可能显著损害M.CviPI定位核小体的能力,因为GCG不到基因组的0.24%,仅占所有GC二核苷酸的5.6%(补充表S1)。 此外,93.4%的GCG三核苷酸的GCH在20 bp以内(其中一半在5 bp以内),可以从中获得核小体占有信息(补充图S1B)。
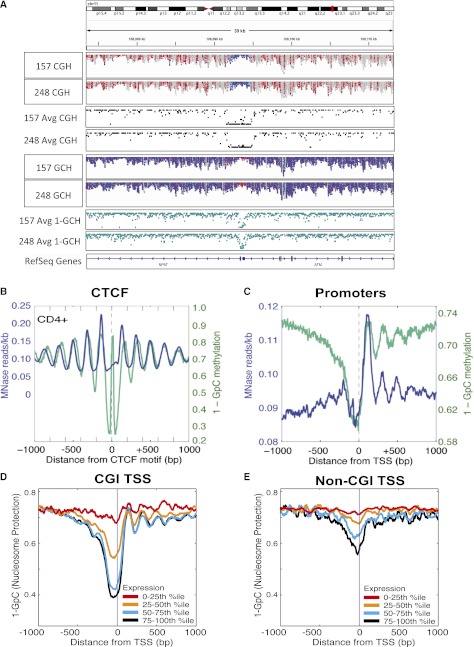
由于全基因组数据的可用性( 李斯特等人,2009年 ; Bernstein等人,2010年 ),我们在IMR90细胞中执行了全基因组NOMe-seq,获得了1.56亿个可唯一对齐的读取,这些读取可以使用新开发的IGV查看器模块从原始BAM对齐文件中显示( 图2A ; 补充图S2; Thorvaldsdottir等人,2012年 ). 我们比较了NOMe-seq准确定位位于CTCF结合位点两侧的核小体的能力。 我们将读取与保守的CTCF结合基序对齐( Xie等人,2007年 )位于距离TSS 2 kb以上的位置,这些TSS已被CTCF实验验证为体内结合(补充表S2; Kim等人,2007年 ; Cuddapah等人,2009年 )并发现NOMe-seq映射的核小体与MNase-seq数据相似( 图2B ; Schones等人,2008年 ). 核小体占有率被绘制为不可接近M.CviPI(1-GpC甲基化)( 图2B ,蓝绿色线条); 因此,保护区域在图中显示为峰值。 与MNase相比,使用NOMe-seq时,CTCF结合位点右侧的第一和第二核小体似乎稍微不同步; 然而,这里我们比较的是来自基准CD4+T细胞数据集的MNase-seq数据( Schones等人,2008年 )在IMR90单元中收集NOMe-seq数据。 当我们比较从IMR90细胞生成的NOMe-seq和MNase-seq数据时,这种相移并不明显(补充图S3)。 NOMe-seq产生的高分辨率还揭示了与CTCF结合位点一致的保护区域,可能反映了CTCF的结合。 为了进一步研究这一点,我们以高分辨率检测了CTCF结合区(补充图S4),并显示出与CTCF基序重叠的谨慎保护模式,其大小(<40bp)与非核小体蛋白一致。 这个保护区被清晰的核小体耗竭(可及性)所包围,而这反过来又被更大的保护区所包围,其大小与核小体占有率一致。 接下来,我们将NOMe-seq读数与所有TSS进行比对,再次发现NOMe-se q与MNase-seq相当,能够识别TSS上游的核小体缺失区域(NDR)和TSS下游位置良好的核小体( 图2C ).
图2。
NOMe-seq显示了特定位点和全球的核小体占有情况。 ( A类 )使用新开发的IGV查看器模块对ATM启动程序进行全景显示( Thorvaldsdottir等人,2012年 )以可视化NOMe-seq BAM对齐文件。 这个 顶部 两个轨道表示两个GBM样本中每个样本的内源性DNA甲基化(在HCG位点),而轨道5和6表示相同GBM样本的GCH可及性。 (红色)甲基化位点(HCG和GCH); (蓝色)非甲基化位点(HCG和GCH)。 ATM和NFAT的启动子未甲基化(蓝色 顶部 两个轨道)和核小体缺失(即,可接近并因此甲基化,因此在轨道5和6中为红色)。 两个GBM样本的甲基化和核小体占用模式相同。 轨迹3和轨迹4显示了从这些轨迹导出的平均甲基化水平-在每个HCG中,该HCG甲基化的读取数除以甲基化和非甲基化的总读取数。 与之前一样计算轨道7和轨道8中的平均GCH甲基化,但将其倒置(1-GCH),以表示整个主要数字中使用的核小体保护。 该工具和源代码可在IGV项目网站上公开下载: http://www.broadinstitute.org/igv/ . ( B、 C类 )NOMe-seq读数与CTCF一致( B )和TSS( C类 ). IMR90细胞中的核小体定位显示在 年 -通过对M.CviPI的不可接近性(1-GpC甲基化;蓝线)和MNase测序读数的数量(蓝线)确定轴。 对于MNase-seq,读取与8709个CTCF站点一致,而8687个CTCF-站点至少有一个GpC站点被至少三个读取覆盖( B ). 对于TSS,42103个启动子用于MNase-seq,41292个启动子至少有一个GpC站点被至少三次读取覆盖。 ( D、 E类 )基因启动子根据转录水平分为四分位( Hawkins等人,2010年 )和相应的M.CviPI 在里面 可达性(1-GCH,蓝绿色线)绘制在 年 -轴。 ( D类 )CpG岛促进者。 ( E类 )非CpG岛促进者。 NDR在更高表达的基因中更强,在某些情况下,可以长达数百bp以容纳多个核小体。
接下来,我们通过根据表达水平将启动子划分为四分位来研究核小体耗竭与表达之间的关系( Hawkins等人,2010年 )并且发现,无论是CGI还是非CGI启动子,位于最低位置(0%–25%)的启动子都被核小体占据( 图2D,E ). 随着表达四分位数的增加,TSS上游的NDR和TSS后核小体的定位对于CGI和非CGI启动子都变得更加明显。 这些结果表明,TSS上游的NDR和TSS下游的定位核小体强烈预测表达水平,并表明CGI和非CGI启动子的表观遗传调控相似。
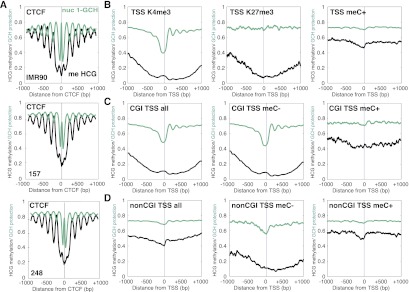
NOMe-seq揭示了特定启动子类型的不同染色质构型 我们检测了CTCF位点的核小体占有率和甲基化模式,以及特定启动子分类( 图3 ; Hawkins等人,2010年 ),以及其他基因组区域,包括增强子和内含子/外显子边界(补充图S6、S7)。 有趣的是,DNA甲基化和核小体占有率与CTCF位点周围强烈反相关,以至于DNA甲基化在核小体之间的连接区达到峰值( 图3A ; 补充图S5)。 为了检测这种相关性是否是细胞类型特异性的,我们在胶质母细胞瘤肿瘤(157和248)的两种原代培养物中进行了NOMe-seq,发现这些细胞中的CTCF位点的DNA甲基化和核小体定位也不相关( 图3A ). 在启动子处,核小体占有率和DNA甲基化与转录潜能一致( 图3B ):H3K4me3标记(活性)启动子未甲基化,TSS上游有一个明显的NDR,TSS下游至少有四个核小体,而H3K27me3标记的(抑制)启动子没有甲基化,但核小体被占据,如M.CviPI不可接近所示。 DNA甲基化(沉默)启动子完全被核小体占据。 NOMe-seq能够在单个实验中区分这三种重要且不同的启动子结构。 令人惊讶的是,在H3K4me3标记的启动子的TSS上游,DNA甲基化出现“突起”,我们发现这是由于M.CviPI的“非靶向”活性,并且只影响从胞嘧啶获得的内源性甲基化信息,而胞嘧啶之前的另一个胞嘧啶位于M.CviPI-可及性峰值区域。 通过消除CCG(补充图S8;补充材料)可以完全消除该伪影,未来的分析方法可以更好地调整已知的离目标活动率。
图3。
NOMe-seq揭示了CTCF位点上不同的染色质配置,并与特定组蛋白修饰和启动子类型相关。 ( A类 )NOMe-seq在IMR90和GBM细胞中的CTCF位点显示出非甲基化NDR,其特征是CTCF部位本身的不可接近性达到峰值。 定位良好的核小体位于CTCF位点两侧,DNA甲基化在核小体之间达到峰值。 0表示CTCF结合基序的中间。 CTCF结合位点来自 GSM935404标准 . ( B )NOMe-seq区分了IMR90细胞启动子处的三种主要启动子状态——活性H3K4me3标记的启动子未甲基化,包含NDR上游和TSS后位置良好的核小体。 TSS显示在 x个 -轴设置为0。 抑制/平衡H3K27me3标记的启动子未甲基化,核小体被占据。 甲基化启动子被核小体占据。 这个 年 -轴表示M.CviPI不可接近性(1-CpG;teal)和CpG甲基化水平。 ( C类 )在IMR90细胞中,CpG岛启动子的特征是缺乏CpG甲基化、上游NDR和TSS后定位良好的核小体。 大多数CpG岛启动子是非甲基化的(11165),并显示相同的模式,而甲基化的CpG岛屿启动子(781)是核小体占据的,M.CviPI无法访问。 ( D类 )非CpG岛启动子通常以CpG甲基化和不可接近M.CviPI为特征,表明核小体占据。 少数非甲基化非CpG岛启动子(1397)在TSS上游的核小体耗尽,而大多数非CpG-岛启动子是核小体占用的,M.CviPI无法访问。 在teal中绘制M.CviPI不可接近性(1-GCH),在黑色中绘制CpG甲基化(CGH)。
接下来我们研究了CGI和非CGI启动子的染色质构型( 图3C、D ; 补充图S7A,B)。 总的来说,CGI启动子在TSS附近的胞嘧啶甲基化水平较低(相对于离TSS 1 kb),TSS上游的NDR明显,TSS下游的核小体位置良好。 将CGI启动子分离为甲基化和非甲基化的启动子表明,CGI启动程序模式主要由非甲基化CGI启动子驱动,少数甲基化的CGI启动因子被核小体占据。 将非CGI启动子分离为甲基化和非甲基化的启动子表明,相对较少的未甲基化的非CGI发起子在TSS上游也有NDR,在TSS下游也有核小体,而更常见的甲基化非CGI的启动子被核小体占据。
为了证明NOMe-seq的重复性,我们对两种胶质母细胞瘤(GBM)原代细胞培养物进行了测序,发现GBM细胞中启动子和增强子的核小体定位模式与IMR90细胞相似(补充图S7)。 使用统计测试来识别TSS附近的NDR(参见方法),我们发现CGI启动子的所有样本之间高度一致; 两个GBM的NDR彼此重叠90%,分别与IMR90重叠88%和91%(补充图S7C)。 许多对细胞功能至关重要的基因(即管家基因)具有CGI启动子; 因此,GBM和IMR90细胞之间如此显著的重叠并不奇怪。 然而,在两个GBM中偶然获得90%的NDR重叠的概率是10 −518 使用超几何测试。 我们发现非CGI启动子在细胞类型之间的重叠显著减少,这与非CGI基因的更大细胞类型特异性一致。 在这些基因启动子中,两个GBM样本重叠了58%,而它们分别重叠了43%和47%的IMR90。
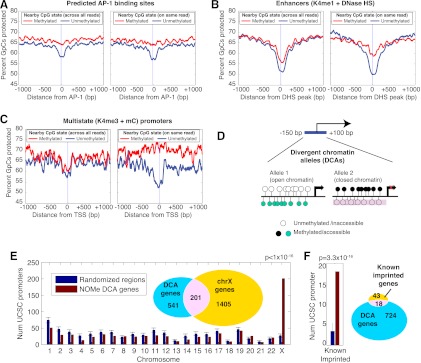
组合表观基因组特征揭示功能染色质 与用于评估核小体占有率或DNA甲基化的任何其他方法不同,NOMe-seq包括单个DNA链的核小体定位和DNA甲基化数据,从而在单分子水平上实现这两个特征之间的关联。 由于单个细胞或样本中不同细胞亚群的两个等位基因上可能存在不同的染色质状态,因此我们预计单个分子上的两个标记组合将产生比细胞群平均水平更多的信息。 为了研究这一点,我们计算了作为DNA甲基化状态函数的基因组元件周围的核小体保护模式,首先使用基因组中相同位置的任何读数的群体平均值中的甲基化信息( 图4A-C ,左面板),然后仅使用相同读取的甲基化状态( 图4A-C ,右侧面板)。 我们研究的一些调控元件,例如带有预测AP-1结合基序的序列,具有可见的NDR,但在群体平均值之间几乎没有差异( 图4A ,左)和读取中的平均值( 图4A ,右侧)。 这些元素表明整个细胞群体中特定染色质状态的一致性。 我们研究的其他元素,例如在IMR90细胞中注释为DNase超敏增强子的元素( Hawkins等人,2010年 ),在单个读数中的DNA甲基化和可及性之间的相关性比在整个读数群体中的相关性更强,这表明在样本中的细胞或等位基因的子集中存在组合染色质特征( 图4B ).
图4。
组合表观基因组特征揭示了功能染色质。 ( A–C )核小体占用水平(“GpCs保护百分比”)由附近CpGs的甲基化状态分层显示(20 bp以内)。 对于每种元素类型,该分析在覆盖与GpC相同基因组位置的所有读数中随机进行两次采样( 左边 标记为“跨所有读取”的图),第二次仅使用相同读取的甲基化状态( 正确的 图,标记为“同一读数”)(参见方法)。 所有三个例子都表明核小体耗竭主要与非甲基化状态有关,但同时预测了AP-1结合基序( A类 )在以相反的K4me3和meC标记的群体和内部阅读资料、增强子和启动子中显示这一点( B、 C类 )仅在内部读取分析中显示此关联。 0表示AP-1结合基序的中心( A类 ),在K4me1标记区域内的DNase HS的峰值( B )和TSS( C类 ). ( D类 )通过在IMR90细胞中搜索TSS区域至少两个具有相反染色质分布的读取来查找不同染色质等位基因(DCA)的搜索策略。 ( E类 )核小体缺失和非甲基化以及核小体占据和甲基化中存在的启动子在X染色体上富集。 742个DCA基因与742个随机基因组进行了比较,共进行了1000次试验,并显示了每条染色体上的数量的标准偏差。 A类 P(P) -该值由X染色体通过二项检验确定,概率由随机试验确定。 ( F类 )将DCA基因与1000个随机基因集进行比较,以确定50kb内已知印迹基因的数量。
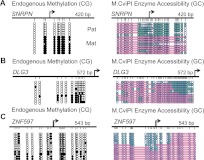
为了研究我们是否可以在可能为单等位基因的区域内检测到组合染色质特征,我们对IMR90细胞中同时具有DNA甲基化和H3K4me3标记的基因启动子应用了相同的方法( 图4C ; Hawkins等人,2010年 ). 这两种状态在启动子上通常是拮抗的,这表明它们可能存在于同一细胞中的两个不同等位基因上,特别是在IMR90这样的遗传雌性细胞系中。 交叉阅读与内阅读的比较表明,当所有阅读平均时,甲基化和核小体占有率之间的任何相关性都会丢失( 图4C ,左),但在查看内部读取相关性时很清楚( 图4C ,右侧)。 为了测试我们是否可以利用这种内读相关性来识别等位基因特异区域,我们搜索了所有启动子(TSS的−150到+100 bp),以寻找两种相反的染色质构象的组合( 图4D )一种含有非甲基化CpG,无核小体保护(等位基因1),另一种含有甲基化Cp G和核小体防护(等位蛋白2)。 我们发现742个启动子区域符合“差异染色质等位基因”(DCA)标准,其中201个定位于X染色体(27%的DCA启动子,而2.7%的全基因组启动子)( 图4E ). 18个DCA启动子与58个已知印迹基因中的一个相关( 网址:http://www.geneimprint.org/ )相比之下,随机选择的启动子组中平均有四个启动子( 图4F ). 为了验证我们的全基因组研究结果,我们对一个X-连锁基因进行了定位NOMe-seq分析( DLG3(数据链路3) ),一个印记基因( SRNPN公司 )和一个新鉴定的DCA启动子( ZNF597型 ),最近有人建议将其压印( 图5 ; Choufani等人,2011年 ; Nakabayashi等人,2011年 ). 我们的结果清楚地表明存在两种不同的染色质结构。 我们进一步显示,与GBM和IMR90样本中共享的DCA等位基因数量相比,两个GBM样本之间的DCA等位基因重叠更多(补充图S9)。 将来自单个DNA链的DNA甲基化和核小体定位信息结合起来,可以识别几个以前没有描述过的单等位基因,我们预计增加测序深度将大大提高我们对这些区域的敏感性。
图5。
DCA启动子的验证。 PCR扩增子被克隆,几个克隆被测序,以显示印迹基因的两种不同染色质构型, SNRPN公司 ( A类 )和一个X连锁位点, DLG3(数据链路3) ( B )和一个新鉴定的DCA基因, ZNF597型 ( C类 ). CpG位点的(黑色)DNA甲基化; (teal)GpC可访问性。 粉色条表示核小体的位置。
讨论 使用一种新的方法来检测核小体占有率和全基因组内源性DNA甲基化,我们在各种启动子和非启动子区域足迹染色质结构。 我们发现TSS上游的NDR可以容纳多个核小体,并指示CGI和非CGI启动子的表达水平。 我们进一步表明,核小体占有率和DNA甲基化之间的关系取决于基因组位置,并且DNA甲基化和核小体信息在同一DNA链中的结合可以促进单等位染色质模式的鉴定。 重要的是,我们表明NOMe-seq可以区分启动子激活(H3K4me3,meC-,NDR+)、抑制/平衡(H3K27me3,meC-,NDR-)和沉默(meC+,NDR-)中发现的三种主要染色质状态。 在单个实验中区分这三种启动子结构的能力,更不用说单个分子了,为表观基因组作图提供了巨大的希望。
传统上,核小体定位的全基因组作图是使用MNase-seq或H3-ChIP-seq完成的,这依赖于DNA断裂。 FAIRE-seq依赖于核小体缺失区对DNA断裂的敏感性增强( Giresi等人,2007年 ; Nagy和Price 2009 ). 而不是使用核酸酶,甲基转移酶为基础的足迹,与CpG甲基转移酶M.SssI( Gal-Yam等人,2006年 ; Lin等人,2007年 ; Bouazoune等人,2009年 ; Kelly等人,2010年 )或GpC甲基转移酶M.CviPI( Kelly等人,2010年 ; Wolff等人,2010年 ; Andreu Vieyra等人,2011年 ; Taberlay等人,2011年 ; You等人,2011年 ),使用DNA上的生化标记(即甲基化)来评估核小体的占有率。 由于GpC二核苷酸不是内源性甲基化的,NOMe-seq在同一条DNA链中提供核小体定位和DNA甲基化。 此外,由于NOMe-seq信号被解释为 百分比 对于给定位置的测序读数,它提供了一种标准化和无扭曲的测量,不依赖于映射到特定基因组位点的读数数量,因此是一种评估核小体占有率的独立方法,可以补充和验证已建立的富集方法的结果。
利用MNase-seq和来自亚硫酸氢盐测序文库的DNA甲基化信息,先前的工作发现核小体DNA优先甲基化( Chodavarapu等人,2010年 ). 虽然这对大多数基因组来说都是事实,但特定类型的元素并不符合全基因组的趋势。 例如,CTCF位点周围的核小体未甲基化,而核小体之间的连接区甲基化,这表明了一类功能重要的染色质之间的新关系。
活性基因的一个特征是TSS上游存在NDR。 先前的工作发现活性组蛋白标记的水平与表达水平相关( Barski等人,2007年 ); 然而,核小体占有率本身并没有被测量,TSS上游区域的核小体似乎同样耗尽,无论转录水平如何。 在这里,我们表明NDR在更高表达的基因中更为显著。 重要的是,CGI和非CGI启动子的表达和核小体耗竭之间的这种相关性是相似的,这表明至少表观遗传基因调控的一些关键方面在CGI和非CGI启动因子之间是共享的。 此外,我们的结果表明NDR足够大,可以容纳多个核小体。 以前的研究无法根据表达检测出细微的核小体耗竭差异,并且低估了NDR大小,这可能反映了超声波作用产生的碎片大小的变化, 并强调了染色质组织的微妙之处,这些细微之处可以通过NOMe-seq识别,但在以前的研究中被忽视了。
全基因组NOMe-seq是一种新的方法,它可以追踪核小体的占有率,同时保留DNA甲基化信息,以识别包括启动子、增强子和绝缘体在内的多种基因组区域的染色质结构。 将这两个表观遗传标记结合在同一分子上,可以识别混合细胞群或等位基因中的组合特征,其敏感性高于单独使用的两个标记。 由这些组合表观基因组图谱生成的表观遗传景观对生物学具有若干重要意义,特别是在分析包含多种细胞类型的复杂组织时。 此外,随着染色质重塑复合物突变与癌症的关系越来越密切( 威尔逊和罗伯茨2011 )全基因组NOMe-seq是解决这些突变对核小体位置和DNA甲基化的影响的理想方法,可以进一步研究染色质重塑缺陷是否依赖于DNA甲基化状态。
方法 细胞培养 根据ATCC建议培养IMR90细胞。 如前所述培养原代GBM细胞( Laks等人,2009年 ). 简言之,神经球培养基中含有补充B27(GIBCO)、bFGF(20 ng/mL,R&D Systems Inc.)、表皮生长因子(EGF;50 ng/mL、Peprotech)、青霉素/链霉素(1%,Invitrogen)和肝素(5μg/mL,Sigma-Aldrich)的DMEM/F12。 每隔3或4天向培养基中添加肝素、bFGF和EGF。在与TrypLE Express(Invitrogen)分离后,每隔7至14天对球体进行传代。
核小体足迹 NOMe-seq是我们甲基化依赖性单启动子分析的改进版( Miranda等人,2010年 ). 如前所述,从IMR90细胞(ATCC)中分离细胞核( Miranda等人,2010年 ). 以前使用特定于本地的NOMe-seq的出版物使用了最小数量的M.CviPI,从而实现了特定关注区域的最佳足迹:100个单位( Wolff等人,2010年 ),200个单位( Taberlay等人,2011年 ; You等人,2011年 ),或200+100个单位( Andreu-Vieyra等人,2011年 ). 由于全基因组NOMe-seq需要各种基因组区域的精确足迹,因此我们进行了剂量-反应曲线( 图1 ; 补充图S1); 将细胞核与100或200单位的GpC甲基转移酶(M.CviPI)一起孵育 S公司 -腺苷蛋氨酸(SAM)在37°C下15分钟,或200单位GpC甲基转移酶(M.CviPI)和SAM在37°C下7.5分钟,然后再增加100单位M.CviP1和SAM 7.5分钟。对于全基因组NOMe-seq,文库是从与200单位Gp C甲基转移物(M.CviPI)孵育的细胞核中生成的 和SAM在37°C下持续7.5分钟,然后增加100个单位的M.CviPI和SAM,持续7.5分钟。停止反应,提取DNA并转化亚硫酸氢,以区分甲基化和非甲基化Cs。 对于感兴趣的单个区域,使用不包含任何CpG或GpC二核苷酸的PCR引物进行PCR,然后进行TA克隆和测序。 PCR引物序列可根据要求提供。
图书馆建设和排序 对于NOMe-seq,如前所述,从5 ug DNA制备文库( 李斯特等人,2009年 ; Kelly等人,2010年 ; Berman等人,2012年 ). 简言之,M.CviPI处理的DNA被片段化为~200-bp片段、END-重配对(Epicenter)、甲基化适配器连接(Illumina)、亚硫酸氢盐转化(Zymo-EZ DNA甲基化),并进行PCR。 根据Illumina协议生成簇,并使用76-bp单端配置在Illuminia Hi-seq 2000上对产生的库进行测序。 除了使用Illumina Hi-seq 2000 Paired-End协议进行测序外,每个胶质母细胞瘤样本都使用相同的方法进行测序。 基本调用由Illumina Real Time Analysis(RTA)软件执行,共产生118万次读取,通过了Illumiana质量过滤器(IMR90)。 GBM培养#157用一条50-bp配对-end(3.1亿读)和一条100-bp配对-end(2.91亿读)的通道进行测序,而培养#248用一条50 bp配对-end的通道(3.13亿读)以及一条100-bp配对-erd的通道(3.01亿读数)进行测序(补充表S4)。
CG和GC甲基化水平的序列比对和提取 基因组比对和亚硫酸氢盐序列分析基本上如前所述( Berman等人,2012年 ),对配对测序进行了一些调整。 对于单端IMR90库,MAQ( Li等人,2008年 )与“-c”亚硫酸氢盐模式一起使用(如 Berman等人,2012年 ),对于配对的GBM库,BSMAP( Xi和Li 2009 )已使用。 IMR90读数与NCBI参考基因组hg18对齐,GBM序列与hg19对齐。 筛选出绘图质量低于30的基因组比对,结果是6.78亿次读取(IMR90)、5.87亿次读取量(GBM#157)和6.91亿次读取数(GBM#248)。 对于IMR90和GBM细胞,我们删除了从与另一个读取完全相同的基因组位置开始的读取(PCR“重复”读取),得出IMR90的总计1.56亿个可分析读取(11.8千兆字节)。 对于GBM paired-en,我们还删除了未“正确配对”的读取(映射到彼此相距500 bp内的相反链),GBM#157的可分析读取总数为4.62亿次(34.0千兆字节),GBM#248的可分析读数为4.92亿次,36.4千兆字节。
在亚硫酸氢盐测序数据中很难或不可能区分C到T单核苷酸多态性,但我们的Illumina协议仅从两条链中的一条恢复亚硫酸氢数据(无论互补胞嘧啶是否甲基化,与胞嘧啶互补的G残基都被读作G)。 对于这种“定向”亚硫酸氢盐文库协议,胞嘧啶位置在序列上显示为C或T,这取决于亚硫酸氢转换,而C对侧链上的互补G仅显示为G( Krueger等人,2012年 ). 因此,我们指的是相对于给定胞嘧啶位置的两条链——“亚硫酸氢盐-C链”(BCS)和“基因型G链”(GGS)。 基因型G链因此被命名,因为它揭示了该位置的真实基因型,不受亚硫酸氢盐转化的影响。 由于Hi-Seq配对序列的特殊性,配对序列的第二个末端始终是BCS序列的反向补码,必须在分析之前进行反向补码才能获得真正的BCS序列。
如果至少90%映射到BSC链的读码为C或T,我们只包括参考基因组中存在的胞嘧啶,这至少包括三个读码。 此外,我们只包括90%映射到GGS的读取为G的胞嘧啶(任何其他碱基都表示遗传变异;重要的是,只有GGS链才能揭示可能导致错误甲基化调用的C>T转换)。 使用相同的标准确定胞嘧啶位于特定的XCX三核苷酸上下文中,例如,仅当90%的读取为g(前一个碱基)和90%的读取是A、C或T(IUPAC“H”符号包括A、C、T)时,才包括GCH位置。 BCS链上的读取按上述方式处理,即C或T可以匹配“X”上下文位置中的C。 该方法用于确定本研究中讨论的以下三核苷酸:HCG(H包括A、C或T)、GCG、WCG(W包括A或T)和GCH。
如中所示 Berman等人(2012) ,我们过滤出具有明显亚硫酸氢不转换的读取的5′端,这在Illumina协议中很常见,可能是因为甲基化的适配器序列附近的碱基对重新退火,因此具有100%的碱基互补性( Hansen等人,2011年 ; Berman等人,2012年 ). 我们通过从测序读数的5′处向内走,忽略任何未转换的胞嘧啶(在任何序列上下文中),直到遇到第一个转换的胞苷,从而实现这一点。 从这一点和读数中的所有3′位置,我们将所有转化和未转化的胞嘧啶纳入甲基化计数。
对于所有下游分析,我们包括CCG三核苷酸,尽管描述的M.CviPI活性轻微偏离目标,仅影响CG甲基化信息。 因此,甲基化平均值包括所有HCG三核苷酸。唯一的例外是内读组合图( 图4 ),其中被平均的大量数据点允许我们排除CCG并仅使用WCG三核苷酸(W:A,T)。
启动子核小体缺失区检测 启动子核小体缺失区域的鉴定(补充图S7)如下:独立考虑UCSC KnownGenes轨迹中的每个独特TSS。 收集与候选NDR区域(-100到+50 bp)重叠的所有测序读数,并对每次读数进行GCH分析。 每个重叠读数上的每一个GCH都被视为一个独立的核小体保护测量值,只有那些基础质量分数大于10的人被包括在内。 那些具有10个或更少此类数据点的TSS从分析中删除,作为序列覆盖不足的区域。 该覆盖过滤器分别去除了IMR90中41054个hg18 TSS中的27312个(66%),以及GBM培养物#157和#248中41017个hg19 TSS中6225个(15%)和4009个(10%)。 对于每个样本,将候选NDR区域内这些独立GCH测量值的甲基化频率与候选-100至+50 bp区域上游4 kb和下游4 kb附近的频率进行比较。 我们使用单尾二项检验来测试候选NDR区域中GCH甲基化的频率是否为 较高的 (即核小体保护更少)。 二项式检验结果为 P(P) -值,分别针对每个样本中的多个假设(Benjamini-Hochberg)进行校正,使用通过该特定样本中初始覆盖过滤器的TSS数量作为假设数量。 所有TSS、候选NDR和周围区域的甲基化频率以及原始和校正的列表 P(P) -每个样品的数值见补充表S5-S7。
来自三个样本的NDR调用与组蛋白标记(补充图S7中的维恩图)之间的交集生成如下:给定维恩图中考虑的TSS“范围”仅包括那些通过交集中包含的所有样本的覆盖过滤器的TSS,即补充图S7C, 仅包括所有三种细胞类型所涵盖的12424个TSS,而在补充图S7D中,仅包括两个GBM样本所涵盖的33425个TSS; 我们考虑了该子集中所有组蛋白标记的TSS。
组合表观基因特征 按DNA甲基化状态分层的核小体保护比较(来自GCH)( 图4 )如下所示:对TSS(或所列其他元素)1 kb范围内参考基因组中的每个GCH进行独立评估。 对于每个这样的基因组位置,分析每个亚硫酸氢盐C链的读映射与“附近”CG的读关联。 上游或下游20 bp范围内的每个WCG被视为“附近”(选择为可以解决核小体定位的距离)。 如果附近的WCG被甲基化,则读取的GCH甲基化值被分层到“甲基化”区( 图4 ); 同样,附近CG未甲基化的读数也进入了未甲基化垃圾桶( 图4 ,蓝线)。 如果单个GCH在多个CG的20 bp范围内,则每次读取的多个CGs中的每个CG的甲基化值作为独立观察值进入适当的(甲基化或非甲基化)仓位。
要在右侧绘图中生成绘图 图4A (标记为“同读”),这些甲基化和非甲基化GCH基因座在所有基因组元件中平均,得出甲基化(红色)和非甲基(蓝色)基因座的两个平均GC图谱。 对于左侧绘图(标记为“所有读取”),除了“附近”的CG甲基化值是从随机选择的读取映射到同一位置,而不是与GC相同的读取之外,整个分析都以相同的方式进行。 通常,多个读数重叠在同一位置,但我们只随机选择一个读数,以保持观测值的数量与“读数相同”的条件相同,从而消除了两种条件之间方差差异的任何可能影响。
发散染色质等位基因启动子检测 带有“发散染色质等位基因”启动子的鉴定( 图4 ; 补充图S9)的执行如下:我们只统计了含有两个或更多GCH和两个或多个HCG的读取,每个类别中90%的胞嘧啶是一致的。 对于UCSC KnownGenes轨迹中的每个TSS,我们选择了一半以上读数在(−150到+100 bp)范围内的读数。 任何至少有一个读取处于“活性”染色质结合状态(CG非甲基化和GC核小体可访问)和另一个读取位于“沉默”状态(CG-甲基化和GC-核小体保护)的基因都被视为DCA基因。 将这些落在X染色体上或与印记基因相关的部分与从基因组中随机选取的大小匹配的集合进行比较,如 图4 传奇。
致谢 我们感谢Charlie Nicolet、Selene Tyndale和Helen Truong在生成测序数据方面的支持,感谢Jones实验室和南加州大学表观基因组中心的成员进行了有益的讨论。 GBM细胞是加州大学洛杉矶分校人类细胞核心智力和发育障碍研究中心的Harley Kornblum博士慷慨捐赠的。 我们还感谢Alvaro Rada-Iglesias和Joanna Wysocka提供NEC ChIP测序数据。 这项工作由NCI 5R37CA082422-13向P.A.J.、T32-CA009320-77和1K99CA160349-01向T.K.K.、T32向F.D.L.提供CBM培训拨款资助,Kenneth T.和Eileen L.Norris基金会向Y.L.和B.P.B.提供慷慨支持。计算在南加州大学高性能计算和通信中心进行( http://www.usc.edu/hpcc/ ).
工具书类
Andreu-Vieyra C、Lai J、Berman BP、Frenkel B、Jia L、Jones PA、Coetzee GA 2011 前列腺癌细胞中缺乏配体时雄激素受体增强子的动态核小体缺失区。 分子细胞生物学 31: 4648–4662 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Barski A、Cuddapah S、Cui K、Roh TY、Schones DE、Wang Z、Wei G、Chepelev I、Zhao K 2007 人类基因组组蛋白甲基化的高分辨率分析。 单元格 129: 823–837 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Berman BP、Weisenberger DJ、Aman JF、Hinoue T、Ramjan Z、Liu Y、Noushmehr H、Lange CP、van Dijk CM、Tollenaar RA等。 2012 结直肠癌局部DNA高甲基化和长程低甲基化区域与核片相关结构域一致。 自然基因 44: 40–46 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Bernstein BE、Stamatoyannopoulos JA、Costello JF、Ren B、Milosavljevic A、Meissner A、Kellis M、Marra MA、Beaudet AL、Ecker JR等。 2010 美国国立卫生研究院路线图表观基因组学测绘联盟。 天然生物技术 28: 1045–1048 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Bouazoune K、Miranda TB、Jones PA、Kingston RE 2009 对单个重塑核小体的分析显示hSWI/SNF导致组蛋白-DNA接触减少。 核酸研究 37: 5279–5294 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Chodavarapu RK、Feng S、Bernatavichute YV、Chen PY、Stroud H、Yu Y、Hetzel JA、Kuo F、Kim J、Cokus SJ等。 2010. 核小体定位与DNA甲基化的关系。 自然 466: 388–392 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Choufani S、Shapiro JS、Susiarjo M、Butcher DT、Grafodatskaya D、Lou Y、Ferreira JC、Pinto D、Scherer SW、Shaffer LG等。 2011 一种新的方法识别与印迹基因相关的新差异甲基化区域(DMR)。 基因组研究 21: 465–476 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Cuddapah S、Jothi R、Schones DE、Roh TY、Cui K、Zhao K 2009 染色质屏障区绝缘体结合蛋白CTCF的整体分析揭示了活性域和抑制域的划分。 基因组研究 19: 24–32 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Gal-Yam EN、Jeong S、Tanay A、Egger G、Lee AS、Jones PA 2006 单分子足迹显示GRP78启动子的组成核小体缺失和有序因子组装。 公共科学图书馆-基因 2:e160 doi:10.1371/journal.pgen.0020160 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Giresi PG、Kim J、McDaniell RM、Iyer VR、Lieb JD 2007 FAIRE(甲醛辅助分离调节元件)从人类染色质中分离活性调节元件。 基因组研究 17: 877–885 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Hansen KD、Timp W、Bravo HC、Sabunciyan S、Langmead B、McDonald OG、Wen B、Wu H、Liu Y、Diep D等。 2011 不同癌症类型表观遗传域甲基化变异增加。 自然基因 43: 768–775 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Hawkins RD、Hon GC、Lee LK、Ngo Q、Lister R、Pelizzola M、Edsall LE、Kuan S、Luu Y、Klugman S等。 2010 多能干细胞和谱系承诺的人类细胞的独特表观基因组景观。 细胞干细胞 6: 479–491 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Jessen WJ、Dhasarathy A、Hoose SA、Carvin CD、Risinger AL、Kladde议员 2004 使用DNA甲基转移酶绘制体内染色质结构。 方法 33: 68–80 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Jessen WJ、Hoose SA、Kilgore JA、Kladde议员 2006 活性PHO5染色质在单个启动子处包含不同数量的核小体。 自然结构分子生物学 13: 256–263 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Kelly TK、Miranda TB、Liang G、Berman BP、Lin JC、Tanay A、Jones PA 2010 H2A。 有丝分裂期间的Z维持揭示了核小体在有丝分裂沉默基因上的移位。 分子电池 39: 901–911 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Kilgore JA、Hoose SA、Gustafson TL、Porter W、Kladde MP 2007 使用DNA甲基转移酶对染色质结构进行单分子和群体探测。 方法 41: 320–332 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Kim TH、Abdullaev ZK、Smith AD、Ching KA、Loukinov DI、Green RD、Zhang MQ、Lobanenkov VV、Ren B 2007 人类基因组中脊椎动物绝缘体蛋白CTCF-结合位点的分析。 单元格 128: 1231–1245 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Krueger F、Kreck B、Franke A、Andrews SR 2012 使用短亚硫酸氢盐测序数据进行DNA甲基体分析。 Nat方法 9: 145–151 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Laks DR、Masterman-Smith M、Visnyei K、Angenieux B、Orozco NM、Foran I、Yong WH、Vinters HV、Liau LM、Lazaref JA等。 2009 神经层形成是恶性胶质瘤临床预后的独立预测因子。 干细胞 27: 980–987 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
李H、阮J、杜宾R 2008 使用映射质量分数映射短DNA测序读取和调用变体。 基因组研究 18: 1851–1858 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Lin JC、Jeong S、Liang G、Takai D、Fatemi M、Tsai YC、Egger G、Gal-Yam EN、Jones PA 2007 核小体占据在MLH1-CpG岛表观遗传沉默中的作用。 癌细胞 12: 432–444 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
李斯特R、佩利佐拉M、多文RH、霍金斯RD、Hong G、Tonti-Filippini J、Nery JR、Lee L、Ye Z、Ngo QM等。 2009 碱基分辨率的人类DNA甲基体显示出广泛的表观基因组差异。 自然 462: 315–322 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Liu Y、Siegmund KD、Laird PW、Berman BP 2012 Bis-SNP:结合DNA甲基化和SNP需要亚硫酸氢盐-seq数据。 基因组生物学 13:R61 doi:10.1186/gb-2012-13-7-r61 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Miranda TB、Kelly TK、Bouazoune K、Jones PA 2010. 染色质结构的甲基化敏感单分子分析。 电流质子分子生物学 89: 21.17.1–21.17.16 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Nagy PL、Price DH 2009 甲醛辅助分离调节元件。 Wiley Interdiscip Rev系统生物医学 1: 400–406 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Nakabayashi K、Trujillo AM、Tayama C、Camprubi C、Yoshida W、Lapunzina P、Sanchez A、Soejima H、Aburatani H、Nagae G等。 2011 互惠全基因组UPDs的甲基化筛选可识别新的人类特异性印迹基因。 人类分子遗传学 20: 3188–3197 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Pardo CE、Carr IM、Hoffman CJ、Darst RP、Markham AF、Bonthron DT、Kladde MP 2010 MethylViewer:针对单个模板(MAPit)项目的亚硫酸氢盐测序和甲基转移酶可及性协议的计算分析和编辑。 核酸研究 39:e5 数字对象标识代码:10.1093/nar/gkq716 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Rada-Iglesias A、Bajpai R、Swigut T、Brugmann SA、Flynn RA、Wysocka J 2011 一种独特的染色质特征揭示了人类早期发育增强因子。 自然 470: 279–283 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Schones DE、Cui K、Cuddapah S、Roh TY、Barski A、Wang Z、Wei G、Zhao K 2008 人类基因组中核小体定位的动态调节。 单元格 132: 887–898 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Taberlay PC、Kelly TK、Liu CC、You JS、De Carvalho DD、Miranda TB、Zhou XJ、Liang G、Jones PA 2011 多梳抑制基因具有启动重编程的允许增强子。 单元格 147: 1283–1294 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
宾夕法尼亚州琼斯市Takai D 2002 人类21号和22号染色体CpG岛的综合分析。 国家科学院程序 99: 3740–3745 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Thorvaldsdottir H、Robinson JT、Mesirov JP 2012 集成基因组查看器(IGV):高性能基因组数据可视化和探索。 生物信息简介 网址:10.1093/bib/bbs017 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Wilson背景,Roberts CW 2011 SWI/SNF核小体重塑剂与癌症。 Nat Rev癌症 11: 481–492 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Wolff EM、Byun HM、Han HF、Sharma S、Nichols PW、Siegmund KD、Yang AS、Jones PA、Liang G 2010 LINE-1启动子的低甲基化激活了患有癌症的膀胱中MET癌基因的替代转录物。 公共科学图书馆-基因 6:e1000917 doi:10.1371/journal.pgen.1000917 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Xi Y、Li W 2009 BSMAP:全基因组亚硫酸氢盐序列MAPping程序。 BMC生物信息学 10: 232 doi:10.1186/1471-2105-10-232 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Xie X、Mikkelsen TS、Gnirke A、Lindblad-Toh K、Kellis M、Lander ES 2007 系统发现人类基因组保守区域的调控基序,包括数千个CTCF绝缘体位点。 国家科学院程序 104: 7145–7150 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Xu M、Kladde MP、Van Etten JL、Simpson RT 1998 识别GpC的胞嘧啶-5-DNA甲基转移酶编码基因的克隆、表征和表达。 核酸研究 26: 3961–3966 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
You JS、Kelly TK、De Carvalho DD、Taberlay PC、Liang G、Jones PA 2011 OCT4建立并维持核小体缺失区域,为其靶基因提供额外的表观遗传调控层。 国家科学院程序 108: 14497–14502 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Genome Research的文章由以下机构提供 冷泉港实验室出版社