介绍 3-磷酸肌醇依赖性蛋白激酶-1(PDK1)是一种关键的蛋白激酶,通过磷酸化调节一组相关蛋白激酶的活性。 这些激酶包括蛋白激酶B(PKB;也称为Akt; 巴西和海明斯,2001年 ; Scheid和Woodgett,2001年 ),p70核糖体S6激酶(S6K; 沃拉雷维奇和托马斯,2001年 ),p90核糖体S6激酶(RSK; Frodin和Gammeloft,1999年 )血清和糖皮质激素诱导蛋白激酶(SGK); 朗和科恩,2001年 ). 这些酶受到激素和生长因子的刺激,磷酸化调节蛋白介导这些激动剂的各种生理效应。
PDK1具有一个N端激酶催化结构域和一个C端pleckstrin同源(PH)结构域( Alessi等人,1997年 ; Stephens等人,1998年 ). PDK1通过在激活环磷酸化这些激酶来激活其底物(在 托克和牛顿,2000年 ; 阿莱西,2001 ). PDK1对PKB的磷酸化依赖于磷脂酰肌醇3-激酶(PI-3-激酶)的事先激活和第二信使磷脂酰肌糖醇3,4,5-三磷酸[PtdIns(3,4,15)P的产生 三 ],其与PDK1和PKB的PH结构域结合。 铂族锡(3,4,5)P 三 不激活PKB或PDK1,而是在质膜上招募并共同定位这些酶,使PDK1激活PKB。 与PKB不同,到目前为止描述的其他PDK1基板不与PtdIns(3,4,5)P相互作用 3 PDK1与PtdIns(3,4,5)P的结合也不会进一步提高PDK1对其磷酸化的速率 三 相反,PDK1磷酸化S6K、SGK和RSK的能力是通过这些酶在位于疏水基序区域激酶催化域C末端的残基上的磷酸化来提高的( Alessi等人,1997年 ; Pullen等人,1998年 ; Kobayashi和Cohen,1999年 ). 磷酸化S6K和SGK疏水基序的激酶尚不清楚,但作为该残基的磷酸化 体内 依赖于PI-3-激酶的激活,疏水基序激酶和/或疏水基序磷酸酶可由PtdIns(3,4,5)P调节 三 在RSK亚型的情况下,ERK1/ERK2 MAP激酶的磷酸化诱导疏水基序的磷酸化(在 Frodin和Gammeloft,1999年 ).
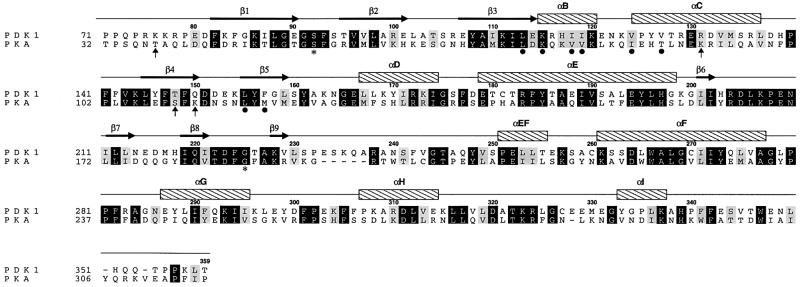
PDK1与其底物属于蛋白激酶的同一亚家族,称为AGC蛋白激酶,与c A类 MP依赖性蛋白激酶(PKA),c G公司 MP依赖性蛋白激酶和蛋白激酶 C类 (PKC)。 PKA是唯一一个晶体结构已被解决的AGC激酶( 泰勒 等 ., 1992 ). 与所有蛋白激酶一样,它的催化核心具有一个主要由β片组成的N末端叶和一个主要为α螺旋的C末端叶( 泰勒 等 ., 1992 ; Husen和Kuriyan,2002年 ). ATP结合位点位于两个叶之间( 骑士 等 ., 1991 ; 约翰逊 等 ., 2001 ). 在C末端,PKA具有一个延伸的环,终止于序列FXXF,类似于S6K和SGK(FXXFS/TY)疏水基序磷酸化位点的第一部分,其中Ser/Thr是磷酸化残基( 比昂迪 等 ., 2000 ). 在PKA结构中,FXXF基序埋藏在PKA催化结构域小叶的疏水口袋中( 骑士 等 ., 1991 )Phe残基的突变会显著降低PKA对肽底物的活性( 埃切舍尔 等 ., 1997 ; 巴特金 等 ., 2000 ). 与其他AGC激酶不同,PDK1的催化结构域没有疏水基序C末端。 然而,有证据表明PDK1在其催化结构域的小叶中具有类似于PKA的疏水囊。 我们已经通过生物化学证明,PDK1与其四种底物[S6K1、SGK1、PKCζ和PKC相关激酶-2(PRK2)]的相互作用被预测为构成该囊袋一部分的残基突变所减少或消除( 巴伦德兰 等 ., 2000 ; 比昂迪 等 ., 2000 ). 此外,预测囊中中心残基Leu155的突变阻止PDK1磷酸化和激活S6K1和SGK1,而不会影响其磷酸化PKB或包含PKB激活环的短肽底物(T308tide; 比昂迪 等 ., 2000 ). PDK1激酶结构域上的疏水口袋被称为“PIF口袋”,以发现结合PDK1的第一个含有AGC激酶疏水基序的肽(PDK1相互作用片段)的名称命名( 巴伦德兰 等 .,1999年a ). 有人建议,PDK1中的PIF-pocket起到对接位置的作用,使PDK1能够与其一些生理基质相互作用。 此外,有证据表明S6K1、SGK和RSK2疏水基序的磷酸化( 巴伦德兰 等 .,1999年b ; 弗罗丁 等 ., 2000 ; 比昂迪 等 ., 2001 )促进这些酶与PDK1的相互作用。 这些发现表明,PDK1上的PIF-口袋可能包含一个磷酸结合位点,一旦这些酶在其疏水基序被磷酸化,可以促进PDK1与底物子集(S6K、SGK和RSK)的结合。 这将导致生理上的磷依赖性相互作用。 此外,有证据表明PIF袋的占据激活了PDK1,因为包含PRK2疏水基序的肽( 比昂迪 等 ., 2000 )和RSK( 弗罗丁 等 ., 2000 )诱导PDK1活化4-6倍。
在这里,我们描述了PDK1激酶结构域的第一个晶体结构,分辨率为2Å。 该结构定义了PIF-pocket并揭示了相邻的磷酸盐结合位点。 此外,我们还进行了基于结构的突变和生化分析,证明存在这样一个磷酸结合位点,它可以介导与底物(如S6K和SGK)的磷酸依赖性对接相互作用。 最后,我们使用了一种新的算法来定义PDK1相对于所有已报道的PKA结构的构象状态,这表明虽然PDK1在晶体中具有活性形式的所有迹象,但其整体构象处于“开放”和“闭合”状态之间。
结果和讨论 总体结构 PDK1催化结构域的结构通过分子置换得到解决,并细化为 R(右) -系数0.19( R(右) 自由的 = 0.22). PDK1假定典型的双叶激酶折叠(图 1 )与唯一解决的其他AGC激酶结构类似,即PKA的PKA[均方根偏差(r.m.s.d.)为1.0Å α 蛋白质数据库(PDB)条目1STC中的原子( Prade等人,1997年 )]. 结晶的PDK1形式包含残基51–359。 如在其他激酶结构中所观察到的那样,激活环的尖端(残基233–236)被扰乱( 约翰逊等人,1996年 ). N端(残基51-70)指向晶体学对称性产生的大空洞,也处于无序状态。 相反,PKA激酶结构域的N末端延伸假定为两亲性α-螺旋(称为αA-螺旋),并紧靠激酶核心( Knighton等人,1991年 ). 在PKA中介导这种相互作用的疏水残基簇不存在于PDK1中,这表明PDK1的N末端可能具有与PKA不同的功能。 有趣的是,最近发现PDK1的N末端(残基1-50)与Ral鸟嘌呤核苷酸交换因子相互作用( 田等人,2002年 ). 因此,该区域可能在PDK1中具有独特的构象,而这种构象不是由此处描述的结构所定义的。
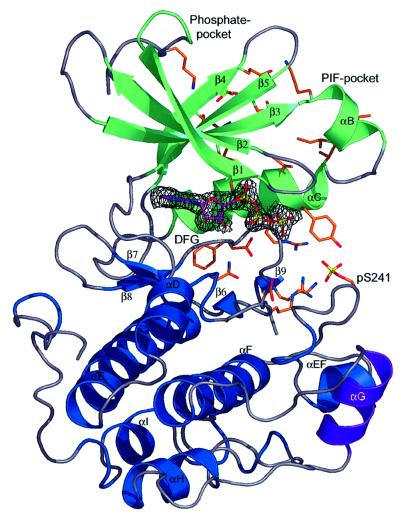
图1。 PDK1结构概述。 PDK1激酶结构域主干以带状表示显示,残基74-163的二级结构元素为绿色,残基164-358的二级构造元素为蓝色。 螺旋αG,包含残基287–295(与对称相关的PDK1分子进行晶体接触,图 2 ),是紫色的。 本文中讨论的关键残留物显示为带有橙色碳的棒状模型。 ATP显示为带有紫色碳原子的棒状模型。 ATP分子的模拟退火省略图以黑色显示,轮廓为3σ。 还显示了本文中讨论的磷丝氨酸(pS241)和硫酸盐。
PDK1在ATP存在下结晶,但没有任何二价阳离子。 在提纯的早期阶段,可以观察到整个ATP分子的明确密度。 ATP采用与其他激酶-ATP复合物中观察到的构象不同的构象(图 1 ). 可能是由于缺乏二价阳离子,PDK1结构中未发现与此类离子相互作用导致的β和γ磷酸盐之间普遍观察到的扭结。
众所周知,PDK1可以在激活环中的残基Ser241上磷酸化自身,并且这种磷酸化是PDK1活性所必需的( 卡萨马约 等 ., 1999 ). 事实上,我们观察到该残留物上附着的磷酸盐的密度(图 1 ),并且观察到该磷酸丝氨酸与来自C末端叶和αC螺旋的残基之间存在广泛的相互作用(图 1 ). Ser241和C末端叶之间的相互作用类似于PKA中的等效相互作用,但如下所述,与αC螺旋的结合不同。
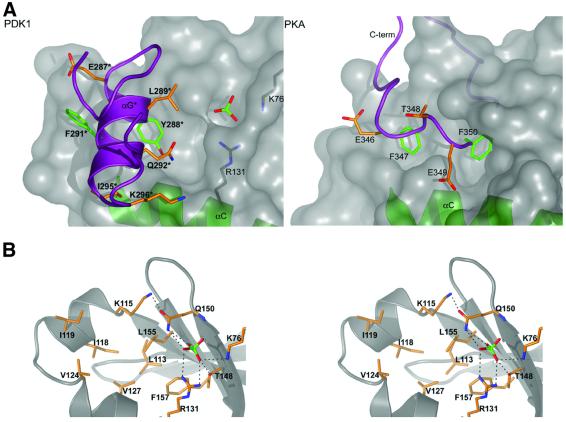
PIF口袋 如引言中所述,PDK1被假定在其催化结构域的小叶中有一个口袋(“PIF-口袋”),这是PDK1与其底物的疏水基序结合所必需的( 比昂迪 等 ., 2000 ). 这里描述的PDK1结构确实揭示了这样一个口袋,并表明它位于类似于PKA中FXXF绑定口袋的位置(图 2 ). αB螺旋上PDK1残基Lys115、Ile118、Ile119(图 2 )αC-螺旋上的Val124、Val127和β-片5上的Leu155形成了一个~5Å的深口袋。 先前的工作表明,Leu155突变为Glu消除了PDK1与包含PRK2疏水基序的肽相互作用的能力(PIFtide; 比昂迪 等 ., 2000 )以及S6K1、SGK1、PKCζ和PRK2( 巴伦德兰 等 ., 2000 ; 比昂迪 等 ., 2000 ). 此外,Lys115、Ile119、Glu150和Leu155突变为Ala,使PDK1对PIF肽的亲和力降低了约10倍,但并没有显著降低PDK1磷酸化和激活S6K1和SGK1的能力( 比昂迪 等 ., 2001 ). 这些结果与PIF-囊的晶体结构一致,因为Leu155位于中心,其他残留物排列在囊壁上(图 2 ). 有趣的是,在我们的结构中,PIF袋被对称相关分子的螺旋αG占据(图 2 ). Tyr288和Phe291在这个口袋中与几乎所有的袖珍残基进行疏水性接触,这让人想起PKA中FXXF基序中的苯丙氨酸相互作用以及它们在激酶结构域等效区域中的疏水对接位点(图 2 ). 此外,对称相关环上的残基Glu287、Gln292、Ile295和Lys296也与PIF囊内的残基形成接触。 总计244Å 2 可接触表面的。 这种相互作用的重要性尚不清楚,因为PDK1的齐聚反应之前尚未在溶液中证明。 事实上,PDK1的分离催化结构域(已结晶)和全长PDK1在凝胶过滤色谱中作为表观单体物种迁移(数据未显示)。
图2。 PIF口袋。 ( A类 )显示了假定的PIF结合囊的表面表示,并与PKA中与C末端FXXF基序相互作用的囊进行了比较。 对于PDK1,对称性相关分子的αG螺旋显示为紫色线圈,在PKA中,C末端显示为紫色圈。 口袋中埋藏的芳香氨基酸显示为带有绿色碳的棒状物,与口袋相互作用的进一步侧链显示为橙色碳。 螺旋αC在PDK1和PKA中均显示为绿色带状。 在PDK1中,还显示了有序硫酸根离子和与之相互作用的碱基残基。 星号表示对称相关分子的残基。 ( B类 )显示了PIF/磷酸盐容器内衬残留物的立体图像。 PDK1主干显示为灰色带状。 侧链显示为橙色碳原子。 硫酸根离子上的氢键显示为黑色虚线。
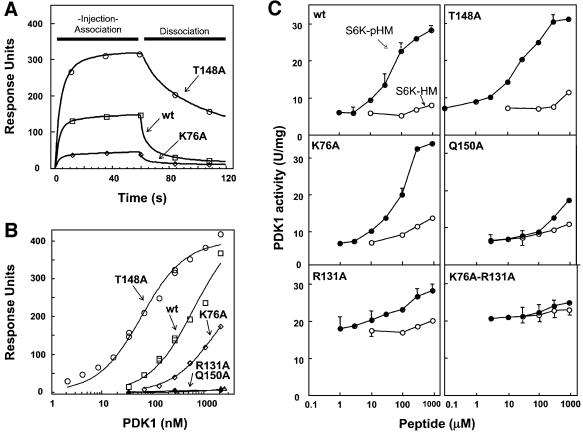
磷酸盐袋 如引言所述,当PDK1的底物(如S6K1)在疏水基序磷酸化时,它们与PDK1中的PIF-囊相互作用,亲和力更高。 这表明调节性磷酸盐结合位点可能位于PIF-囊袋附近。 在对PDK1结构进行精细化的过程中,很明显,在PIF袋的旁边存在另一个小口袋,由四面体氧离子占据(图 2 ). 由于结晶条件中存在2.0 M硫酸盐,因此将其指定为硫酸根离子。 该离子与口袋里的四个残基相互作用,即Lys76、Arg131、Thr148和Gln150。 由于其靠近PIF囊(~5Å),因此该硫酸盐占据的囊可能代表磷酸肽上磷酸的结合位点。 为了进一步研究这一点,我们将Lys76、Arg131、Thr148和Gln150突变为Ala,以验证这些残基中的每一个在使PDK1能够与包含S6K1疏水基序的肽相互作用中的作用,其中相当于Thr412的残基被磷酸化(称为S6K-pHM)。 基于表面等离子共振的定量结合分析(图 4 A) 表明野生型PDK1与S6K-pHM相互作用 K(K) d日 0.6µM,但该肽的非磷酸化形式(S6K-HM)无法检测到。 在本试验中,PDK1[R131A]和PDK1[Q150A]突变体未与S6K-pHM发生可检测的相互作用(图 4 B) ,确认这些残基在PDK1结构中的相互作用至关重要。 PDK1[K76A]突变体与3倍低亲和力相互作用( K(K) d日 =1.7µM),具有S6K pHM。 然而,PDK1[T148A]突变体的突变率高出约10倍( K(K) d日 =0.06µM)对S6K-pHM的亲和力高于野生型PDK1。 此外,PDK1[T148A]与S6K-pHM的分离明显慢于野生型PDK1或PDK1[K76A](图 4 A) ●●●●。 这些发现出乎意料,因为Thr148位于硫酸盐离子的氢键距离内(图 2 ),但表明该残基可能在使PDK1从S6K-pHM分离中发挥作用。
图4。 PDK1结合和活化研究。 野生型和突变型PDK1与源自S6K1疏水基序的磷酸肽的结合和激活。 如材料和方法中所述,通过表面等离子共振分析野生型(wt)PDK1和所示突变体与包含S6K1疏水基序的磷酸肽的结合(S6K-pHM:SESANQVFLGFT*YVAPSV,其中T*表示磷酸三氢嘌呤)。 ( A类 )传感器芯片SA涂有生物素化S6K-pHM肽的12 RU,并在注射270 nM野生型PDK1、PDK1[T148A]和PDK1[K76A]后分析其结合。 使用PDK1[R131A]或PDK1[Q150]未观察到与S6K-pHM的可检测结合(数据未显示)。 ( B类 )如(A)所示,但对PDK1浓度范围(2–2150 nM)的结合进行了分析。 绘制稳态结合时的响应水平与PDK1浓度的对数。 估计 K(K) d日 通过使用Kaleidrograph软件将数据拟合到公式[m1×m0/(m0+m2)]获得。 K(K) d日 对于野生型PDK1等于642±131 nM,PDK1[T148A] K(K) d日 =64±7 nM和PDK1[K76A] K(K) d日 =1744±167毫微米。 野生型PDK1或任何突变株均未检测到PDK1与非磷酸化S6K-HM肽(SESANQVFLGFTYVAPSV)的结合(数据未显示)。 ( C类 )S6K-pHM和S6K-HM激活指定形式的PDK1。如材料和方法中所述,在存在指定浓度的S6K-pHM(闭合圈)和S6K-HM(开放圈)的情况下,使用肽底物(T308tide)测量PDK1活性。 试验分为三次进行,两次单独的试验获得了类似的结果。 结果为单个实验的平均±SD。
PDK1与PIF肽的结合刺激PDK1磷酸化包含PKB激活环基序的小肽(称为T308肽; 比昂迪 等 ., 2000 )表明PDK1的PIF-口袋的占用激活了酶。 类似地,与RSK疏水基序对应的磷酸肽的结合刺激PDK1活性6倍( 弗罗丁 等 ., 2000 ). 我们现在还发现,S6K-pHM与野生型PDK1的结合诱导了最大5倍的活化,在约50µM的浓度下发生了半最大活化(图 4 C) ●●●●。 接下来,我们在没有或存在增加浓度的S6K-pHM的情况下,分析了PDK1[K76A]、PDK1[R131A]、PDK1[T148A]和PDK1[Q150A]突变体的特异性活性(图 4 C) ●●●●。 在没有S6K-pHM的情况下,PDK1[K76A]突变体对T308tide具有与野生型PDK1相同的特异性活性,但需要高出约3倍浓度的S6K-pHM才能半最大限度地激活PDK1[K76A],这与这种形式的PDK1对S6K-pKM的亲和力降低一致(图 4 A和B)。 在没有S6K-pHM的情况下,PDK1[R131A]突变体对Thr308肽具有3倍的高比活性(图 4 C) ,正如先前在PDK1的某些其他PIF口袋突变体(PDK1[K115A]和PDK1[L155E]; 比昂迪 等 ., 2000 ). 然而,根据PDK1[R131A]在BiaCore分析中不能结合S6K-pHM(图 4 B) ,当S6K-pHM浓度低于0.1 mM时,其活性没有显著激活,仅通过添加高浓度(0.3和1 mM)的S6K-pHM使其活性适度增加(图 4 C) ●●●●。 有趣的是,我们的研究结果表明,Arg131具有良好的调节作用,既参与了PDK1基础活性的抑制(因为Ala突变增加了基础活性),也参与了磷酸囊的激活过程。 其中Lys76和Arg131均变为Ala的PDK1突变体的活性被这些高浓度的S6K-pHM激活得更少。 在没有S6K-pHM的情况下,PDK1[T148A]和PDK1[Q150A]突变体对T308tide的特异性与野生型PDK1相似。 在存在S6K-pHM的情况下,PDK1[T148A]突变体与野生型PDK1相似地被激活。 与PDK1[Q150A]不能与S6K-pHM相互作用一致,此PDK1突变体在低于0.1 mM的S6K-pHM浓度下未被显著激活,但在0.3和1 mM的肽浓度下,观察到2到3倍的激活(图 4 ).
在非常高的肽浓度(0.3–1 mM)下,非磷酸化的S6K-HM肽诱导PDK1的少量(<2倍)活化(图 4 C) ●●●●。 有趣的是,尽管PDK1[K76A]和PDK1[R131A]突变体与磷酸化S6K-pHM肽的相互作用明显不如野生型PDK1,但高浓度的S6K-HM肽激活PDK1[K76A]和PDK1[R131A]的程度与野生型PDK1相似, 表明这些突变体与S6K-HM肽弱相互作用的能力没有受到影响。
我们评估了胰岛素/生长因子激活的AGC家族激酶(PKBα、S6K1、SGK1和RSK1)磷酸囊中的序列保守性。 序列比对表明该口袋在这些激酶中是保守的(图 5 B) ●●●●。 最保守的残基是Gln150,它存在于所有这些AGC激酶中,与Lys76等价的残基总是一个基本残基(图 5 B) ●●●●。 Arg131在S6K1、SGK1、RSK1和PKCβ中保守,但在PKBα、PKBβ或PKBγ中不保守,后者是Asn或Ser。 Thr148在PKBα和SGK1中保守,但在S6K1/RSK1中是Ala,在PKCβ中是Cys。 有趣的是,我们发现PDK1中的Thr148Ala突变没有破坏磷酸囊(图 4 ). 由于PKBα、S6K1、SGK1和RSK1需要在其疏水基序处磷酸化才能被最大限度地激活,因此很容易推测,当磷酸化时,这些酶的C末端疏水基序会与自身的PIF/磷酸囊结合,从而产生类似于PDK1的相互作用网络。 与不具有磷酸囊的PKA一致,Lys76和Gln150在PKA中不保守(图 三 )事实上,在PKA结构中没有观察到这种口袋(图 2 ).
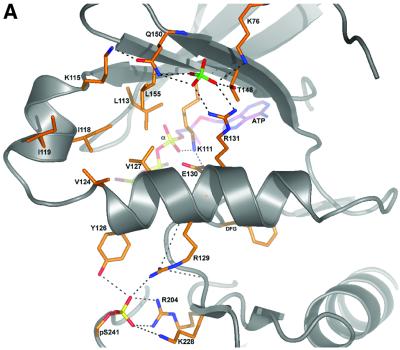
图5。 调节性磷酸盐与αC-螺旋的相互作用。 ( A类 )PDK1主干显示为带状,螺旋αC位于视图中心。 关键残留物显示为带有橙色碳的木棍。 还显示了活化回路上的硫酸根离子和磷酸盐。 ATP的棒状模型显示为紫色的碳纤维。 氢键显示为黑色虚线。 ( B类 )将构成PDK1上磷酸囊的一部分的氨基酸序列与所示AGC激酶的等效区域对齐。 PDK1的Lys76、Arg131、Thr148和Gln150对应的残基用粗体表示。
图3。 基于结构的序列对齐。 PKA和PDK1序列根据WHAT IF中执行的结构叠加进行对齐( 弗里德,1990年 ). 序列编号根据PDK1。 根据DSSP,PDK1结构显示了β股(箭头)和α螺旋(条)( 卡布施和桑德,1983年 )二级结构分配,并标记为与PKA建议的二级结构元素名称一致( Taylor和Radzioandzelm,1994年 ). PIF袋中的残留物用黑点表示。 与硫酸根离子形成氢键的残留物用箭头表示。 PKA中相当于Ser53和Gly186的PDK1残基用星号标记。
在传统PKC的情况下,疏水基序的磷酸化在稳定这些酶而不是触发它们的激活中发挥作用( Bornancin和Parker,1997年 ; 爱德华兹和牛顿,1997年 ). 最近的研究表明,PDK1在疏水基序磷酸化后,也与分离的PKCβC末端片段以更高亲和力的数量级相互作用( 高 等 ., 2001 ). 然而,有趣的是,当PDK1的疏水基序未磷酸化时,PDK1只能与全长PKC相互作用,并且该基序的磷酸化被证明以某种方式掩盖了该区域,阻止了它与PDK1相互作用( 高 等 ., 2001 ). 常规PKC亚型在去磷酸化状态下的疏水基序可能可用于与PDK1的相互作用,但在其磷酸化后,它将与自身激酶结构域中的等效PIF/磷酸结合囊相互作用更强烈,因此无法与PDK1.结合( 高 等 ., 2001 ). 我们的结果支持PKC亚型中存在一个磷酸结合位点,这可以解释磷酸化C末端疏水基序与其自身囊袋的高亲和力结合。 图中的对齐 5 A预测PKCβ中的Arg336、Lys391、Cys409和Gln411将包含PKCβ激酶催化域中的疏水基序磷酸结合位点。 因此,AGC激酶与PDK1的结合不仅取决于疏水基序周围的序列及其磷酸化状态,还取决于该序列的相互作用可用性。 PDK1的结构并不能解释为什么PDK1与PRK2(PIF)疏水基序的相互作用比S6K1和PKB在疏水基序磷酸化的等效片段更强( 比昂迪 等 ., 2001 ). PDK1与底物或包含疏水基序的肽结合的共结晶可以为PDK1与其底物之间的其他接触提供线索。
αC-螺旋 PDK1结构表明,与其他蛋白激酶一样( 约翰逊 等 ., 2001 ; Husen和Kuriyan,2002年 )αC-螺旋(残基124–136)是激酶核心中的关键信号整合基序。 PDK1αC-螺旋的一圈(残基129–131,图 三 和5)将N端叶、C端叶和活性部位连接在一起。 Arg129指向激活环并与磷酸化的Ser241形成两个氢键,而Arg131与磷酸盐囊中的硫酸盐形成两个氢键(图 5 ). Glu130与Lys111协调,Lys111与结合ATP的α-磷酸盐形成氢键。 这种相互作用在所有蛋白激酶中都是保守的,并且被证明对激活至关重要( 约翰逊 等 ., 2001 ; Husen和Kuriyan,2002年 ). 另一个残基Tyr126与磷酸化的Ser241形成第三个氢键。 αC螺旋上的Val124和Val127参与了PIF囊的形成(图 5 ). αC-螺旋在假定的磷酸肽结合囊和激活环中的磷酸丝氨酸之间提供了一个结构连接。 R131A比野生型PDK1具有更高的基础活性这一事实可能表明,该残基在PDK1结构中起调节作用,不仅参与磷酸根离子存在时PDK1的活化,而且在没有S6K-pHM的情况下保持酶向非活性构象的平衡。 据我们所知,这是首次报道αC-螺旋由螺旋两侧的两个调节性磷酸结合位点定位的激酶结构(图 5 ). 这提供了一种可能的传感器机制,用于将激活环的磷酸化状态和PIF-囊中的磷肽结合事件与PDK1活性联系起来。 我们的研究结果可以为以下观察提供解释:非活性的去磷酸化PKCβ可以与PDK1相互作用,而磷酸化的PKCβ则不能( 高 等 ., 2001 ). 这是可能的,因为在缺乏活化-磷酸化的情况下,αC-螺旋可能不会固定在能够形成疏水和磷酸囊的位置,从而允许疏水基序与其自身的激酶结构域结合。 在PDK1磷酸化激活环和疏水基序的自磷酸化后,PKCβ疏水和磷酸囊将像PDK1晶体结构中一样稳定。
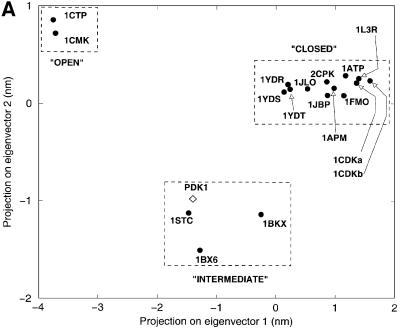
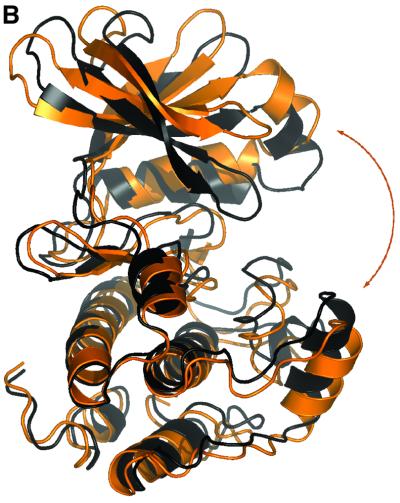
激活状态 迄今为止,PKA的所有结构都显示出磷酸化的T环,因此被认为处于活性状态。 除了PKA的非磷酸化状态和磷酸化状态外,后者还可能存在两种主要构象状态( 郑 等 ., 1993 ; 约翰逊 等 ., 2001 ). 在活性的闭合构象中,所有残基都被定位以促进磷酰转移。 相反,在没有核苷酸的情况下可以看到无活性的开放构象,并且通过N端和C端结构域的构象变化与闭合构象不同。 此外,从PKA中描述了三种“中间”结构,具有腺苷(PDB条目1BKX; 那罗延 等 ., 1997 )或抑制剂staurosporine(PDB条目1STC; 普拉德 等 ., 1997 )和巴洛诺(PDB条目1BX6; 那罗延 等 ., 1999 )ATP-结合位点。 泰勒 等 . (1992) 描述了一种基于三个距离区分活性构象和非活性构象的方法:His87–pThr197(αC螺旋定位)、Ser53–Gly186(富含甘氨酸环的开放)和Glu170–Tyr330(C末端到活性位点的尾部距离; 约翰逊 等 ., 2001 ). 在PDK1中,由于序列守恒,只能测量其中一个距离,即富含甘氨酸环的开放状态(图 三 ). 这个距离是12.4Å,类似于PKA中间体构象[PKA中的这个距离对于开放构象是14.2Å,对于中间体是11.8Å,对于封闭构象是10.0Å( 约翰逊 等 ., 2001 )]. 为了更直接地比较PDK1结构和可用的PKA结构,我们使用一种新的方法详细分析了PDK1的构象状态,该方法涉及主成分分析[也称为“基本动力学”( 阿马迪 等 ., 1993 )]晶体坐标。 简而言之,这涉及到构建一个协方差矩阵,其中包含所有可用PKA晶体结构系综中原子位移(相对于平均结构)之间的相关性。 该矩阵的对角化给出了特征向量/特征值集,它们描述了原子(特征向量)的协同位移以及结构(特征值)的相应均方波动。 这种方法允许仅使用几个自由度对PKA构象状态进行浓缩描述,如之前对一系列其他蛋白质所示( 范·阿尔滕 等 ., 1997 , 2000 ; 德格罗 等 ., 1998 ). 将由残基37–196、198–283和286–305的主链原子构建的协方差矩阵对角化,得到一组描述PKA主链协同运动的特征向量。 在图中 6 A、 所有PKA结构都投影到由前两个特征向量(即具有两个最大特征值的特征向量)跨越的子空间上。 PKA结构似乎沿着第一特征向量在三个主要区域聚集。 在平均结构的左侧(根据定义,所有特征向量上的投影为0.0)是已知处于“开放”构象的结构(图 6 A) ●●●●。 平均结构的周围是显示为“中间”构象的结构(与抑制剂staurosporine、balanol和腺苷的络合物)。 平均结构右侧更多的是已知处于“闭合”构象的PKA结构。 因此,我们捕获了PKA在单个变量中的构象状态,即沿着第一个特征向量的平移。 通过对笛卡尔空间中该特征向量所描述的原子位移的研究,进一步阐明了这一点(图 6 B) ●●●●。 在N端和C端叶之间观察到铰链弯曲运动,打开和关闭活动部位。 现在可以通过将结构(仅骨干原子)投影到PKA特征向量上直接比较PDK1构象状态。 图 6 A表明PDK1的构象接近于处于“中间”构象的PKA结构,与上述其他结构分析一致。
图6。 基本动力学。 ( A类 )将所有可用的PKA晶体结构(标记点)和PDK1结构(钻石)投影到根据PKA结构计算的前两个特征向量(具有两个最大特征值的特征向量)上。 ( B类 )沿着第一个特征向量的运动的图形表示,通过在–4 nm(黑色)和+4 nm(橙色)处投影两个结构生成。
结论 我们已经报道了PDK1催化结构域的结构,该结构域虽然与PKA类似,但也揭示了一些重要特征,这些特征增加了我们对PDK1调控机制的理解。 该结构与突变分析一起定义了一个磷酸肽结合囊,由一个单独的疏水性PIF囊和一个磷酸结合位点组成,该位点介导PDK1与S6K磷酸化疏水基序的相互作用。 这与之前的假设一致,即S6K和SGK的磷酸化( 比昂迪 等 ., 2001 )以及RSK( 弗罗丁 等 ., 2000 )在它们的FXXFS/T疏水基序是它们相互作用和被PDK1磷酸化的触发因素。 在这种机制中,当Ser/Thr残基被磷酸化时,PIF-囊在生理上仅与Phe残基相互作用。 此外,由于磷酸囊在其他AGC激酶中是保守的,PDK1上磷酸囊与αC螺旋相互作用的结构特征和网络可以提供对其他AGC酶激活模式的了解。
许多人类癌症,例如PTEN抑癌基因突变的癌症,PtdIns(3,4,5)P显著升高 三 导致PKB和S6K组成性激活的水平,这可能是这些肿瘤细胞增殖和提高生存率的主要因素( 莱斯利和唐斯,2002年 ). PDK1抑制剂可以通过抑制PKB和S6K活性来证明对这些类型的癌症的治疗有效。 PDK1催化结构域的结构为开发靶向ATP结合位点或PIF结合口袋的抑制剂提供了分子框架。 这两种药物都会阻碍PDK1的功能 体内 ,但后一类药物的优点是,它们可能比ATP-竞争性抑制剂更具特异性,因此不太可能抑制细胞中其他蛋白激酶的活性。
材料和方法 材料 哺乳动物和昆虫细胞培养试剂来自Life Technologies。 SensorChips SA来自BiaCore AB(Stevenage,英国)。 谷胱甘肽–Sepharose以及预包装HiTrap Q HP和Hiload Superdex 200预处理级色谱柱来自Amersham Biosciences。 透析盒来自Slide-A-Lyzer系列(Pierce)。 Ni-NTA–琼脂糖来自恰根氏。 一次性超滤装置(聚醚砜膜)来自Vivascience。 结晶研究工具(主筛、添加剂筛和结晶板)来自汉普顿研究公司。 肽由英国布里斯托尔大学的G.Blomberg博士合成。
一般方法 分子生物学技术使用标准协议进行。 按照制造商提供的说明,使用QuikChange试剂盒(Stratagene)进行定点突变。 根据制造商的协议,使用Qiagen质粒Mega试剂盒从细菌中纯化用于转染的DNA构建物,并验证其序列。 人肾胚胎293细胞在直径10 cm的培养皿中培养,培养液为含有10%胎牛血清的Dulbecco改良Eagle's培养基。
缓冲器 低盐缓冲液:25 mM Tris–HCl pH 7.5,150 mM NaCl; 高盐缓冲液:25 mM Tris–HCl pH 7.5,500 mM NaCl。 溶解缓冲液:25 mM Tris–HCl pH 7.5,150 mM NaCl 0.07%β-巯基乙醇,1 mM苯甲脒和20µg/ml PMSF。 缓冲液A:50 mM Tris–HCl pH 7.5,1 mM EGTA,1 mM-EDTA,1%(按质量计)Triton X-100,1mM原钒酸钠,50 mM氟化钠,5 mM焦磷酸钠,0.27 M蔗糖,1µM微囊藻毒素-LR,0.1%(按体积计)β-巯基乙醇和“完全”蛋白酶抑制剂混合物(每50 ml一片;罗氏)。 缓冲液B:50 mM Tris–HCl pH 7.5,0.1 mM EGTA,10 mMβ-巯基乙醇和0.27 M蔗糖。
PDK1激酶结构域的表达、纯化和表征 在pCMV5载体中使用全长人PDK1 cDNA,通过PCR扩增了一个编码人类PDK1氨基酸残基51–359的cDNA,其终止密码子插入位置360( Alessi等人,1997年 )作为模板。 所采用的PCR策略在基因的5′端包含一个起始的蛋氨酸,一个His 6 标记后接PreScission蛋白酶识别序列,该序列位于PDK1的Met51残基之前(GGATCCATAAATGGCACATCATCATCATCATCTGGAAGTTCCAGGCCATGGACCACGGCGCCGG)。 该反应中使用的3′引物为:5′-GGA-TCCTCAGTGGAGCTTCGGAGGCGTGTGGTG-3′。 将得到的PCR产物连接到PCR 2.1 TOPO载体(Invitrogen)中,然后亚克隆为 巴姆 HI–高 巴姆 将HI片段插入pFastbac1载体(Life Technologies)中用于杆状病毒蛋白表达。 然后,按照制造商的协议,使用Bac-to-Bac系统(生命科技)将得到的构建物用于生成重组杆状病毒。 用产生的杆状病毒感染1.5×10的Sf21细胞 6 /毫升。 感染后72小时离心收集感染细胞。 将相当于7 l培养物的细胞颗粒重新悬浮在900 ml Lysis缓冲液中,并在氮气空化室中溶解细胞。 然后通过离心法造粒细胞碎片,上清液通过添加4 M NaCl生成0.5 M NaCl,然后与Ni-NTA–琼脂糖(10 ml树脂)孵育1 h。然后用40 ml含有0.5 M NaCl的Lysis缓冲液清洗树脂10×,然后放置在一次性Econo-Pac柱(Bio-Rad)中, 其中,用700 ml高盐缓冲液进一步清洗树脂,然后用100 ml低盐缓冲液清洗树脂,这两种缓冲液都补充有10 mM咪唑。 在高盐缓冲液中用200mM咪唑进行洗脱,并收集2ml级分。 将含有蛋白质的级分合并,用25 mM Tris–HCl pH 7.5稀释至200 mM NaCl,并加载到5 ml Hi-trap Q琼脂糖柱上。 将该步骤产生的含有PDK1的流出液浓缩至4 ml,然后使用AKTA Explorer系统(Amersham Biosciences)在16/60 Superdex 200凝胶过滤柱上进行色谱分析,该系统与高盐缓冲液平衡,并添加1 mM二硫苏糖醇(DTT)。 PDK1在单体预期大小的大对称峰中洗脱。 再次汇集、浓缩含有PDK1的峰,并用300µg谷胱甘肽孵育 S公司 -转移酶(GST)–切前蛋白酶(表达结构由英国伯明翰大学John Heath善意提供)在冰上放置4小时。 为了消除已切割的His-tag序列以及任何剩余的未切割His-PDK1和GST-PreScission蛋白酶,将混合物与200µl谷胱甘肽-Sepharose和200µl Ni-NTA-琼脂糖树脂的混合物培养15分钟,并收集未结合的PDK1蛋白。 产生的蛋白质由PDK1(51–359)组成,其N端有Gly-Pro。 在SDS-聚丙烯酰胺凝胶上电泳20µg蛋白质并用考马斯蓝R250染色后,纯化阶段的蛋白质明显均匀,显示为单一条带(数据未显示)。 电喷雾质谱显示,PDK1片段的主峰质量接近预期大小。 PDK1(51–359)对肽T308肽的比活性及其在PIF肽存在下的活化与野生型全长PDK1相同( Biondi等人,2000年 )胰蛋白酶肽质量指纹图谱显示PDK1在Ser241处被定量磷酸化(数据未显示)。 在BiaCore实验中,PDK1(51–359)与肽PIFtide的稳态结合类似于之前描述的His-tag PDK1蛋白(51–556)( Balendran等人,1999年a ).
结晶和数据收集 将PDK1(51–359)蛋白浓缩至最终浓度8.5 mg/ml(根据以牛血清白蛋白为标准的Bradford分析测定)。 采用坐滴蒸汽扩散法生产晶体。 将1µl蛋白质溶液+0.2µl EDTA(100 mM)与1µl母液溶液(0.1 M Tris–HCl pH 8.5,2.0 M硫酸铵,16.6 mM ATP)混合,形成坐液。 六角晶体(表 我 )PDK1在20°C下从含有0.1 M Tris–HCl pH 8.5、2.0 M硫酸铵、16.6 mM ATP的母液中生长。 晶体在1天后出现,20天后增长到0.05×0.05×0.2 mm。 将晶体浸泡在0.075 M Tris pH 8.5、1.5 M硫酸铵和25%甘油中后,在氮气流中冷冻。
表I.PDK1激酶结构域的数据收集和结构改进细节 一 。
波长(Å) 0.933
“空间”组
P(P) 三 2 21
单位电池(Å)
一 =123.01, b条 =123.01, c(c) = 47.62
分辨率(Å) 25–2.0 (2.07–2.0)
观察到的反射 77 315
独特的反射 27 643
冗余 2.8(2.5)
完整性(%) 98.0(93.5)
R(右) 合并
0.091 (0.454)
我 /σ 我
7.3 (2.0)
R(右) 自由的 反思
579
R(右) 晶体
0.195
R(右) 自由的
0.222
组的数量
蛋白质残留物 71–359
水 200
列车自动防护系统 1
SO公司 4个 5
甘油 8
威尔逊B(Å 2 ) 22.4
< B> 蛋白质 25.6
< B> 水 35.7
< B> 列车自动防护系统 38.8
理想几何的R.m.s.d
粘合长度(Å) 0.005
结合角(°) 1.34
主链B(Å 2 ) 1.5
GST–PDK1野生型和突变型的表达和纯化 野生型PDK1( Alessi等人,1997年 )pEBG2T载体中的PDK1[R76A]、PDK1[R131A]、PDK1[R76 A、R131 A]、PD K1[T148A]和PDK1[Q150A]被用于表达野生型和指示的PDK1突变体,这些突变体通过其N末端融合到GST。 GST融合蛋白在人胚肾293细胞中表达。 为了表达每个构建物,培养了20个直径为10 cm的培养皿(293个细胞),并使用改进的磷酸钙方法将10µg的pEBG-2T构建物转染到每个培养皿中。 转染后36小时,将细胞溶解在0.6 ml冰镇缓冲液A中,将溶解液混合,在4°C下离心10分钟,在13000 克 GST融合蛋白通过谷胱甘肽-Sepharose亲和层析纯化,并在补充有20mM谷胱甘肽的缓冲液B中洗脱,如前所述( Alessi等人,1997年 ). 根据SDS-PAGE判断(数据未显示),每个GST融合蛋白通常在1到2 mg之间,每个蛋白的纯度>75%。
PDK1催化活性测量 野生型和突变型PDK1磷酸化合成肽T308肽(KTFCGTPEYLAPEVRR)的能力; 比昂迪 等 ., 2000 )在含有100 ng野生型或突变型PDK1、50 mM Tris–HCl pH 7.5、0.1%β-巯基乙醇、10 mM MgCl的30µl试验中进行 2 ,100µM[γ- 32 P] ATP(200 c.P.m/pmol)、0.5µm微囊藻毒素-LR、1 mM T308肽,存在或不存在指定浓度的S6K-pHM肽[SESANQVFLGFT(P)YVAPSV]或S6K-HM肽(SESANQVFLGFTYVAPSV)。 在30°C下培养10 min后,将25µl所得混合物点在P81磷酸纤维素纸(2×2 cm)上,并按照前面所述对纸张进行清洗和分析,以进行MAP激酶的检测。 平行进行对照分析,其中省略PDK1或肽底物; 这些值始终小于在这些试剂存在下测得的活性的5%。 PDK1活性的一个单位被定义为在1分钟内催化1 nmol T308肽磷酸化所需的量。
BiaCore分析 结合在BiaCore 3000系统(BiaCoreAB)中进行分析。 生物素化S6K-pHM[生物素-C 12 -SESANQVFLGFT(P)YVAPSV]或该肽的非磷酸化形式S6K-HM与链霉亲和素涂层传感器芯片(SA)结合(12个响应单位,RU)。 以30µl/min的流速将30µl野生型或所示突变型GST–PDK1等分样品注入补充有1 mM DTT的缓冲液HBS-P[10 mM HEPES pH 7.4,0.15 M NaCl,0.005%(体积)聚山梨酯-20]中。 S6K-pHM和PDK1蛋白之间的特定相互作用在2–2150 nM PDK1的浓度范围内获得。 在每个浓度下测定稳定结合。 在1分钟内监测PDK1与磷酸肽的分离。 通过10µl注入0.05%十二烷基硫酸钠对传感器芯片表面进行再生。 如前所述,PDK1与PIFtide结合( 比昂迪 等 ., 2000 ),使用BiaCore获得的交互数据不符合简单的1:1交互模型。 对于所有测试的PDK1结构,与PIFtide结合相比,S6Kp-HM的关闭率较高,S6K-pHM发生50%分离的时间为30秒,而PIFtied为1000秒。 这可能是S6K-pHM取代PIFtide结合能力降低的原因( 比昂迪 等 ., 2000 ).
数据收集、结构解决和细化 使用ADSC Q4 CCD探测器,在欧洲同步辐射设施(法国格勒诺布尔)光束线ID14-EH1收集PDK1晶体的数据。 使用氮气冷冻流将晶体温度保持在100K。 使用HKL包处理数据( Otwinowski和Minor,1997年 ),统计数据如表所示 我 。
PDK1的结构通过AMoRe的分子置换来解决( 纳瓦扎,1994年 )使用PKA与抑制肽复合物的结构作为搜索模型(PDB条目1YDB),针对8-4Å数据。 发现一种分离良好的单一溶液 R(右) -系数为0.479(相关系数=0.428)。 该结构是使用warpNtrace自动构建的( Perrakis等人,1999年 )发现了262个可能的309个残基,给出了一个初始的蛋白质模型 R(右) = 0.293 ( R(右) 自由的 =0.318)在CNS中模拟退火后( Brünger等人,1998年 ). O中的迭代蛋白质构建( Jones等人,1991年 )再加上CNS的改进,包括纳入ATP模型、活化环中的磷丝氨酸、溶剂分子和关键硫酸盐分子,最终形成了一个具有 R(右) = 0.195 ( R(右) 自由的 = 0.222). 残基51–70(结构的N末端)和233–236(激活环的尖端)未观察到电子密度。
所有人物都是用PyMOL制作的( http://www.pymol.org ).
PDB坐标 坐标和结构系数已存放在PDB中(条目1h1w)。
致谢 致谢 我们感谢格勒诺布尔欧洲同步辐射设施(European Synchrotron Radiation Facility,Grenoble)在束线ID14-EH1上提供的时间,感谢阿格涅斯卡·基洛赫(Agnieszka Kieloch)在组织培养方面提供的协助,感谢费利西蒂·纽厄尔(Felicity Newell)在杆状病毒生产方面提供的帮助,感谢尼克·莫里斯(Nick Morrice)在质谱分析方面提供的支持,感谢邓迪大学生命科学, 用于DNA测序。 我们还感谢Jennifer Gallop和Evangelis Dioletis在这项工作的早期阶段提供的帮助。D.K.得到了MRC产前研究金(C.C.T.)、BBSRC CASE学生金(D.M.F.v.a.)、Wellcome Trust职业发展研究金(D.R.a.)、英国糖尿病医学研究委员会(英国)、, 国际癌症研究协会(Association for International Cancer Research),以及支持邓迪信号转导治疗部门的制药公司(阿斯利康(AstraZeneca)、博林格殷格海姆(Boehringer Ingelheim)、葛兰素史克(GlaxoSmithKline)、诺和诺德(Novo-Nordisk)、辉瑞(Pfizer))。
工具书类
Alessi D.R.(2001)发现胰岛素信号转导中缺失的环节之一PDK1。 生物化学。 Soc.Trans.公司。, 29, 1–14. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
阿莱西·D.R。 等 (1997)3-磷酸肌醇依赖性蛋白激酶-1(PDK1):与 果蝇属 DSTPK61激酶。 货币。 生物学,776–789。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Amadei A.,Linssen,A.B.M.和Berendsen,H.J.C.(1993)蛋白质的基本动力学。 蛋白质,17,412–425。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Balendran A.、Casamayor A.、Deak,M.、Paterson,A.、Gaffney,P.、Currie,R.、Downes,C.P.和Alessi,D.R.(1999a)Pdk1在含有源自PRK2羧基末端的合成肽的情况下获得PDK2活性。 货币。 生物,9393–404。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Balendran A.、Currie,R.、Armstrong,C.G.、Avruch,J.和Alessi,D.R.(1999b)3-磷酸肌醇依赖性蛋白激酶-1介导p70 S6激酶磷酸化的证据 体内 Thr-412和Thr-252。 生物学杂志。 化学。, 274, 37400–37406. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Balendran A.、Biondi,R.M.、Cheung,P.C.F.、Casamayor,A.、Deak,M.和Alessi,D.R.(2000)PDK1对蛋白激酶Cζ(PKCζ)和PKC相关激酶2进行磷酸化需要3-磷酸肌醇依赖性蛋白激酶-1(PDK1)对接位点。 生物学杂志。 化学。, 275, 20806–20813. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Batkin M.,Schvartz,I.和Shaltiel,S.(2000)将PKA催化亚单位的羧基末端末端捕捉到其核心:通过诱变表征位点。 生物化学,39,5366–5373。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Biondi R.M.、Cheung,P.C.F.、Casamayor,A.、Deak,M.、Currie,R.A.和Alessi,D.R.(2000)PDK1激酶结构域中与PIF和PKA C末端残基相互作用的口袋的鉴定。 EMBO J.,1979-988年。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Biondi R.M.、Kieloch,A.、Currie,R.A.、Deak,M.和Alessi,D.R.(2001)PDK1中的PIF-结合囊对S6K和SGK的激活至关重要,但对PKB则不然。 EMBO J.,20,4380–4390。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Bornancin F.和Parker,P.J.(1997)丝氨酸657上蛋白激酶C-ζ的磷酸化控制活性酶的积累,并有助于其抗磷酸酶状态。 生物学杂志。 化学。, 272, 3544–3549. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
巴西D.P.和Hemmings,B.A.(2001)蛋白质激酶B信号传递的十年:很难遵循Akt。 生物化学趋势。 科学。, 26, 657–664. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
布伦格A.T。 等 (1998)结晶和核磁共振系统:一种新的大分子结构测定软件系统。 《水晶学报》。 D、 54、905–921。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Casamayor A.,Morrice,N.A.和Alessi,D.R.(1999)Ser-241的蛋白质磷酸化对3-磷酸肌醇依赖性蛋白激酶-1的活性至关重要:五个磷酸化位点的鉴定 体内 .生物化学。 J.,第342页,第287–292页。 [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
de Groot B.L.、Hayward,S.、van Aalten,D.M.F.、Amadei,A.和Berendsen,H.J.C.(1998)噬菌体T4溶菌酶的结构域运动:分子动力学和晶体学数据之间的比较。 蛋白质,31116-127。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Edwards A.S.和Newton,A.C.(1997)保守羧基末端疏水基序的磷酸化调节蛋白激酶C.J.Biol的催化和调节域。 化学。, 27218382-18390之间。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Etcheshere L.C.、Van Bemmelen、M.X.P.、Anjard,C.、Traincard,F.、Assemat,K.、Reymond,C.和Veron,M.(1997) 粘液菌 cAMP依赖性蛋白激酶N端结构域和C端残基的催化活性和稳定性。 《欧洲生物化学杂志》。, 248, 820–826. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Frodin M.和Gammeloft,S.(1999)90kDa核糖体S6激酶(RSK)在信号转导中的作用和调节。 分子细胞。 内分泌。, 151, 65–77. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Frodin M.,Jensen,C.J.,Merienne,K.和Gammeloft,S.(2000)蛋白激酶RSK2中的磷酸丝氨酸调节对接位点,可招募和激活PDK1。 EMBO J.,192924–2934。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Gao T.Y.,Toker,A.和Newton,A.C.(2001)蛋白激酶C的羧基末端提供了一个开关来调节其与磷脂依赖性激酶PDK1的相互作用。 生物学杂志。 化学。, 276, 19588–19596. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Husen M.和Kuriyan,J.(2002)蛋白质激酶的构象可塑性。 电话:109275–282。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Johnson D.A.、Akamine,P.、Radzio-Andzelm,E.、Madhusudan和Taylor,S.S.(2001)cAMP依赖性蛋白激酶的动力学。 化学。 版次:1012243–2270。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Johnson Negal,Noble,M.E.M.和Owen,D.J.(1996)《活性和非活性蛋白激酶:调控的结构基础》。 细胞,85,149–158。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Jones T.A.、Zou,J.Y.、Cowan,S.W.和Kjeldgaard,M.(1991)电子密度图中构建蛋白质模型的改进方法以及这些模型中错误的位置。 《水晶学报》。 A、 47、110–119。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Kabsch W.和Sander,C.(1983)《蛋白质二级结构词典:氢键和几何特征的模式识别》。 生物聚合物,222577–2637。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Knighton D.R.、Zheng,J.H.、Teneyck,L.F.、Ashford,V.A.、Xuong,N.H.、Taylor,S.S.和Sowadski,J.M.(1991)环磷酸腺苷依赖性蛋白激酶催化亚基的晶体结构。 科学,253407-414。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Kobayashi T.和Cohen,P.(1999)激活磷脂酰肌醇3-激酶的激动剂激活血清和糖皮质激素调节蛋白激酶是由3-磷酸肌醇依赖性蛋白激酶-1(PDK1)和PDK2介导的。 生物化学。 《华尔街日报》,第339、319–328页。 [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Lang F.和Cohen,P.(2001)血清和糖皮质激素诱导的蛋白激酶亚型的调节和生理作用。 科学,108,RE17。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Leslie N.R.和Downes,C.P.(2002)PTEN:PI 3-激酶信号的负面影响。 单元格。 信号。, 14, 285–295. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Narayana N.,Cox,S.,Xuong,N.H.,Ten Eyck,L.F.和Taylor,S.S.(1997)cAMP依赖性蛋白激酶和腺苷催化亚单位的二元复合物进一步定义了构象的灵活性。 结构,5921–935。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Narayana N.,Diller,T.C.,Koide,K.,Bunnage,M.E.,Nicolaou,K.C.,Brunton,L.L.,Xuong,N.H.,Ten Eyck,L.F.和Taylor,S.S.(1999)强效天然产物抑制剂巴洛尔与cAMP依赖性蛋白激酶催化亚基复合物的晶体结构。 生物化学,38,2367–2376。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Navaza J.(1994)AMoRe:分子替换的自动化包。 《水晶学报》。 A、 50、157–163。 [ 谷歌学者 ]
Otwinowski Z.和Minor,W.(1997)振荡模式下收集的X射线衍射数据的处理。 方法酶学。, 276, 307–326. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Perrakis A.,Morris,R.和Lamzin,V.S.(1999)结合迭代结构细化的自动化蛋白质模型构建。 自然结构。 生物学,6458–463。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Prade L.,Engh,R.A.,Girod,A.,Kinzel,V.,Huber,R.和Bossemeyer,D.(1997)Staurosporine诱导的cAMP依赖性蛋白激酶催化亚单位的构象变化解释了抑制潜力。 结构,51627–1637。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Pullen N.,Dennis,P.B.Andjelkovic,M.,Dufner,A.,Kozma,S.C.,Hemmings,B.A.和Thomas,G.(1998)p70的磷酸化和活化 6000万美元 由PDK1提供。 科学,279707-710。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Scheid M.P.和Woodgett,J.R.(2001)PVB/Akt:遗传模型的功能见解。 国家分子细胞生物学杂志。, 2, 760–768. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
斯蒂芬斯·L。 等 .(1998)介导磷脂酰肌醇3,4,5-三磷酸依赖性激活蛋白激酶B的蛋白激酶B激酶。科学,279710-714。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Taylor S.S.和Radzioandzelm,E.(1994)三种蛋白激酶结构定义了一个共同的基序。 结构,2345–355。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Taylor S.S.、Knighton,D.R.、Zheng,J.H.、Teneyck,L.F.和Sowadski,J.M.(1992)蛋白质激酶家族的结构框架。 每年。 细胞生物学评论。, 8, 429–462. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Tian X.J.、Rusanescu,G.、Hou,W.M.、Schaffhausen,B.和Feig,L.A.(2002)PDK1通过激酶依赖机制介导生长因子诱导的Ral-GEF活化。 EMBO J.,第21期,1327–1338页。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
Toker A.和Newton,A.C.(2000)《细胞信号:围绕PDK1旋转》。 细胞,103,185–188。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
van Aalten D.M.F.,Conn,D.A.,de Groot,B.L.,Berendsen,H.J.C.,Findlay,J.B.C.和Amadei,A.(1997)晶体结构簇衍生的蛋白质动力学。 生物物理学。 J.,73,2891–2896。 [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]
van Aalten D.M.F.,Chong,C.R.和Joshua-Tor,L.(2000)羧肽酶A与D-半胱氨酸在1.75Å处络合的晶体结构:抑制剂诱导的构象变化。 生物化学,39,10082–10089。 [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Volarevic S.和Thomas,G.(2001)S6磷酸化和S6激酶在细胞生长中的作用。 掠夺。 核酸研究分子生物学。, 65, 101–127. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Vriend G.(1990)假设:分子建模和药物设计程序。 J.摩尔图。, 8, 52–56. [ 内政部 ] [ 公共医学 ] [ 谷歌学者 ]
Zheng J.H.、Knighton,D.R.、Xuong,N.H.、Taylor,S.S.、Sowadski,J.M.和Teneyck,L.F.(1993)cAMP依赖性蛋白激酶的肉豆蔻酰化催化亚基的晶体结构揭示了开放和闭合构象。 蛋白质科学。, 2, 1559–1573. [ 内政部 ] [ PMC免费文章 ] [ 公共医学 ] [ 谷歌学者 ]