附加文件中提供了以下章节中使用的符号摘要1:表S1。
模型和规范化
读取计数K(K)
ij公司
对于基因我在样品中j个用带对数链接的负二项族的GLM描述:
(1)
(2)
为了简化符号,这里的方程式使用自然对数作为链接函数,尽管DESeq2公司软件在log2尺度上报告估计的模型系数及其估计的标准误差。
默认情况下,标准化常量秒
ij公司
在样品中被视为常数,秒
ij公司
=秒
j个
,并使用之前描述和使用的比率中位数方法进行估计DESeq公司[4]和DEX当量[30]:
或者,用户可以提供标准化常数秒
ij公司
使用其他方法计算(例如,使用立方厘米[13]或EDASeq公司[14])这可能因基因而异。
扩展的设计矩阵
为了与我们的软件文档保持一致,在下面的文本中,我们将使用对统计语言。在线性建模中,分类变量或因素可以采用两个或多个值,或者水平在标准设计矩阵中,选择其中一个值作为参考值或基准面并被截获。在标准GLM中,基准水平的选择不会影响对比度(LFC)的值。然而,在我们的方法中,当设计矩阵中存在两个以上水平的因素时,系数(如下所述)上的脊状再回归收缩不再是这种情况,因为基准面不会收缩,而其他水平会收缩。
为了恢复所有级别之间所需的对称性,DESeq2公司使用扩展设计矩阵,其中包括一个用于每个除截距列外,每个因子的水平(即,没有任何水平被截距吸收)。虽然这样的设计矩阵不再具有满秩,但存在唯一的解决方案,因为以零为中心的先验分布(见下文)提供了正则化。对于色散估计和LFC先验宽度估计,使用标准设计矩阵。
对比
此类对比度的水平和标准误差之间的对比度可以按照标准设计矩阵的情况进行计算,即使用:
(3)
(4)
哪里表示数值对比度,例如,1和−1指定简单两级对比度的分子和分母,以及,定义如下。
分散度估算
我们假设色散参数α
我
遵循以趋势为中心的对数正态先验分布,该趋势取决于基因的平均标准化读取计数:
(5)
在这里,α tr公司是基因平均归一化计数的函数,
它描述了先验的均值相关期望。σ d日是先验值的宽度,这是一个超参数,描述单个基因在趋势上的真实分散程度。对于趋势函数,我们使用的参数化与我们使用的相同DEXSeq公司[30]即,
(6)
我们通过三个步骤从该模型中获得最终的离散度估计,这实现了对完全经验贝叶斯处理的计算快速近似。我们首先分别使用每个基因的计数数据来获得初步的基因离散度估计通过最大似然估计。然后,我们拟合离散趋势α tr公司最后,我们将可能性与趋势相结合,以获得最大值后部(MAP)值作为最终分散估计值。以下是三个步骤的详细信息。
基因离散度估计获得基因的基因离散度估计我,我们首先拟合一个负二项式GLM,在设计矩阵之前没有LFCX(X)基因计数数据。该GLM使用基于组内方差和平均值的离散度估计的粗略方法。初始GLM是获得初始拟合值集所必需的,然后,根据拟合值,我们将Cox–Reid调整后的离散可能性最大化从初始拟合开始,得到全基因估计即。,
具有
(7)
哪里(f) 注(k个;μ,α)是具有平均值的负二项分布的概率质量函数μ和分散α,第二项提供了Cox-Reid偏差调整[47]. 这种调整,首次用于SAGE数据的色散估计[48]然后针对HTS数据[三]英寸边缘R,修正了使用拟合值的最大似然估计值得出的色散估计值的负偏差(类似于常用样本方差公式中的贝塞尔修正;有关详细信息,请参见[49],第10.6节)。它由拟合值的Fisher信息构成,此处计算为det(X(X) t吨W公司 X(X)),其中W公司是标准迭代加权最小二乘算法的对角线权重矩阵。由于GLM的链接功能是克(μ)=对数(μ)其方差函数为V(V)(μ;α)=μ+α μ 2,对角矩阵的元素W公司
我
由以下人员提供:
方程中的优化(7)按对数的比例执行α对满足Armijo条件的已接受提案使用回溯线搜索[50].
分散趋势形状的参数曲线(6)通过回归基因离散度估计进行拟合归一化计数的平均值,.围绕真值的基因离散估计的抽样分布α
我
可以是高度扭曲的,因此我们不使用普通的最小二乘回归,而是使用γ族GLM回归。此外,离散异常值可能会扭曲拟合,因此使用了一种排除此类异常值的方案。
超参数一 1和α 0第页,共页(6)通过迭代拟合γ族GLM获得。在每次迭代中,离散度与拟合值之比超出范围[10的基因−4,15]被忽略,直到新系数与旧系数的平方LFC之和小于10−6(与中的方法相同DEXSeq公司[30]).
参数化(6)是基于我们和其他人关于许多数据集中离散度对平均值依赖性降低的报告[三]-[6],[51]. 需要注意的是,要从估计偏差的影响中分离出真正的潜在依赖性,因为估计偏差可能会产生离散度对平均值的感知依赖性。考虑一个负二项分布的期望随机变量μ和分散α.其方差v(v)=μ+α μ 2有两个组件,v(v)=v(v) P(P)+v(v) D类,泊松分量v(v) P(P)=μ独立于α和过分散分量v(v) D类=α μ 2.何时μ很小,μ≲1/α(附加文件中的垂直线1:图S1),泊松分量占主导地位,从某种意义上说,而观测数据提供的关于α因此α当μ≲1/α这导致了偏见的出现。为了简单起见,我们在不考虑尺寸因素影响的情况下陈述了上述论点,秒
j个
,关于的价值μ这是允许的,因为通过构造,我们的尺寸系数的几何平均值接近1,因此,非标准化读取计数的样本平均值,,以及标准化读取计数的平均值,,将大致相同。
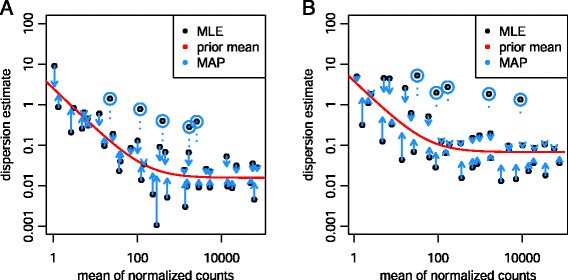
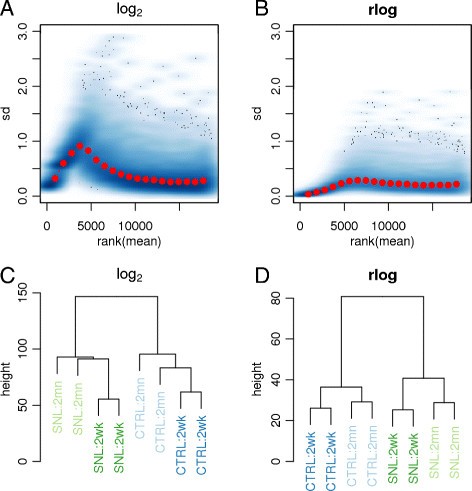
这种现象可能会导致明显的α在μ.弥散平均值的形状可能适合Bottomly数据(图1A) 可以这样解释:渐近色散是α 0≈0.01,平均离散图的非零斜率限制在平均计数的范围内,约为100α 0然而,高估了α在这个低计数范围内,对推断几乎没有影响,因为在这个范围内,方差v(v)无论如何都是由α-独立泊松分量v(v) P(P)Pickrell数据的情况有所不同:这里,对于明显高于渐近弥散倒数的计数,观察到弥散对平均值的依赖性α 0(图1B) ,因此不仅仅是由于估计偏差。仿真(显示在附加文件中1:图S25)证实,观察到的估计分散度和平均值的联合分布与单个恒定分散度不兼容。因此,参数化(6)是一种灵活且适度保守的建模选择:如果存在色散均值依赖性,它能够拾取色散均值依赖性,而在低计数范围内,由于有高估色散的趋势,它可能会导致功率的轻微损失。
色散优先正如吴所观察到的等。[6],对数正态先验拟合了典型RNA-seq数据集的观测弥散分布。我们使用以下参数解决了在处理非共轭数据之前的计算困难:趋势拟合的对数残差,,由两个贡献引起,即趋势周围真实对数离散度的散布,由方差先验给出,以及带方差的离散估计量对数的抽样分布色散估计器的采样分布近似为按比例χ 2分配米−第页自由度,具有米样品数量和第页系数的数量。的对数方差-给出了分布随机变量[52]通过三角函数ψ 1,
因此,也就是说,方差或离散估计量的对数的抽样方差在基因间近似为常数,并且仅取决于模型的自由度。
其他文件1:表S2比较了对数弥散估计方差的近似值与模拟负二项数据的对数Cox–Reid调整弥散估计的方差,在不同样本大小、参数数量和用于创建模拟数据的弥散值的组合上。近似值接近各种典型值的样本方差米,第页和α.
因此,先验方差通过从对数残差方差估计值中减去预期抽样方差得到,:
先验方差将阈值设置为最小值0.25,以便分散估计值不会完全缩小到如果对数残差的方差小于预期的抽样方差。
为了避免通货膨胀由于离散离群值(即该先验没有很好地捕捉到的基因;见下文),我们使用了标准偏差的稳健估计秒 爱尔兰对数残差,
(8)
其中,mad代表绝对偏差中值,通常除以比例因子Φ −1(3/4)。
三个或更少的残余自由度当有三个或更少的剩余自由度(样本数减去要估计的参数数)时,先验方差的估计使用对数残差的观测方差倾向于低估在这种情况下,我们通过模拟来估计先验方差。我们将对数残差的分布与模拟对数残差密度相匹配。这些是的对数-分布式随机变量添加到随机变量,以解释由于先验造成的价差。模拟分布被−log移位(米−第页)以说明χ 2分配。我们在数值网格上重复模拟,并选择从对数残差的观测密度到模拟密度的Kullback–Leibler发散最小化的值。
最终分散估算我们根据Cox–Reid调整后的对数似然形成了离散度的对数后验(7)和对数先验(5)并使用其最大值(即MAP值)作为离散度的最终估计,
(9)
哪里
是先验密度的对数,直到一个加法常数(5). 同样,使用回溯线搜索来执行优化。
分散离群值对于一些基因,基因的估计远超预期假设优先基因适用于该基因是不合理的。如果这些基因的离散估计值朝着拟合的趋势下调,这可能会导致假阳性。因此,如果趋势拟合的残差大于对数残差的两个标准差,我们使用将基因视为离散离群值的启发式方法,秒 爱尔兰(见方程式(8)),即如果
对于这些基因,基因评估不会缩小到趋势前均值。而不是MAP值,我们使用基因估算作为后续步骤中的最终分散值。此外,上述参数分散趋势的迭代拟合过程避免了此类分散离群值影响先验平均值。
对数褶皱变化的收缩率估计
为了合并LFC的经验Bayes收缩,我们假设系数的正态先验为零中心β
红外
模型的(2)代表LFC(即,通常,除截距外的所有系数β 我0):
(10)
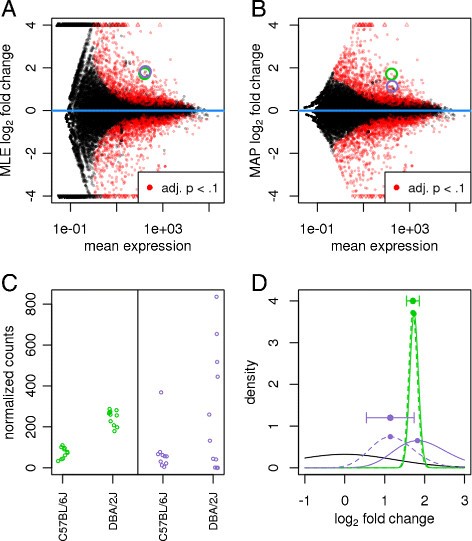
正如使用微阵列的差异表达分析所观察到的那样,具有低强度值的基因往往遭受小的信噪比。可以找到比折叠变化的标准计算更稳定的替代估计值,作为每个条件的平均观测值的比率[53]-[55].DESeq2公司的方法可以看作是这些方法的扩展,用于稳定估计计数数据的基因表达倍数变化。
经验先验估计获取经验先验宽度值σ
第页
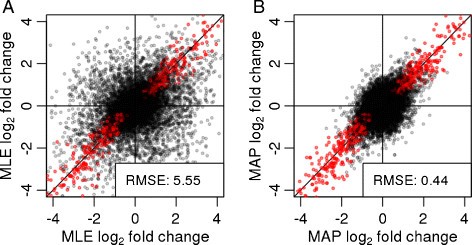
对于模型系数,我们再次近似于完全经验贝叶斯方法,就像离散先验估计一样,尽管这里我们没有从最大似然估计的观测方差中减去预期抽样方差。LFC先验宽度的估计值计算如下。我们使用标准的迭代加权最小二乘算法[12]对于每个基因的模型,方程式(1)和(2),以获得系数的MLE。然后,我们对每一列进行拟合第页设计矩阵(除截距外)的零中心正态分布到MLE折叠变化估计的经验分布.
为了使拟合对具有极高绝对LFC值的异常值具有鲁棒性,我们使用分位数匹配:宽度σ
第页
选择(1−第页)观测到的LFC绝对值的经验分位数,,匹配(1−第页/2) 先验的理论分位数,,其中第页默认设置为0.05。如果我们把正态分布的理论上分位数写成问
N个
(1−第页)MLE LFC的经验上分位数为,则先验宽度计算为:
确保之前的宽度σ
第页
将独立于基准水平的选择,分位数匹配过程中的估计值将在所有可能的因子水平对比中对每个因子进行平均。确定经验上分位数时,LFC极值(,或以2为基数的10)除外。
对数褶皱变化的最终估计向量的对数后验,,属于模型系数β
红外
对于基因我是GLM的对数似然之和(2)和先验密度的对数(10),并且其最大值产生最终MAP系数估计:
哪里
和α
我
是基因的最终离散估计我即。,,除了分散离群值,其中.
术语Λ(β)也就是说,正态先验密度的对数(直到一个加法常数)可以被视为岭惩罚项,因此,我们使用迭代重加权岭回归算法[56],也称为加权更新[57]. 具体来说,给定基因的更新形式如下
具有和
其中当前拟合值根据当前估计值计算在每个迭代中。
费希尔信息。以零为中心的正态先验的影响可以理解为基于实验为该系数提供的信息量收缩MAP LFC估计,我们在此简要阐述这一点。具体来说,对于给定的基因我,LFC的收缩率β
红外
取决于观察到的Fisher信息,由提供
哪里是似然的对数,对LFC进行偏导数β
红外
对于负二项式GLM,观察到的Fisher信息或轮廓似然对数的峰值性受到许多因素的影响,包括自由度、估计的平均计数μ
ij公司
和基因的离散估计α
我
当似然密度和先验密度相乘以计算后验时,先验影响MAP估计。具有低估计平均值的基因μ
ij公司
或高分散性估计α
我
与剩余自由度很少的数据集一样,具有更平坦的轮廓可能性,因此在这些情况下,以零为中心的先验将MAP估计从高不确定性MLE拉近到零。
沃尔德试验
Wald测试比较了β估计值β
红外
除以其估计标准误差SE(β
红外
)达到标准正态分布。估计的标准误差是估计协方差矩阵对角元素的平方根,Σ
我
,对于系数,即。,。通过使用(三)和(4). 对于系数具有正态先验的GLM,我们使用以下公式计算系数协方差矩阵[56], [58]:
将标准正态分布的尾部积分乘以2,以实现双尾检验。Wald测试P(P)使用Benjamini和Hochberg程序对通过独立筛选步骤的基因子集的值进行调整,以进行多次测试[21].
独立过滤
只要测试统计量的分布与过滤器统计量有一定的独立性,独立过滤就不会影响I类错误控制在零假设下[22],我们在下面论证,这就是我们应用程序中的情况。中的筛选器统计信息DESeq2公司是基因归一化计数的平均值,而测试统计是第页,的P(P)Wald测试的值。我们首先考虑这样一种情况,即大小因子相等,并且对每个基因使用基因离散估计,即没有离散收缩。负二项分布族的参数化为θ=(μ,α). 除了离散性第页由于计数低,对于给定的μ,分布第页在零假设下是一致的(0,1),所以第页是一个辅助统计数据。基因计数的样本平均值我,,有界完全足以μ然后根据巴苏定理,和第页都是独立的。
而对于非常低的计数,可以观察到第页在零假设下,DESeq2公司不使用第页在其估算过程中-例如,DESeq2公司不使用以下分布估计无效基因的比例第页–所以这种依赖第页在μ不会导致I型错误增加。
如果样本中的尺寸系数不相等,但与条件无关,则以平均值为条件归一化的计数还应提供均匀分布第页就像对计数平均值的调节一样,我们可以考虑一种病理情况,其中大小因子与条件完全混淆,在这种情况下,即使在无效假设下,平均数低的基因也会有不均匀的分布第页,因为一种情况可能有正计数,而另一种情况通常为零计数。这可能导致第页在零假设下;然而,这种病理情况会给许多平均值差异的统计检验带来问题。
我们通过仿真证明,对于色散收缩,测试统计量的零分布与滤波器统计量的独立性仍然成立。附加文件1:图S26显示了第页在平均归一化计数范围内。尽管由于数据的离散性,平均数最低的基因分布出现峰值,但这些密度在平均表达强度范围内几乎是一致的。
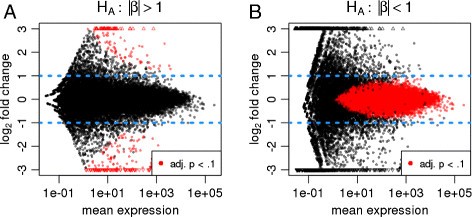
复合零假设
DESeq2公司为形式的复合零假设提供测试寻找LFC显著超过阈值的基因θ>0.复合零假设被两个简单的零假设取代:和.双尾P(P)值是通过对以为中心的正态分布进行积分而生成的θ标准偏差SE(β
红外
)来自|β
红外
|朝向∞然后将积分的值乘以2,并以1为阈值。此程序控制I类错误,即使在β
红外
=±θ,与标准相当DESeq2公司 P(P)值,当θ=0.
相反,当搜索绝对LFC显著低于阈值的基因时,即当测试无效假设时,的P(P)值被构造为简单零假设的两个单侧测试的最大值:和.片面P(P)值是通过对以θ标准偏差SE(β
红外
)来自β
红外
朝向−∞,并积分以−为中心的正态分布θ标准偏差SE(β
红外
)来自β
红外
朝向∞.
请注意,虽然LFC上的以零为中心的先验与测试小型LFC的零假设是一致的,但在测试大型LFC的无效假设时不应使用它,因为先验会支持替代假设。DESeq2公司要求在测试大型LFC的零假设时没有使用先验信息,因此数据本身必须提供证据来反对零假设。
互动
默认情况下的两个例外DESeq2公司LFC估计步骤用于带有交互项的实验设计。首先,当设计中包含任何交互项时,主效应项的LFC先验宽度不是从数据中估计的,而是设置为一个宽值(,或以2为基数的1000)。这确保了主效应项的收缩不会导致对交互作用意义的错误肯定。第二,当包含交互项且所有因素都有两个水平时,则使用标准设计矩阵而不是扩展模型矩阵,以便仅使用一个项来测试无效假设,即两种效应的组合在对数尺度上只是相加的。
正则对数
rlog转换计算如下。实验设计矩阵X(X)用设计矩阵替换,除了截距列外,每个样本都有一个指示变量。方程式中描述的模型(1)和(2)在非干扰项之前使用以零为中心的法线拟合,并使用拟合的离散值,它捕获数据集的总体方差-依赖性。真正的实验设计矩阵X(X)然后仅用于估计所有基因的方差-均值趋势。对于非监督分析,例如样品质量评估,实验设计最好对转换没有影响,因此DESeq2公司默认情况下,忽略设计矩阵,并重新估计将所有样本作为复制品处理的分散度,即使用失明的色散估计。rlog转换值是拟合值,
哪里β
ij公司
是以2为基数的收缩LFCj个第个样本。通过将以零为中心的正态分布与观测LFC相匹配,使用与微分表达式类似的方法设置先验方差。首先,通过取归一化计数的对数(以2为底)加上伪计数来计算LFC矩阵每个样本除以归一化计数平均值加上伪计数。的伪计数允许计算所有基因的对数比,并且对之前或最终rlog变换的方差估计几乎没有影响。然后,LFC矩阵表示每个样本与仅使用截距的拟合值的共刻度对数比。通过将以零为中心的正态分布的97.5%分位数与LFC矩阵中绝对值的95%分位数相匹配,可以找到先验方差。
用于异常值检测的库克距离
的MLE用于计算库克距离。考虑一个基因我和样品j个,库克对GLM的距离由下式给出[59]:
哪里对
ij公司
是样本的皮尔逊残差j个,τ是过度分散参数(在负二项式GLM中,τ设置为1),第页是包括截距在内的参数数量,以及小时
日本
是j个hat矩阵的第个对角元H(H):
皮尔逊残差对
ij公司
计算为
哪里μ
ij公司
由负二项GLM估计,无需LFC先验,并使用方差函数V(V)(μ)=μ+α μ 2.动量估计方法,使用稳健的方差估计为了对异常值提供鲁棒性,这里使用了:
R/生物导体组件
DESeq2公司作为R统计环境的包实现,并且可用[10]作为生物导体项目的一部分[11]. 计数矩阵和元数据,包括基因模型和样本信息,存储在从总结性实验的类基因组范围包装[60].总结性实验使用summarizeOverlaps(摘要重叠)的功能基因组比对包装[61]. 此工作流自动将基因模型存储为元数据以及其他信息,例如基因组和基因注释版本。获取计数矩阵的其他方法包括htseq计数脚本[62]和生物导体包easyRNA序列[63]和功能计数[64].
这个DESeq2公司软件包附带了一个详细的vignette,它通过对真实数据集的一些示例差分表达式分析,以及使用rlog转换进行质量评估和可视化。单个函数,称为DESeq公司,用于运行默认分析,而低级功能也可用于高级用户。
读取底部对齐等。和皮克雷尔等。数据集
使用TopHat2校准仪校准读数[65],并使用summarizeOverlaps(摘要重叠)的功能基因组范围包装[60]. Pickrell的序列读取存档fastq文件等。[17]数据集(登录号[SRA:SRP001540])与智人参考序列GRCh37于2013年3月从Illumina iGenomes下载。读取数计算在Illumina iGenome中包含的Ensembl GTF文件70版定义的基因中。Bottomly的序列读取存档fastq文件等。[16]数据集(登录号[SRA:SRP004777])与小家鼠参考序列NCBIM37于2013年3月从Illumina iGenomes下载。读取数计算在Illumina iGenome中包含的Ensembl GTF文件(版本66)定义的基因中。
可复制代码
在一个包中可以找到用于复制本文中所有图形和表格的Sweave渐晕图,包括用于上述实验的数据对象,以及用于对齐读取和基准测试的代码DESeq2纸张[66].