wwPDB存放政策和wwPDB生物保护程序
B部分:wwPDB生物保护程序
由wwPDB注释人员编写
2024年1月4.4版
目录
前言
1.条目标题、作者和引文信息
2.高分子信息
3.聚合物序列和序列数据库参考分配
4.配体
5.坐标剖面
6.链ID分配
7.标题分配
8.装配
9.其他记录
10.结构基因组学条目
11.X射线结构的特定信息
12.核磁共振结构的特定信息
13.电子显微镜结构的特定信息
14.病毒衣壳和其他复杂组件
15.重新定义另一作者的数据
附录:
A.标题列表
B.结构系数格式
前言
自1999年以来,wwPDB一直负责与RCSB PDB、PDBe和PDBj的沉积中心处理PDB数据。处理的条目遵循符合PDB交换字典(PDBx)的mmCIF格式
https://mmcif.wwpdb.org/dictionaries/mmcif_pdbx_v50.dic/索引/.
PDB文件的版本及其与文件格式指南的对应关系将包含在wwPDB处理和发布的所有文件中。
本文件介绍了当前的处理程序。wwPDB工作人员将继续根据不断发展的结构确定和注释方法更新注释实践。
2008年12月:初始版本为2.2
2009年3月:第2.3次小修订,更新了引文、标题列表、SITE记录,并增加了结构因子的cif模板。
2009年4月:次要修订2.4,更新标题列表。
2011年11月:主要版本2.5,澄清内容,更新标题列表和UniProt序列提交。
2012年12月:澄清条目标题。
2014年1月:澄清重新定义。
2017年8月:澄清核磁共振条目的最佳代表性一致性。
2018年5月:更新生物组件注释程序。
2021年7月:主要修订版4.0,以明确内容并以mmCIF格式提供示例。
2021年11月:为结构测定过程中使用的新配体保留ID。
2022年11月:聚合物链中的肽键需要朝着同一方向。
2023年11月:裂解聚合物链的独特链ID。
2024年1月:链ID长度限制
1.条目标题、作者和引文信息
条目标题(_struct.Title)
条目标题应具有描述性,并与条目内容相匹配。如果标题不明确或与结构内容不匹配,PDB工作人员可以在数据处理期间或条目发布之前修改条目标题。
作者可以选择隐藏有关条目作者和标题的信息,以便在条目发布给公众之前不会出现在PDB网站上。
作者信息(_audit_Author.name)
PDB条目的作者可以与主要引文的作者相同,也可以是引文作者的子集。或者,与引文作者相比,条目中列出的作者可能更多。通常,在主要引文的作者列表中,至少应包括条目的作者之一。
条目的作者由首席研究员(PI)决定。如果有多个PI负责输入,他们需要共同决定作者身份。
引文信息
鼓励作者在出版前交存其作品。主要引文是描述PDB条目中结构的论文。
论文
会议记录和论文只能作为主要引文,但不能作为次要引文。
PubMed ID
PubMed ID可用于PDB、mmCIF和XML文件中条目的主要引用。如果可用,还包括DOI编号。
专利
可以记录专利id(_citation.pdbx_database_id_Patent),日志名称为“Patent”。
未发布的结构
如果作者表示该条目永远不会发布,则将在citation.journal_abbrev记录中添加“未发布”的期刊名称。
年少者。
后缀为“junior”的期刊和条目作者姓名将表示为“Jr.”,而不是“juniro”,例如“Smith Jr.,J.”。
标题大小写
所有标题、名称等均以混合大小写形式包含在mmCIF格式文件中,以匹配文献。
2.高分子信息
如何分配大分子名称和同义词?
蛋白质分子名称
蛋白质分子名称和同义词经过标准化,与相应的UniProt条目保持一致,并列在_entity.pdbx_description和_entity_name_com.name记录中。例外情况如下:
- 如果UniProt名称是指前体,且条目中包含成熟蛋白质,则蛋白质名称中不会包含前体一词。如果作者确实指明了推定,则措辞将保留。
- 在酶原的情况下,如果UniProt名称为“胰蛋白酶原”,但结构中存在活化蛋白,则使用“胰蛋白酶”作为名称。
- 如果UniProt条目包含蛋白质的完整基因序列,该蛋白质被加工成多个链,则相应的多肽名称将作为PDB条目中的名称。例如,如果UniProt名称为胰岛素,则蛋白质名称对应于链名称:例如胰岛素A链和胰岛素B链。
- 对于合成多蛋白的病毒蛋白,以及PDB条目包含所有成分的病毒蛋白,可以使用多蛋白的名称。否则,将使用UniProt中的片段名称。例如,如果“基因组多蛋白”由衣壳、包膜蛋白和主要包膜蛋白组成,但沉积的结构仅为包膜蛋白,则使用的名称为“包膜蛋白”。
- 如果样品中没有蛋白质的完整UniProt序列,则将填充片段部分。但是,片段部分不会填充代表完整成熟蛋白质的区域,例如完整胰蛋白酶分子或包膜蛋白等。
- 如果未指定UniProt名称(即,它是“假设的”),并且作者有蛋白质的名称,则将使用作者的蛋白质名称。
- 如果作者的分子名称与UniProt分子名称不同,则UniProt主名将用于分子名称。作者的分子名称将首先列在同义词列表中,然后是UniProt中提供的同义词。
- 如果没有相应的UniProt条目,将使用作者提供的蛋白质名称。
- 抗体将根据作者提供的信息命名。示例:
- Fab碎片将被命名为Fab重链和Fab轻链。
- 对于免疫球蛋白G链(包括fab和fc区域),蛋白质名称为“抗体重链”和“抗体轻链”
- 如果作者提供了抗体的特定名称,则将使用这些名称,例如Fr62单克隆抗体轻链和Fr62单克隆抗体重链。
- 对于嵌合蛋白质,蛋白质名称以逗号分隔,可能指存在连接子(protein_1,linker,protein_2)。有关嵌合体的其他详细信息可以在entity.details和pdbx_entry_details.sequence_details中提供。
核酸分子名称
对于所有核酸序列,应在可用时使用生物名称。生物名称要么由作者提供,要么从序列数据库中获得,例如“16S核糖体RNA”。
如果序列短于24个核苷酸,并且没有可用的生物名称,则分子名称为短序列:
5'-D(*CP*GP*CP*GP*(8OG)P*AP*TP*TP*CP*GPS*CP*G)-3’
例如,对于等于或大于24个核苷酸的序列,当没有可用的生物名称时,该名称可能被列为“50-mer RNA”。
碳水化合物聚合物
截至2020年7月,我们对含有碳水化合物聚合物的条目适用的规则如下:
(1) 残留物从还原端到非还原端排序。对于常见的糖基化反应,只有一个还原端,非还原端有分支。这就像一棵树,排序总是从根开始(减少端),因为从分支的末端(非减少端)计数是不一致的。
(2) 将最长的分支作为主分支。如果两个枝条的长度相同,则先从下节开始。这是根据1996年国际捕鲸委员会/国际捕鲸生物委员会的建议进行的。有关更多信息,请咨询https://www.wwpdb.org/documentation/carbohydrate-remedition网站
碎片信息是如何表示的?(实体.pdbx碎片)
如果与UniProt条目相比,PDB条目包含序列记录中的蛋白质片段,则可以包含片段名称,例如:N末端结构域、C末端结构域,催化结构域、配体结合结构域等。
如何分配E.C.编号(entity.pdbx_ec)?
E.C.编号自动从UniProt条目中提取。如果UniProt条目没有E.C.编号信息,则将使用作者提供的信息。如果作者不同意UniProt分配的E.C.编号,wwPDB工作人员将联系UniProt工作人员,并试图澄清差异。如果问题无法解决,作者提供的E.C.信息可以添加到实体详细信息中,并显示为“作者提供的C.C.编号为xxxx”。
突变是如何指示的(_entity.pdbx_mutation)?
mmCIF文件中将以以下方式描述确切的突变:Y20K。序列差异记录(struct_ref_seq_dif)将包含注释为“工程化突变”的相应记录。
高分子源生物是如何指示的?
源生物通过其学名和NCBI分类数据库中列出的分类id进行识别。在条目注释期间,通常从基于NCBI分类数据库的UniProt数据库中获取官方学名。如果聚合物分子是自然获得的,则源生物名称和分类id将列在_entity_src_nat mmCIF类别下;如果聚合物分子经过遗传操作(表达),则列在_entity_src_man下;如果是化学合成的,则列于_pdbx_entity_src_syn下。如果NCBI分类数据库中列出了通用名称,则通用名称将映射到_entity_src_gen.gene_src_common_name字段,否则该字段将保留为空。源生物和宿主生物的学名、质粒和基因名将包含在混合大小写中,以匹配标准科学文献。
嵌合蛋白每个部分的源生物体名称列在mmCIF文件中,并带有相应的残留范围。
例如:
回路__实体源代码_实体_源.pdbx _源id_实体源标记_实体_源.pdbx _设备_类型_实体名称_实体rc_gen.pdbx end_seq num_实体src_gen.gene_src_common_name_实体rc_gen.gene_src_gen_实体src基因pdbx基因src基因_实体src_gen.gene_src_species_实体src_gen.gene_src_strain_实体src_gen.gene_src_tissue_实体rc_gen.gene_src_tissue_fraction_实体rc_gen.gene_src_details_实体src_gen.pdbx_gene_src_fragment_实体src_gen.pdbx_gene_src_scientific_name_实体src_gen.pdbx_gene_src_ncbi_taxonomy_id_实体_源.pdbx _源_源_变量_实体src_gen.pdbx_gene_src_cell_line_实体src生成pdbx生成src生成tcc_实体src生成pdbx生成src生成rgan_实体src_gen.pdbx gene_src_organelle_实体src_gen.pdbx生成src_cell_实体src_gen.pdbx_gene_src_cellular_location_实体rc_gen.host_org_common_name_实体_源.pdbx _主机_源_科学名称_实体src_gen.pdbx_host_org_ncbi_taxonomy_id_实体rc_gen.host_org_gen_实体src_gen.pdbx_host_org_gene_实体rc_gen.pdbx主机org_organ_实体rc_gen.host_org_species_实体rc_gen.pdbx主机org_tissue_实体rc_gen.pdbx主机org_tissue_fraction_实体rc_gen.pdbx主机org_strain_实体rc_gen.pdbx主机arg_variant_实体src_gen.pdbx_host_org_cell_line_实体rc_gen.pdbx主机arg_atcc_实体src_gen.pdbx_host_org_culture_collection_实体rc_gen.pdbx主机org_cell_实体src_gen.pdbx主机org_organelle_实体src_gen.pdbx主机org_cellular_location_实体rc_gen.pdbx主机arg_vector_type_实体rc_gen.pdbx主机org_vector_实体rc_gen.host_org_details_实体rc_gen.expression_system_id_实体名称_实体rc_gen.plasmid_details_实体_源.pdbx描述1 1样本“生物序列”1 121肌肉10090??????????????’大肠杆菌'562?1 2样本“生物序列”122 1729606年智人大肠杆菌'562?
如果使用了表达系统,表达系统的科学生物名称也将从NCBI分类数据库中获取,并列在_entity_src_gen.pdbx_host_org_NCBI_Taxonomy_id记录下。
关于源代码和表达式系统的其他信息不是强制性的,只有作者提供了这些信息,才会包含在文件中。
噬菌体显示
_entity_src_gen类别将填充首次分离基因序列的生物体,以及用于生成最终蛋白质的过表达系统的宿主生物体。有关噬菌体展示的其他信息可以映射到_entity_src_gen.pdbx_description,例如:“噬菌体显示选择的蛋白质”。
无细胞合成、体外转录和体外翻译
无细胞合成、体外转录和体外翻译都将被称为“无细胞合成”。目前用于生产无细胞蛋白质的常见细胞提取物由大肠杆菌(ECE)、兔网织红细胞(RRL)、小麦胚芽(WGE)、昆虫细胞(ICE)和酵母Kluyveromyces(D2P系统)制成。所有这些都将有遗传操作源(_entity_src_gen类别),其中包含上述源生物体和相关分类ID。有关合成的其他信息,例如“无细胞合成”可以添加到_entity_src_gen.pdbx_description中。
杆状病毒
如果使用杆状病毒,它将被列在载体类型下。如果提供了一个单元格行,它将被添加到条目中。
合成
术语“合成”是指“使用非生物方法合成”。合成源的分类ID为32630。
未定义
该术语用于无法识别源生物体的情况。未定义源的分类ID为32644。
3.聚合物序列和序列数据库参考分配
聚合物序列(entity_poly,SEQRES)的定义是什么?
聚合物序列是以线性方式共价连接以形成聚合物的连续化学成分的列表。本清单中包含的化学成分可以是标准或改性氨基酸或核酸残基。它还可以包括与聚合物中的标准主链相连的其他残基。与多肽侧链共价连接的化学成分或基团或与核酸聚合物中的糖和/或碱连接的基团将不在此处列出。
含有三个或三个以上残基的蛋白质,形成两个连续的标准肽键,在同一方向将被分配聚合物序列记录(分子名称、来源、序列、序列数据库参考)。
含有两个或多个由标准核苷酸键连接的残基的核酸将被分配给聚合物序列记录(分子名称、来源、序列、序列数据库参考)。
聚合物序列记录必须表示实验中使用的每个样品的完整序列,包括任何表达标签和未在坐标中建模的残基(例如,由于局部无序而未观察到的区域)。在实验之前或期间从聚合物序列中裂解的残留物不被视为序列的一部分。聚合物序列可以包括相邻的交联残基(例如发色团)和修饰的氨基酸。
如果样品的确切序列未知怎么办?
如果由于蛋白质水解等原因,样品的确切序列未知,则序列应与坐标匹配,并应添加带有解释的专用序列注释(_pdbx_entry_details.sequence_details,remark 999)。在这种情况下,如果条目是晶体结构,马修系数和溶剂含量将列出作者提供的值,而不是计算值。
序列数据库引用(_struct_ref、_struct_ref_seq、DBREF)是什么?
序列数据库引用提供了聚合物序列和有效序列数据库引用之间的映射。
哪些聚合物链被分配了序列数据库记录?
每个有适当序列数据库匹配的蛋白质或核酸链将列出序列数据库条目的交叉引用。当没有序列数据库引用可用时,序列将被自我引用(即,数据库引用将是PDB条目本身)。
将使用哪些序列数据库?
- 蛋白质
UniProt是目前首选的蛋白质序列数据库。如果来自同一生物体、菌株和序列标识的同一蛋白质有多个UniProt条目,则将使用注释最多的UniProt条目的条目。此外,包含完整蛋白质序列的UniProt条目将优先于表示蛋白质片段的条目。
- 核酸
自然获得的核酸序列可以参考GenBank。
- 非核糖体肽
NORINE数据库用于一些序列数据库引用。NORINE的引用目前已停止。使用UniProt参考(如果可用)或自我参考。
- DNA和RNA
所有DNA都将自我参照。RNA聚合物长度超过50个残基,如果可用,将提供GenBank序列参考。
如果UniProt参考不适用于蛋白质序列怎么办?
UniProt不包含免疫系统的可变或超可变区域或非自然序列,因此此类结构的PDB条目将自我引用。如果蛋白质不属于这些类别并且没有UniProt引用,UniProt会自动从PDB获取序列并将其添加到UniProt。
如果蛋白质序列的UniProt参考已经可用或在UniProt数据库中发生了更改,该怎么办?
PDB条目包含处理条目时可用的序列交叉引用。如果将来某个时候序列确实出现在UniProt中,或者UniProt登录代码已更改,则用户可以通过SIFTS项目获得序列数据库匹配的更新版本。不得修改PDB条目以添加/更新序列交叉引用。有关SIFTS的信息,请访问https://www.ebi.ac.uk/pdbe/docs/sifts/
如果具有相同序列的相关条目没有UniProt引用,则序列引用将引用每个PDB条目本身,而不是引用包含特定序列的第一个PDB条目。例如,如果条目1ABC、1DEF和1GHI都具有相同的抗体序列,则这些条目将分别指1ABC,1DEF与1GHI。
嵌合体是如何处理的?
嵌合体或融合蛋白应该以单链的形式存放,并带有一个链ID,因为它们是以单链形式表达的。用不同链ID精炼的嵌合体应该为嵌合体的所有部分(包括任何链接器区域)保存一个链ID。与UniProt条目匹配的嵌合体部分都有数据库引用。嵌合体中与UniProt条目不匹配的部分将自行参考。
序列参考差异是什么(_struct_ref_seq_dif,SEQADV)?
序列差异记录描述了序列数据库和PDB条目中序列之间的任何合理不一致。
不同类型的序列差异是什么?它们是如何注释的?
- 工程突变:
PDB序列和UniProt条目之间的差异将被列为“工程突变”。
- 克隆项目:
术语克隆伪影是为引入序列差异的情况而保留的,例如在克隆期间的PCR实验中或通过随机突变。这种情况很少见。
- 改性残留物:
此处仅列出修改后残基的父代与序列数据库引用不匹配的实例。例如,如果在UniProt条目中列出了THR,并且PDB残基是MSE(硒代蛋氨酸),那么将生成MSE/THR的struct_ref_seq_dif(SEQADV)记录,并解释为“工程突变”,所有其他修改残基的情况将列在pdbx_struct_mod_residue(MODRES)记录下。
- 微观异质性:
请参阅下面的“如何处理微观异质性/多态性?”一节。
- 发色团:
参见下文“相邻的链内交联基团”一节。
- 冲突:
聚合物序列中的残基与UniProt参考文献中列出的残基之间的序列冲突(无法解释)将在此处列出,并解释“冲突”,还可能提供其他序列细节。
- 表达式标记:
额外的N-和/或C-末端残基,包括先导序列、His-tags、其他类型的标记和/或起始蛋氨酸。
- 插入:
位于不属于UniProt序列的序列中间
- 删除:
其中,具有UniProt参考的序列中间的蛋白质结构被删除。
- 链接器:
样本序列中连接嵌合实体分区的区域。
没有列出哪些序列差异?
- 没有UniProt参考(自我参考)的聚合物。
- 母体残留物与数据库参考相匹配的修改残留物。
如果残留物来自母体残留物,则将为修改后的残留物创建MODRES记录(_pdbx_struct_mod_residue)。例如,当UniProt条目中列出Met时,MSE(硒代蛋氨酸)只有pdbx_struct_mod_residue(MODRES)条目。
- D-氨基酸
如何处理微观异质性/多态性?
具有相同序列的聚合物链必须具有相同的化学性质(均质)。如果一条链在一个位置具有微异质性,但在同一位置的另一条链中没有,则这两条聚合物链将被视为具有两个序列的两个不同聚合物实体。如果一个残基在链中的特定位置具有多个同一性,这称为微观异质性。
对于异质链,与相应UniProt残基不匹配的残基将在序列中列出,而不管其占有率如何。显示微观异质性的残留物的不同身份的总占用量应小于或等于1。
如果在相应的序列位置没有任何残基标识与UniProt残基匹配,则具有较高占用率的残基将列在序列中。UniProt序列和聚合物序列之间的差异将生成序列差异记录,并解释“微观异质性”。如果没有UniProt参考,则较高的占用剩余量将列在序列中,并且不会生成序列差异记录。
如果一个残基在一个特定的残基编号上有两个身份,其中一个身份是修饰残基,另一个身份为未修饰形式,那么修饰残基将被列在聚合物序列中,并在序列差异类别中生成“微异质性”记录。这是一个例外,即不会为修改后的残留物创建序列差异记录。
替代位置指示器将用于微异质性残余物的坐标。聚合物序列中列出的残基将首先列在坐标中,并标记为替代位置标识符A。另一个标识将被指定为替代位置标识B。如果存在第三个或第四个残基标识,或者其中一个标识具有自己的替代构象,这些将按字母顺序分配备用ID。
在mmCIF文件中,微异质性涉及的所有残基标识和微异质性标记将列在_entity_poly_seq.hetro和_poly_seq_scheme.hetro中。
示例(PDB条目5ZA2):
回路__实体_多边形_ eq.实体_id_实体_ poly _ seq.num_实体_ poly _ seq _ mon_id_实体_多元_异质...2 56 ILE n 2 57 GLY n 2 58 9月2 58序列号2 59 VAL n 2 60序列号...回路__pdbx_poly_seq_scheme.asym_id_pdbx _ poly _ seq _方案实体id_pdbx _ poly _ seq _方案seq _id_pdbx poly_seq方案mon_id_pdbx poly_seq方案ndb_seq编号_pdbx poly_seq方案pdb_seq编号_pdbx _ poly _ seq _ scheme授权_ seq _num_pdbx_poly_seq_scheme.pdb_mon_id_pdbx授权模式_pdbx poly_seq方案pdb_strand_id_pdbx_poly_seq_scheme.pdb_ins_code(pdbx_poly_seq_scheme.pdb_ins_code)_pdbx _ poly _ seq _ scheme杂波...B 2 56 ILE 56 62 ILE B号。n个B 2 57 GLY 57 63 63 GLY乙。n个B 2 58 9月58日64 9月64日9月B日。年B 2 58 SER 58 64 64 SER B系列。年B 2 59 VAL 59 65 65 VAL B(阀B)。n个B 2 60 SER 60 66 66 SER B系列。n个...
对于特定残基数下所有残基的微观异质性的完整解释,可以通过附加的序列细节文本注释进行进一步阐述。
相邻的链内交联基团
本节描述了相邻的链内交联的情况,通常涉及2个或多个相互反应形成一个“残基”的序列氨基酸。例如,PDB条目1yjf中的残基65、66和67发生反应,形成循环三肽发色团(图1)。
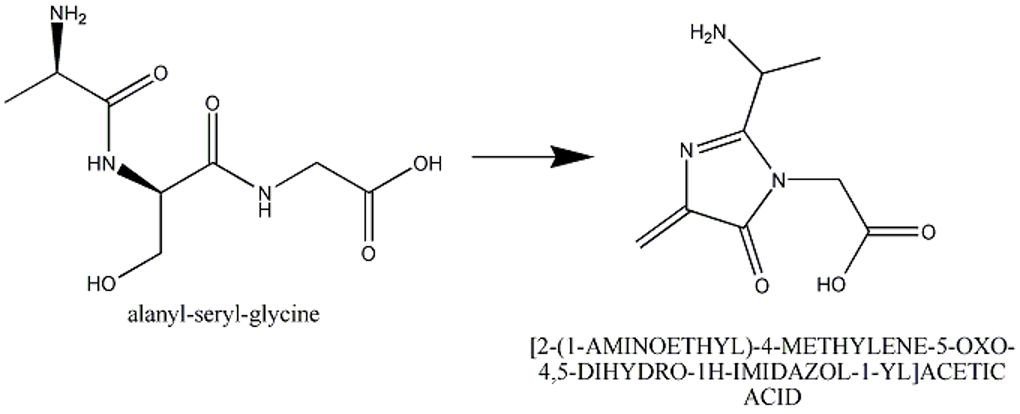
图1:在条目1yjf中,残基65、66和67(相邻的丙氨酸、丝氨酸和甘氨酸残基)发生反应并生成一种称为[2-(1-氨基乙基)-4-甲基-5-OXO-4,5-二氢-1H-咪唑-1-YL]乙酸的生色团产物。
要在PDB条目中描述此情况,请使用以下注释:
- 相邻的链内交联基团将在坐标和序列中列为一个化学基团。
- 如果三个氨基酸参与反应形成发色团,相邻的链内交联基团将有三个亲本残基。因此,将有三条pdbx_struct_mod_residue(MODRES)记录。
- 将生成解释为“生色团”的struct_ref_seq_dif(SEQADV)记录。任何其他信息都可以添加到pdbx_entry_details.sequence_details(REMARK 999)。
4.配体
wwPDB鼓励储户提供InChI或SMILES字符串和/或化学名称和/或化学品图纸,包括配体的键类型、键顺序和立体化学,以便于正确注释。
配体的身份是如何验证的?
使用专门的软件获取键类型、立体化学,并在可能的情况下获取结构中每个配体的IUPAC兼容名称。通过将坐标中的配体结构与wwPDB化学成分词典(CCD)中定义的配体进行比较来进行验证。当在CCD中发现配体匹配时,坐标中的原子名称将与字典定义匹配。
配体身份是如何分配的?
每个成分(配体、氨基酸、核酸)在CCD中分配一个唯一的代码。在策展期间,解说员使用多种方法将组件与CCD进行比较。如果立体化学、键类型和连接性与CCD中的现有定义相匹配,则将现有定义指定给配体。如果没有匹配项,则使用任意分配的唯一标识符向CCD添加新的CCD定义。
如何将新配体添加到化学成分词典中?
为了指定新的配体,我们鼓励在结构测定过程中使用以下id:DRG、LIG、INH、00-99(2位数字)。这些id不包括在CCD中,而是保留用于CCD新配体的初始沉积。使用几个软件程序根据作者存放的坐标生成CCD定义。组件CIF文件中的所有原子(包括氢原子)都有坐标。如果坐标中有原子因无序而丢失,它们将被添加到组件CIF文件中。将为整个分子生成一组理想坐标和符合IUPAC的化学名称。对化学名称、配体代码和连通性进行检查,以验证CCD中没有添加重复项。
如果一个结构包含一个已经存在于化学成分词典中的配体,会发生什么?
如果结构中的特定配体已经在CCD中定义,配体3个字母的标识符、化学名称和原子命名将在条目注释期间根据字典进行更新。注意,原子名称总是以元素类型开头。
肽样抑制剂是否被视为配体?
如果肽样抑制剂是一种天然产物,或者如果它有序列数据库参考,或者如果大多数残留成分是标准氨基酸或修饰氨基酸,则将其视为聚合物。否则,该分子被视为配体。
充电状态
只要可能,新配体定义的总电荷应为中性。这提供了与其他化学数据库(如CAS和PubChem)的最大兼容性。配体的总电荷包含在CCD中。单个原子在原子记录中可能有电荷。
例外情况:
- 四个键合伙伴都是重原子的四价氮原子必须携带正电荷才能满足价态规则
- 硝基(R-NO2)以电荷分离状态(R-[N+](=O)[O-])表示,以更好地满足价规则。
- 金属有机化合物
核磁共振结构中的配体产生了独特的挑战,因为结构中使用的所有氢原子都必须出现在字典中。对于可能存在于多质子化状态的分子,在单一化学成分定义中很难实现精确的化学描述。为了描述标准氨基酸和核苷酸的复杂性,创建了一个特殊的成分字典(https://www.wwpdb.org/data/ccd). 在这本词典中是对观测到的质子化状态的完整化学描述,其中包括每种情况下的精确公式、形式电荷和IUPAC原子命名法。
配体名称和同义词是如何分配的?
_化学成分名称(HETNAM)只要有可能,就会使用专门的化学命名软件自动生成的名称。这一规则的例外情况是常见的(由注释人员判断)生物名称和药物品牌名称。如果ACDlabs、Chemdraw或PubChem无法预测名称,则可以使用普通名称或存款人提供的名称。
_chem_comp.pdbx同义词,_pdbx_chem_comp_synonyms(HETSYN)储户要求的其他名称可以作为同义词(_pdbx_chem_comp_synonyms)包括在内,由注释者自行决定。值得注意的是,存款人提供的任何同义词都应该是配体的有意义且广泛使用的名称。
例外/问题:只要将符合IUPAC的名称列为同义词名称,就可以将已建立的通用名称用作分子名称。例如,“氟康唑”可用作分子名称,“2-(2,4-二氟苯基)-1,3-双(1,2,4-三唑-1-基)丙醇”可用作同义词。如果更改了配体名称或添加了同义词,则包含该配体的每个PDB条目都将更新。
金属在配位络合物中处理与格键和有机金属络合物中的π键存在困难。这些无机分子需要逐一构建,以反映其化学性质。在当前化学信息软件不充分或无法描述真实化学结构的情况下,此类分子的化学成分文件中包含一个模糊标志(_chem_comp.pdbx_ambigous_flag)。
许多被定义为“与多种水结合的金属”的配体已被标记为过时(OBS状态代码),这意味着它们不再是有效的配体,今后不应使用。虽然水配位金属可以被强烈结合,但这些基团并没有在所有结构中一致使用,用户分析将受益于标准化单离子表示。更新了含有此类配体的条目,将其拆分为金属和水分子。
离开基团:为了准确地表示化学成分词典中条目的聚合潜力,所有在连接过程中可能从配体中丢失的原子都将用一个特殊的标记标记,表示这些原子是“离开原子”。例如,标准氨基酸具有羰基氧原子OXT,并且N末端氢HN2总是被标记为离开原子,因为这些原子在形成多肽链的过程中丢失。然而,这并不意味着这些原子总是从坐标中消失,而是这些原子总是以聚合状态消失,这取决于化学实体的位置和键状态。
缺失的配体部分:配体被定义为实验中存在所有原子。如果在实验中只看到部分配体(例如,晶体结构的密度中缺少一个环),则使用的配体代码是完全定义的配体。如果配体是新的,缺失的原子将添加到配体的定义中。
改性氨基酸和核苷酸:如果一个氨基酸或核苷酸被大于10个原子的化学基团修饰,则残基将分为两组:氨基酸/核苷酸基团和修饰。将在氨基酸/核苷酸基团和修饰物之间生成链接记录。改性氨基酸和核苷酸将遵循标准的原子命名法。
核苷酸残基标识符:标准核苷酸使用以下DNA残基标识符表示:DA、DG、DC、DT和以下RNA残基标识符:A、G、C、U。
UNX/UNL/UNK的使用有时氨基酸残基、核苷酸、原子或配体无法识别。在下列情况下应使用这些配体代码:
- UNX:未知原子或离子
- UNL:未知配体
- UNK:未知氨基酸
- N/DN:未知核苷酸
联合国X当作者不知道原子或离子的身份时,UNX是一个原子或离子本身的代码。注:配体名称为UNX,但原子名称为UNK。原子类型为“X”。
UNL(解锁)UNL是未知配体的代码。这是因为作者在坐标中添加了原子以满足电子密度,但真正的配体身份尚不清楚。例如,请参见PDB条目3MHO。
UNK公司UNK是未知氨基酸的代码。例如,如果作者不知道坐标如何与序列对齐,并且残数编号是任意的,那么poly-ALA或poly-GLY链将被处理为poly-UNK。序列将是poly-UNK,坐标中的残基将列为UNK。如果序列已知,可以在序列详细信息部分列出。如果作者确实知道序列和坐标的对齐,则应更改poly-ALA或poly-GLY残基以匹配序列。UNK的原子名为N、CA、CB、CG、O、C,原子类型为N、C、C、O、C。
无DN是未观察到碱基的未知核苷酸的代码。
如果存款人提供的配体标识与配体的软件和注释器标识冲突怎么办?
作者对配体的识别与配体的软件和/或注释器识别之间的冲突引起了作者的注意。如果寄存人和wwPDB工作人员之间存在分歧,配体名称将基于专业化学命名软件从坐标中得出的内容。如果立体化学不能由程序确定,则使用作者对立体化学的描述。对于不明确的立体化学或键长,可能会在mmCIF文件中添加_pdbx_entry_details.nonpolymer_details(REMARK 600)和标志。
生物有趣分子参考词典(BIRD)
生物有趣分子参考词典(BIRD)包含PDB档案中有关生物有趣的肽类抗生素和抑制剂分子的信息。
BIRD是一个外部参考文件(类似于CCD),提供有关这些分子的化学、生物和结构的信息。
BIRD条目包括分子量和分子式、聚合物序列和连接性、结构特征和功能分类描述、天然来源(如有)以及对应UniProt或Norine条目的外部参考。
整个BIRD资源可以从wwPDB FTP区域下载:
定期审查BIRD的一致性和准确性,并用于统一注释包含这些分子的PDB条目。随着相应的PDB条目在PDB存档中发布,词典每周都会更新新的定义。
相应的BIRD ID代码仅出现在条目的PDBx格式文件中。有关BIRD注释的其他详细信息,请访问:https://www.wwpdb.org/data/bird
5.坐标剖面
交替构象
如何处理单个原子、侧链或整个残基的交替构象?
有时一个原子、几个原子或整个侧链具有多个构象。原子的每组坐标被指定为交替位置A和B,或者如果有三个交替位置A、B和C等,则交替位置的组合占用率不得超过1.00。通常,替代构造A应具有较高的占用率。
例子
回路__原子_位置_分组_ PDB_atom_site.id_原子站点类型符号_原子站点标签原子id_原子站点标签alt_id_原子站点标签comp_id_原子站点标签异步id_原子站点标签实体id_原子站点标签序列id_原子_位置.pdbx_PDB_ins_code_atom_site。Cartn_x公司_atom_site。Cartn_y(_y)_atom_site。Cartn_z公司_atom_site占用率_atom_site。B_同等或同等_atom_site.pdbx正常充电_atom_site.auth_seq_id_atom_site.auth_comp_id_atom_site.auth_asym_id_atom_site.auth_atom_id_atom_site.pdbx_PDB_型号_num...原子1179 C CA A GLU A 175?3.108 5.119 18.896 0.58 12.70 ? 167 GLU A CA 1号机组原子1180 C CA B GLU A 175?3.163 5.208 18.892 0.42 12.51 ? 167 GLU A CA 1号机组…
如何处理化学基团的交替构象?
化学基团交替构象的处理方式与氨基酸相同,除非交替构象涉及两个不同的化学基团。
如果两个不同的化学基团处于交替构象中怎么办?
作者可能会说,两个化学基团是彼此交替的构象,但具有不同的身份。例如,作者可能会说,在特定位置的化学基团是锌和铜。与多态残基不同,配体不能分配相同的残基数;必须指定不同的残数。在本例中,锌为残渣100,铜为残渣101。锌将被分配交替构象A和铜将被指定为交替构象B。占有率较高的配体通常被指定为替代构象A,但并不总是这样。每个配体的占有率应小于1.00,组合的应小于或等于1.00。
例子
回路__原子_位置_分组_ PDB_atom_site.id_原子站点类型符号_原子站点标签原子id_原子站点标签alt_id_原子站点标签comp_id_原子站点标签异步id_原子站点标签实体id_原子站点标签序列id_原子_位置.pdbx_PDB_ins_code_atom_site。Cartn_x公司_atom_site。Cartn_y(_y)_atom_site。Cartn_z公司_atom_site占用率_atom_site。B_同等或同等_atom_site.pdbx正常充电_atom_site.auth_seq_id_atom_site.auth_comp_id_atom_site.auth_asym_id_atom_site.auth_atom_id_atom_site.pdbx_PDB_型号_num...HETATM 2239 C C A CO2 C 3-6.625 1.156 17.412 0.33 6.76 ? 301二氧化碳A C 1大气中二氧化碳含量2240 O O1-7.570 1.048 16.676 0.33 7.80 ? 301二氧化碳A O1 1HETATM 2241 O O2 A CO2 C 3-5.717 1.284 18.182 0.33 7.89 ? 301二氧化碳A氧气1赫塔姆2242 C C B BCT D 4-5.601 0.708 16.643 0.67 9.63 ? 公元前303年甲丙1赫塔姆2243 O O1 B BCT D 4-6.583 1.348 16.099 0.67 12.22 ? 公元前303年A O1 1HETATM 2244 O O2 B BCT D 4-5.469 -0.535 16.378 0.67 9.77 ? 303 BCT A氧气1HETATM 2245 O O3 B BCT D 4-4.819 1.444 17.442 0.67 12.28 ? 303 BCT A O3 1号机组…
不应包括终端原子的处理
末端原子(蛋白质的OXT、HXT、H1、H2和H3原子,核酸的OP3、HOP3、HO3和HO5原子)不应包括在非序列末端残基的残基中,因为这不能描述晶体的真实含量,而且在化学上是不正确的。如果链中存在此类原子,wwPDB将删除它们。
缺少残留物(_pdbx_unobas_or_zero_occ_residues,备注465)和原子(_pdbx _unobas或_zero_ occ_atoms,备注470)
如果在_pdbx_unobas_or_zero_occ_residues中列出聚合物组分的缺失残基(例如:在N-或C-末端或柔性环中)和缺失侧链原子,则在REMARK 465中列出,并带有polymer_flag(Y)和occupancy_flag(1)。
类似地,如果在_pdbx_unobas_or_zero_occ_atoms中列出缺少的原子,则在REMARK 470中列出这些原子,并带有polymer_flag(Y)和occupancy_flags(1)。离开聚合物键(氨基酸中的OXT、核酸中的OP3、糖中的O1)和氢等原子的原子不会被列为缺失。
例子
对于缺失的残留物
回路__pdbx_unobas或_zero_cc_residues.id_pdbx_unobas或_zero_cc_resideues。PDB_型号_编号_pdbx_unobas或_zero_cc_residues.polymer_flag_pdbx_unobas_或_zero_occ_residues.occupancy_flag_pdbx_unobas_or_zero_occ_residues.auth_asym_id_pdbx_unobas或_zero_cc_residues.auth_comp_id_pdbx_unobas_or_zero_occ_residues.auth_seq_id_pdbx_unobas或_zero_cc_resideues。PDB_输入代码_pdbx_unobas或_zero_cc_residues.label_asym_id_pdbx_unobs_or_zero_occ_sidues.label_comp_id_pdbx_unobas或_zero_cc_residues.label_seq_id11年1月1日?一名MET 12 1 Y 1 A ARG 2?A ARG 2号机组
对于缺失的原子
回路__pdbx_unobas_or_zero_occ_atoms.id_pdbx_unobas或_zero_cc_atoms。PDB_型号_编号_pdbx_unobas或_zero_cc_atoms.polymer_flag_pdbx_unobas或_zero_occ_atoms.occupancy_flag_pdbx_unobas_or_zero_occ_atoms.auth_asym_id_pdbx_unobs_or_zero_occ_atoms.auth_comp_id_pdbx_unobs_or_zero_occ_atoms.auth_seq_id_pdbx_unobas或_zero_cc_atoms。PDB_输入代码_pdbx_unobas_or_zero_occ_atoms.auth_atom_id_pdbx_unobas_or_zero_occ_atoms.label_alt_id_pdbx_unobas_or_zero_occ_atoms.label_asym_id_pdbx_unobas或_zero_cc_atoms.label_comp_id_pdbx_unobas_or_zero_occ_atoms.label_seq_id_pdbx_unobas_or_zero_occ_atoms.label_atom_id1 1 Y 1 B ILE 137?CG1?B文件137 CG12 1 Y 1 B ILE 137?CG2?B ILE 137 CG2号机组3 1 Y 1 B ILE 137?CD1?B ILE 137 CD1号机组
零入住剩余
一些精化程序允许在坐标文件中包含缺失的残基和侧链原子,作为占据0.00的原子。由于这些原子在精炼过程中通常被忽略,因此它们的位置和性质可能不可靠。因此,如果这些原子包含在存放的坐标文件中,这些原子将保留在文件中,但也会在单独的备注中列出:如果在_pdbx_unobas_or_zero_occ_residues中列出,则备注475如果pdbx_unobas_or_zero_occ_atoms中列出了polymer_flag(Y)和occupancy_flags(0),则备注480。离开聚合物键(氨基酸中的OXT、核酸中的OP3、糖中的O1)和氢等原子的原子不会被列为缺失。
对于零占用残留物:
回路__pdbx_unobas或_zero_cc_residues.id_pdbx_unobas或_zero_cc_resideues。PDB_型号_编号_pdbx_unobas或_zero_cc_residues.polymer_flag_pdbx_unobas_或_zero_occ_residues.occupancy_flag_pdbx_unobas_or_zero_occ_residues.auth_asym_id_pdbx_unobas或_zero_cc_residues.auth_comp_id_pdbx_unobas_or_zero_occ_residues.auth_seq_id_pdbx_unobas或_zero_cc_resideues。PDB_输入代码_pdbx_unobas或_zero_cc_residues.label_asym_id_pdbx_unobas或_zero_cc_residues.label_comp_id_pdbx_unobas或_zero_cc_residues.label_seq_id1 1 Y 0专业38?专业人士12 1 Y 0 A HIS 39?A HIS 2
对于零占有原子:
回路__pdbx_unobas_or_zero_occ_atoms.id_pdbx_unobas或_zero_cc_atoms。PDB_型号_编号_pdbx_unobas或_zero_cc_atoms.polymer_flag_pdbx_unobas或_zero_occ_atoms.occupancy_flag_pdbx_unobas_or_zero_occ_atoms.auth_asym_id_pdbx_unobs_or_zero_occ_atoms.auth_comp_id_pdbx_unobs_or_zero_occ_atoms.auth_seq_id_pdbx_unobas或_zero_cc_atoms。PDB_输入代码_pdbx_unobas_or_zero_occ_atoms.auth_atom_id_pdbx_unobas_or_zero_occ_atoms.label_alt_id_pdbx_unobas_or_zero_occ_atoms.label_asym_id_pdbx_unobas或_zero_cc_atoms.label_comp_id_pdbx_unobas_or_zero_occ_atoms.label_seq_id_pdbx_unobas_or_zero_occ_atoms.label_atom_id1 1 Y 1 A GLN 14?重心?一个GLN 16 CG2 1 Y 1 A GLN 14?CD?GLN 16光盘3 1 Y 1 A GLN 14?OE1?一个GLN 16 OE14 1 Y 1 A GLN 14?NE2?A GLN 16 NE2
结构中不属于任何聚合物(蛋白质或核酸)的配体或杂原子基团也可能缺少原子或原子占据率为零。在这种情况下,杂原子基团或配体的名称、链ID和型号(如果适用)将列在具有polymer_flag(N)和occupancy_flag1)的_pdbx_unobas_or_zero_occ_atoms中,或者如果pdbx_unbos_or_zero_occ_atoms中具有0.00个占有率的原子具有polymer_flagN和occuancy_flang(0)。
6.链ID分配
链ID的定义
链ID是每个大分子聚合物和与其相关的所有化学基团(包括水)的唯一标识符。可接受的长度最多为四个字符。
哪些部分被分配了链ID?
PDB条目坐标部分中的所有原子都将被分配一个链ID。
如何分配链ID?
每个聚合物(氨基酸、核酸或2个或多个共价键残基的碳水化合物聚合物)都分配了一个唯一的链ID。所有结合部分和水的链ID都是根据它们与最近聚合物的接近程度(接触次数)分配的。对于例如,蛋白质/DNA/RNA聚合物周围的所有水和化学基团都被指定为其周围聚合物的链ID。如果聚合物链在实验之前或期间被裂解,则应为聚合物的每个片段分配一个唯一的链ID。
为什么链ID是这样分配的?
wwPDB制定了这一规则,以提高结构数据的可用性和解释能力。为与聚合物相关的所有部分指定相同的链ID可以快速、统一地识别特征分析。
如何将链ID分配给化学组和水?
聚合物链中任何原子的5埃范围内的所有化学基团和水都将与该链相关联,并将被赋予与该聚合物相同的链ID。如果一个特定的化学基团/水与多个链等距,则链ID被随机分配为这些聚合物中任何一种的链ID。在进入过程中,距离聚合物5埃以上的水将自动移动,这些水可以通过对称方式移动到5埃以内。距离任何聚合物超过5埃的水,如果不能通过应用对称性使其更接近聚合物链,则应提请存款人注意。这些水将被列为_pdbx_distant_solvent_atoms-cif类别,并与最近的大分子保持距离。
回路__pdbx驻留溶剂原子.id_pdbx杂质溶剂原子。PDB_型号_编号_pdbx驻留解决方案原子身份验证原子id_pdbx驻留溶剂原子标签替代id_pdbx驻留解决方案原子身份验证_pdbx驻留解决方案原子身份验证_pdbx驻留解决方案原子身份验证_pdbx杂质溶剂原子。PDB_输入代码_pdbx距离溶剂原子相邻大分子距离_pdbx距离solvent_atoms.neighbor距离11欧?A HOH 1146?6.79 .21欧?B怎么1113?5.96 .
链ID如何与残渣编号相关?
文件中的所有残留物和化学基团应唯一标识。一旦为与其相关的聚合物和化学基团分配了链ID,每个链ID的所有残留物、化学基团和水的编号必须是唯一的。此处还应考虑由于实验数据有限而未建模的残留物编号。wwPDB鼓励沉积带有序列残基编号的聚合物链。对于蛋白质链,鼓励作者尽可能遵循UniProt残基编号。应尽可能避免使用非连续的残数编号和插入代码,以便更大的科学界容易解释结构。如果作者提供的坐标残数在特定链ID中是唯一的和连续的,则不会对残数进行重新编号。如果作者已经将配体正确地分类到与其相关联的聚合物链上,则不会重新分配这些配体。
应如何分配聚合物链ID?
链ID应使用大写和小写字母、数字(0-9)及其组合。在使用所有大写字母和数字之后,最好使用小写字母和字母/数字组合。不得将符号用于链ID。
在PDB结构和手稿中一致使用链ID
在结构注释期间,与聚合物链相关的化学基团和水的编号和链ID可以根据wwPDB链ID规则进行更改。wwPDB强烈鼓励储户在任何出版物中使用wwPDB-指定的链ID和残数。在准备手稿之前,结构的存放和处理将确保手稿和PDB文件中链ID和残数的一致使用。注释过程中生成的验证和结构分析报告也可能有助于手稿的准备。
结构与PDB格式不兼容
无法以传统PDB文件格式表示的结构(PDB不兼容),例如,包含多个字符链ID、>62个链、PDB不兼容性二级结构记录和/或>99999个ATOM行的结构,将作为代表整个结构的单个PDBx/mmCIF文件在PDB存档中提供。此外,这些结构将具有TAR文件,其中包含PDB文件格式的尽力而为/最小文件集合,以支持仅依赖PDB文件形式的用户和软件工具。对于此类结构,在mmCIF文件中,_pdbx_database_status.pdb_format_compatible标志将设置为N:
_pdbx数据库状态pdb格式兼容
7.标题分配
如何分配标题关键字(_struct_keywds.pdbx_keywords)记录?
一个关键词用于简要描述PDB条目中存在的大分子的广泛功能。分类列表见本文件末尾和https://files.wwpdb.org/pub/pdb/doc/format_descriptions/class.dat.
标题函数由注释器分配
注释器通常在查看作者提供的UniProt关键字和关键字后分配函数。带注释的标题也将包含在条目关键字(_struct_keywords.text)中,如果标题是复数(/分隔),则将在关键字中添加单词“complex”。如果蛋白质是一种酶,则使用一般种类的酶。例如,如果E.C.编号以1开头,则标题被指定为氧化还原酶。参见附录A或https://files.wwpdb.org/pub/pdb/doc/format_descriptions/class.dat获取可用标题的列表。
- 如果蛋白质没有已知功能,则使用“未知功能”。
- 如果功能是假定的,例如“假定水解酶”,则标题将根据假定的赋值进行赋值,即“hydrolase”。
- 如果注释器不确定函数,注释器会要求作者从标准头列表中选择合适的头。
- 如果该函数是PDB中的新函数,则描述该函数的通用头将添加到标准头列表中。
可以使用以下规则组合两个或多个标题:
- 对于大分子络合物,标题用正斜杠分开(HEADER1/HEADER2)。例如,转录/DNA(注意蛋白质首先列在蛋白质/核酸复合物中)。
- 单词“complex”不会在标题记录中使用,但只要标题中有“/”,它就会出现在关键字记录中。
- 多功能高分子在不同标题之间有逗号:HEADER1、HEADER2
- 大分子的抑制剂、活化剂、受体、底物等功能可以添加到现有的集管(集管抑制剂)中,例如,水解酶抑制剂、水解酶活化剂、水解蛋白酶受体、水解液调节器。
如何分配关键字(_struct_keywords.text)?
关键词由条目作者提供。标题关键字(来自_struct_keywds.pdbx_keywords)记录也将在此处列出。
8.装配
四元组件(_pdbx_struct_assembly,_pdbx_struct_assembly_gen,_pdbx _struct_oper_list)
PDB条目中的四元结构可以包括软件计算的四元组装和/或作者确定的生物组装,或者有实验证据的分子的生物相关形式。
我们根据相互作用的表面积和大分子的结合推导出了结构的可能低聚物状态。通常,这种衍生结构和生物组装是相同的。然而,由于晶体堆积力、实验证据或作者的观点,生物组装和衍生低聚物结构可能不同。例如,大分子可以在晶体中被赋予六聚四元结构,但生物组装体可以是单体的。众所周知,PISA等第四纪结构项目1和PQS2不适用于所有情况,例如抗体,或生物过程具有低关联/分离特性的任何情况。除了作者提供的生物组装信息(从PDB档案中的文献或类似结构中得知)外,四元组装由注释人员计算和评估。
构成四元结构的矩阵将在_pdbx_struct_oper_list mmCIF类别中报告,并由wwPDB注释器分配。如果作者对生物组合的描述与晶体结构的显示不一致,则可由寄存人选择生物组合,并在文件中报告。如果两种聚合物具有界面,则会列出总表面积、埋表面积和自由能增益。
术语:寡聚物复合体中的亚基数量使用以单体结尾的名称描述(希腊语表示“部分,亚基”)。正式的Greco-Latinate名称通常用于前十种类型,最多可用于二十个亚单位,而高阶复合体通常由亚单位的数量描述,然后是-个亚单位。
在pdbx_struct_assembly.oligomeric_details和pdbx_struct_assembly.illogomeric_count中,将长度为3个或更多氨基酸或2个或更多核苷酸的任何多肽视为单体或二聚体等四元结构的命名:
| 1=单体 |
8=八元 |
15=十五碳 |
| 2=二聚体 |
9=非聚合物 |
16=十六进制 |
| 3=三聚体 |
10=十倍体 |
17=十七碳 |
| 4=四聚体 |
11=非摄像 |
18=十八碳 |
| 5=五元 |
12=十二元 |
19=非癸烷 |
| 6=六聚体 |
13=十三元 |
20=二十倍体 |
| 7=七元 |
14=十四碳 |
21-meric等。 |
请注意,PDB备注350中描述的多聚物代表该条目的同聚物或异聚物。
9.其他记录
文件版本控制和修订历史记录
wwPDB维护一个文件版本系统,允许记录保存人更新他们自己以前发布的条目。
使用#-#标识符指定每个PDB存档条目的版本号。第一个数字指定主要版本,第二个数字指定次要版本。记录结构(即发布的原子坐标的初始集合)被指定为版本1-0。此后,主版本数字随着给定条目的每次实质性修订而递增(例如,当原子坐标首次被记录保存人替换时,版本2-0)。“主要版本更改”定义为更新配体的原子坐标、聚合物序列和/或化学标识。所有其他变更均定义为“次要变更”。进行重大更改时,次要版本号重置为0(例如,1-0到1-1到2-0)。wwPDB保留PDB存档中条目的所有主要版本和最新次要版本。
特殊审计类别用于捕获文件更改的详细信息,直至条目修订的类别级别:
http://mmcif.wwpdb.org/dictionaries/mmcif_pdbx_v50.dic/Groups/audit_group.html
例如:
回路__pdbx审核修订历史记录序号_pdbx审核修订历史记录数据内容类型_pdbx审核修订历史记录主要修订_pdbx审核修订历史记录_pdbx审核修订历史记录修订日期1“结构模型”1 0 2020-03-252“结构模型”1 1 2020-04-083“结构模型”1 2 2020-05-06#_pdbx审核修订细节序号1_pdbx审核修订详细信息修订普通1_pdbx_audit_revision_details.data_content_type“结构模型”_pdbx_audit_revision_details.provider存储库_pdbx_audit_revision_details.type“初始版本”_pdbx_audit_revision_details.description?_pdbx_audit_revision_details.details?#回路__pdbx审核修订组序号_pdbx审核修订组修订普通_pdbx审核修订组数据内容类型_pdbx审核修订组1 2“结构模型”“数据库引用”2 2“结构模型”“结构总结”3 3“结构模型”“数据库引用”4 3“结构模型”“源和分类”5 3“结构模型”“结构总结”#回路__pdbx审核修订类别序号_pdbx审核修订类别修订普通_pdbx_audit_vision_category.data_content_type_pdbx审核修订类别类别1 2“结构模型”实体2 2“结构模型”pdbx_related_exp_data_set3 3“结构模型”实体4 3“结构模型”entity_name_com5 3“结构模型”entity_src_gen6 3“结构模型”struct_ref7 3“结构模型”struct_ref_seq#回路__pdbx审核修订项目序号_pdbx审计修订项目修订普通_pdbx审核修订项目数据内容类型_pdbx审核修订项目1 2“结构模型”“_entity.pdbx_description”2 3“结构模型”“_entity.pdbx_description”3 3“结构模型”“_entity.pdbx_ec”4 3“结构模型”“_entity_src_gen.gene_src_common_name”5 3“结构模型”“_entity_src_gen.pdbx_gene_src_gene”6 3“结构模型”“_struct_ref.db_code”7 3“结构模型”“_struct_ref.db_name”8 3“结构模型”“_struct_ref.pdbx_align_begin”9 3“结构模型”_struct_ref.pdbx_db_accession10 3“结构模型”“_struct_ref.pdbx_seq_one_letter_code”11 3“结构模型”“_struct_ref_seq.db_align_beg”12 3“结构模型”“_struct_ref_seq.db_align_end”13 3“结构模型”“_struct_ref_seq.pdbx_db_accession”
链接记录(_struct_conn、Link、SSBOND)
LINK记录将使用标准软件自动生成。对于各种链接记录,此软件中指定了各种截止距离。链接记录可以包括共价键合、金属配位等。作者也可以添加链接记录。
SSBOND记录是为参与二硫键的半胱氨酸残基创建的,不包括其他残基或配体之间的二硫键。标准软件会自动将struct_conn类别添加到文件中,列出金属离子和周围残留物之间的链接记录距离,符合标准配位几何。以下是PDB条目1W3M中LINK记录的摘录。mmCIF格式:
回路__结构conn.id_结构conn.conn类型id_struct_conn.pdbx离开原子标记_结构_注释.pdbx_PDB_id_struct_conn.ptnr1标签asym_id_结构conn.ptnr1标签comp_id_结构conn.ptnr1标签seq_id_结构conn.ptnr1标签原子id_结构conn.pdbx_ptnr1标签alt_id_struct_conn.pdbx_ptnr1_PDB_ins_code结构代码_结构conn.pdbx_ptnr1\standard_comp_id_struct_conn.ptnr1_对称_结构conn.ptnr2_label_asym_id_结构conn.ptnr2标签comp_id_结构conn.ptnr2_label_seq_id_结构conn.ptnr2_label_atom_id_结构conn.pdbx_ptnr2_label_alt_id_结构_签名.pdbx_ptnr2_PDB_ins_code_结构_签名.ptnr1_auth_asym_id_结构确认.ptnr1_auth_comp_id_结构_签名.ptnr1_auth_seq_id_结构_签名.ptnr2_auth_asym_id_结构名称ptnr2_auth_comp_id_结构_签名.ptnr2_auth_seq_id_struct_conn.ptnr2_对称_结构conn.pdbx_ptnr3_label_atom_id_结构_注释.pdbx _注释3_标签_序列_id_结构_注释.pdbx _注释3_标签_组件id_结构conn.pdbx_ptnr3_label_asym_id_结构conn.pdbx_ptnr3_label_alt_id_结构_编号pdbx_ptnr3_PDB_ins_code_struct_conn.详细信息_结构名称pdbx_dist_value_struct_conn.pdbx值顺序共价1共价两者?M液化天然气。C1 A?1_5 55 A ASP 1 N?液化天然气0 A ASP 1 1_555?1.306 ?共价2共价二者?M液化天然气。C1 B?1_5 55 A ASP 1 N?液化天然气0 A ASP 1 1_555?1.350 ?共价3共价1?ASP 1 C?1_5 55安VLL 2牛顿?ASP 1 A VLL 2 1_555?1.334 ?共价4共价两者?VLL 2摄氏度?1_5 55 A CPI 3 N?一个VLL 2一个CPI 3 1_555?1.340 ?metalc1金属c?VLL 2 O?1_5 北加利福尼亚州55号。加利福尼亚州?A VLL 2 A CA 3013 1_555?2.335?共价5共价1?VLL 2 NG2?1_5 55 A专业11 C?一个VLL 2
二级结构记录
螺旋线和图纸记录由Promotif算法自动生成,并存储在struct_conf、struct_sheet、struct_sheet_order、struct_sheet_range和pdbx_struct_sheet_hbond类别中。希望提供自己的螺旋线和表记录的作者可以这样做。
从2021年6月起,不再为PDB条目生成PDB格式的文件,其中板材拓扑无法以PDB格式生成(复杂的beta板材拓扑,其中beta板材中的绞线定义无法以线性描述形式表示)。然而,在PDBx/mmCIF格式的文件中,这个限制并不是问题,在该文件中可以正确地捕获这些复杂的测试表拓扑。因此,对于这些示例,wwPDB将继续在PDBx/mmCIF格式的文件中提供带有螺旋线和图纸信息的二级结构。
验证、计算粘结、角度、扭转偏差等。
蛋白质条目的键和角度偏差的计算将基于更新的Engh&Huber氨基酸目标值三对于核酸,帕金森等人的统计数据是用于这些计算4。所有偏离标准目标值6倍以上的键和角度都将标记为偏差。PHI/PSI值基于Kleywegt的计算5.
偏差以以下mmcif类别和PDB格式文件的REMARK 500报告。
在同一非对称单元中闭合触点(_pdbx_validate_CLOSE_contact)
对称相关闭合触点(_pdbx_validate_symm_contact)
债券长度(_pdbx_validate_rmsd_BOND)
连接角(_pdbx_validate_rmsd_angle)
扭转角(_pdbx_validate_TORSION)
非CIS、非TRANS(_pdbx_validate_peptide_omega)
侧链平面组(_pdbx_validate_planes)
主链平面度(仅蛋白质)(_pdbx_validate_MAIN_CHAIN_plane)
手性中心(仅蛋白质C-α)(_pdbx_validate_cardical)
其他条目详细信息
其他条目详细信息在_pdbx_entry_details中进行了注释,例如,在_pdbx _entry.details.compound_detail(REMARK 400)中化合物的其他功能详细信息,在_pdb2_entry-details.sequence_detail斯(REMARK 999)中的其他聚合物序列详细信息,以及关于_pdbx_entry_details.nonpolymer_details中配体的任何其他信息(REMARK 600)。
相关条目
作者可以提供关于相关条目(pdbx_database_related,REMARK 900)的信息,以将其他条目与当前条目相关联。
_struct_site(site)记录的折旧
2021年6月,生成_struct_site和_struct_site_gen记录的内部遗留软件退役。当时,wwPDB不再为新存放的PDB条目生成这些类别,但现有条目不受影响。
10.结构基因组学条目
结构基因组学(SG)条目通常是由世界各地的各种结构基因组学团体沉积的X射线或核磁共振结构。在美国,这些结构主要由几个蛋白质结构倡议(PSI)小组保存。欧洲、英国、加拿大和日本也有几个结构基因组学中心。SG结构的处理方式与其他PDB沉积相同。对于这些条目的注释,只有一些额外的规则。如下所示:
- 通常,SG条目以“立即发布”状态存放。在特殊情况下(如CASP竞赛),可以在发布之前处理并保留一段预定的时间。
- 对于SG组存放的条目,作者列表还包括SG中心的名称(如JCSG、MCSG、BSGC等)。
- 关键词中还包括以下单词和短语:SG中心名称(全称和缩写)、Structural Genomics。如果条目来自PSI中心,则将缩写“PSI”和单词“Protein Structure Initiative”添加到关键字。PSI项目第二阶段的条目标记为PSI-2。
- 对于由中心存放的SG条目(也将目标存放到TargetDB),条目中每个序列的TargetDB ID都包含在文件中,并显示在_pdbx_database_related(REMARK 900)中。
- 项目名称、中心名称和中心缩写包含在_pdbx_SG_project mmCIF类别中。
11.X射线结构的特定信息
X射线结构的沉积
所有由晶体X射线衍射确定的结构,如果结构是非病毒衣壳的结构,则应包含整个晶体不对称单元(ASU)的原子坐标。ASU被定义为可以旋转和平移以仅使用晶体对称性允许的对称算子生成一个单元单元的最小单元。不对称单元可以是一个多聚蛋白的一个分子或一个亚单位,但也可以是多个。
结构因子文件中包含的信息
结构因子(sf)文件应包括单元、空间组、对称性、波长和条目反射次数等信息。sf文件标题中包含的日期将是发布日期,而不是沉积日期。如果作者包含多个结构因子文件(例如一个用于细化的集、多个用于阶段化的集等),则数据将作为多个数据块存档。例如,细化反射将列在类别“_refln”中,阶段化数据集将列在类“_phasing_set_refln.”中不同的数据集可以根据晶体和/或波长ID进行区分。如果存在不同的单元格尺寸,此信息也将包含在文件中。用于细化的数据集应列为第一个数据块,并应包含衍射指数h、k、l、观测振幅和/或强度、它们各自的σ值和细化测试集。
结构系数文件示例见附录B。
精炼和数据收集统计重要数字
作者提供的值将保留在mmCIF和PDB文件中。
晶体结构中的氢
无论分辨率如何,晶体结构中的氢都将被保留,并由储户提供占用空间。
马修斯系数和溶剂含量
对于晶体结构,马修斯系数和溶剂含量将使用以下方程式自动计算:
马修斯系数6=单位细胞的体积/(大分子的分子量*Y*X)其中Y是单位细胞中不对称单元的数量(即空间群中对称算符的数量)。未知变量X是不对称单元中的分子数。
溶剂含量=1-1.23/(马修斯系数)
分子量包括基于序列的蛋白质和核酸,不含水和配体。对于病毒衣壳和蛋白水解片段,马修斯系数和溶剂含量应由作者提供,不会自动计算。
反射次数
细化反射次数是指满足分辨率和观测极限的晶体唯一测量反射次数。数据采集的反射次数是指根据标准σ(I)或标准σ。即使使用了Friedel对,报告用于细化的反射数也应小于或等于报告用于数据收集的反射数。
孪生结构
基于孪晶衍射数据的结构可以通过使用mmCIF公共交换字典pdbx_twin中的孪晶标记来识别。标记可用于识别孪生运算符、类型和分数。
孪生结构的结构因子文件应首先包括用于细化的孪生数据。如果作者有详细的数据,它也可能包含在文件中。结构因子文件中应包含pdbx_reflns_twin标记。
MAD数据
如果收集了MAD数据,则鼓励作者提供用于结构求解和优化的所有数据集。用于优化的数据集应首先列在结构因子文件中。鼓励作者提供其他(分阶段)数据集。还应提供所有数据集的波长、源和其他数据收集信息。
BioSync和同步加速器数据采集信息
有关同步辐射源和束线的信息将与BioSync数据库中使用的标准名称保持一致(http://biosync.rcsb.org/).
12.核磁共振结构的特定信息
伪原子(Q原子)
为NMR条目提交的伪原子(也称为Q原子)将从条目中删除。
叠加模型
NMR条目的至少一个域应该在所有模型中重叠。众所周知,对于多畴核磁共振结构,畴运动阻止整个结构通过分子的长度排列。然而,为了突出区域的相对运动,在PDB加入码下存放的所有模型中叠加至少一部分结构是有意义的。这不会减损坐标集或实验的科学价值,相反,它可以突出相对于固定点的域运动。叠加不一定是任意的,但可以由存款人选择。这将使更大的科学界容易识别蛋白质折叠。它也有助于识别结构不同部分的模型变化。
实验方法:有两种类型的核磁共振实验方法(EXPDTA,_exptl.method):
固态核磁共振溶液核磁共振
溶液核磁共振
集合的同质性
沉积中的所有模型应以适当的作者确定的方式叠加,并且只应使用一种叠加方法。不同实验的结构或结构的不同域不应作为沉积模型进行叠加和沉积。
核磁共振系综中的所有模型都必须是均匀的——每个模型必须具有完全相同的原子(氢原子和重原子)、序列和化学性质。最小平均结构的沉积必须伴随着系综,并且必须与系综一致。
型号类型
MDLTYP记录包含与条目中显示的坐标相关的附加注释。该记录将显示最小平均结构,以及最小平均结构的型号。相应的cif是_struct.pdbx_model_type_details。MDLTYP最小平均值,X型
最佳代表一致性
该记录定义了集合中最具代表性的一致性(pdbx_nmr_representative.conformer_id,REMARK 210)。
例子:
_pdbx_nmr_representative.conformer_id 1_pdbx_nmr代表性选择标准“最低能量”
在结构注释过程中,最好的代表性一致性被移至模型1,因为HELIX/SHEET、SITE和LINK记录是基于坐标中的第一个模型生成的。将通知作者注释人员如何处理此情况。
13.电子显微镜结构的特定信息
3DEM有两种常用的实验方法:电子显微镜(EM;包括单粒子和螺旋重建技术、层析成像和亚层析平均)和电子晶体学(EC)。有三种主要的数据文件类型与3DEM关联:
电磁沉积可能仅包含地图、坐标和地图,或仅包含坐标。任何存放的原子坐标都必须有一个关联的地图,即这些坐标的衍生地图。该相关地图必须是共享沉积的主地图(参见下面的地图信息部分)(如果沉积包含地图和坐标)或不同沉积的主图。在任何情况下,地图的EMDB ID必须包含在_pdbx_database_related(REMARK 900)中,作为“相关EM体积”(任何其他相关地图将被指定为“其他EM体积”)。此外,如果没有向EMDB同时或提前发布其相关地图,则不会向PDB发布任何EM原子坐标。
EC沉积通常只包含坐标和结构因子,类似于X射线晶体学。有时,沉积中也会包含地图,或与不同沉积相关。在没有结构因素的情况下,必须有一个关联的映射。EC沉积的地图必须与EM沉积的地图相同。
除了三种主要数据文件类型外,还有一些辅助文件类型:
- 地图图像文件(对于包含地图的沉积来说是必需的)
- FSC曲线XML(可选;应该是用于估计报告分辨率的FSC曲线)
- 层线(层析成像条目可选)
地图信息
地图可采用CCP4和MRC两种格式,并在沉积后转换为CCP4格式。每个地图有三个由储户提供的数据项:体素大小(以像素/埃为单位;对于所有地图都是必需的)、建议的等高线级别(用于等高线地图的最佳显示;对于除层析图以外的所有地图都为必需的)和注释详细信息(地图的简要描述;对于所有贴图都是可选的)。有四种不同的子类型分配给映射:
- 主地图。主地图是地图沉积(和EMDB条目)的中心。如果存放地图,则必须提供主地图。每个沉积层只有一张主地图,wwPDB提供的地图验证将集中在主地图上。如果放置的坐标是从地图中导出的,则该地图必须是主地图。
- 半幅地图。半图是在重建主图期间用于交叉验证的两个图(基于数据集的备用半)。放置半幅地图是可选的,但鼓励这样做。如果提供了半幅地图,那么必须有两幅,而且只有两幅,并且它们应该是未经修改和裁剪的。
- 面具。掩模在地图处理过程中用于去除噪声、提高地图质量、隔离地图部分(分割)等。掩模的沉积是可选的,并且可以将任意数量的掩模作为沉积的一部分包括在内。
- 其他地图。沉积中包含的任何非原始地图、与原始地图相关联的半幅地图或掩模的地图都被标记为“附加地图”。这些可以包括原始地图、无遮罩地图、分段等。附加地图的沉积是可选的,任何数量的附加地图都可以包含在沉积中。
EM元数据
相对于其他PDB沉积,收集了3DEM沉积的其他元数据。通常,这些涉及与3DEM数据采集和3D体积重建相关的设备和参数,以及相关原子坐标的细化。在PDBx/mmCIF中,这些元数据被指定为_em_*(其中*是通配符)。它们对应于为EMDB使用而收集的元数据,尽管PDB也将使用部分但不是全部元数据。
病毒衣壳和其他复杂组件
为了注释的目的,复杂组件被定义为通过对一组沉积坐标应用一组非晶体学旋转/平移变换来构建完整生物组件和/或晶体学不对称单元的结构。
二十面体病毒
二十面体病毒是沉积在PDB上的最常见的复杂组合体。作者通常放置二十面体非对称单元的坐标,并提供一组60个变换矩阵,应用于坐标以生成完整的生物组合。我们将继续向作者索取这些矩阵。根据作者提供的矩阵和坐标,我们将计算60个有序矩阵的标准集,以及将复数移动到标准二十面体框架(与ViperDB使用的框架相同)的转换。计算出的矩阵、帧转换以及如何将它们应用于坐标以构建程序集的描述将存储在_pdbx_struct记录中。
对于晶体结构,我们还将要求完整描述如何构建晶体非对称单元,该描述将存档在_pdbx_struct记录中。如果在晶体框架中提供坐标,则非晶体学对称变换也将放置在struct_ncs_oper记录中,并将出现在MTRIX记录中,从而可以根据结构因子数据进行验证。
规则对称性
二十面体点对称只是一种可以被复杂组件采用的对称。其他点对称(见下表)或螺旋对称也是可能的。对于作为复杂组件存放的所有结构,我们将在_pdbx_point_symmetry或_pdbx_helical_symmetric记录中适当地存档对称信息。
| 点对称性 |
Schoenflies符号 |
#同等职位 |
| 圆形的 |
抄送 |
n个 |
| 二面体的 |
Dn(数字) |
第2个 |
| 四面体的 |
T型 |
12 |
| 八面体的 |
O(运行) |
24 |
| 二十面体的 |
我 |
60 |
摘自:国际结晶学表,A卷,第4版,表10.4.2,第782-783页
点对称性
点对称信息存储在pdbx_point_symmetry类别(REMARK 300)中。
例子:
_pdbx_point_symmetry.entry_id 2BK1_pdbx点对称。舍恩弗里斯_符号C_pdbx点对称环形对称38
T=四边形
D=双螺旋
O=八面体
I=ICOSAHEDRAL公司
15.重新定义另一作者的数据
以下文本是作者重新定义另一作者数据(pdbx_database_remark,remark 0)时的专用注释。注释将始终出现在作者修改其他人数据的条目中。该入口将被视为实验结构。
基于1T3N数据的条目1ZET示例:
_pdbx数据库标记id 0_pdbx数据库标记文本;本条目1ZET反映了由作者D.T.NAIR、R.E.JOHNSON、S.PRAKASH、L.PRAKASH、A.K.AGGARWAL确定的R1T3NSF原始数据中结构数据的替代建模;#
中原始作者的论文将被列在引文类别中,引文_id=Original_data_1:
回路__引文.id_引文.标题_引用.journal_abbrev_引文.期刊卷_引文第页_引文.page_last_引用年份_引文.日志_美国材料试验协会_引用.国家_引文.日志_id_ISSN_引用.journal_id_CSD_引文图书出版商_引文.pdbx数据库_id_PubMed_引文.pdbx数据库_id_DOI初级“DNA聚合酶:DNA复制中的Hoogsteen碱基连接?”自然437 E6’7;讨论E7’2005 NATUAS UK 0028-0836 0006?16163299 10.1038/性质04199original_data_1“Hoogsteen base-pairing通过人类DNA聚合酶iota进行复制。”《自然》430 377 380 2004 NATUAS UK 0028-0836 0006?15254543 10.1038/自然02692#回路__引用_作者.引用_id_引文作者姓名_引文_作者序数初选“Wang,J.”1original_data_1“Nair,D.T.”2original_data_1“R.E.约翰逊”3原始数据_1“Prakash,S.”4原始数据_1“Prakash,L.”5original_data_1“阿加瓦尔,A.K.”6
注:在如上所述包含pdbx_database_remark(remark 0)的条目中,pdbx_database_related(remark 900)也将反映现有实验数据的重用,如下例所示:
#_pdbx_database_related.db_name PDB_pdbx_database_related.db_id 1T3N_pdbx_database_related.details“DNA聚合酶Iota与DNA和dTTP复合物催化核心的结构”_pdbx数据库已更新内容类型重新定义
SF文件包含以下内容,以及作者添加内容的附加信息(如果适用):
_审核修订id 1_0_审核创建日期2005-07-19_审核.更新_记录;初始版本中,作者使用了pdb条目1t3n中的sf文件,并添加了列Fcalc、phases和FOM;
附录:
A.标题列表(struct_keywords.pdbx_keywords)
以下是处理PDB条目时要使用的标题列表:
过敏原
抗生素(肽,含糖)
抗冻蛋白
抗菌蛋白
抗菌蛋白
抗毒素
抗肿瘤蛋白
抗病毒蛋白
细胞凋亡
吸引力
生物合成蛋白质
血液净化
碳水化合物
细胞粘附
电池周期
细胞侵袭
CHAPERONE公司
昼夜节律蛋白
收缩蛋白
细胞因子(包括白细胞介素、干扰素)
DE NOVO蛋白(人工设计,经常合成)
DNA
DNA-RNA杂交(当生物组装包含混合DNA和RNA残基或链时使用)
电子运输
内吞作用
脱细胞症
黄素蛋白
荧光蛋白
基因调节(仅在转录、复制、,翻译不适用)
荷尔蒙
水解酶(E.C.3.-.-.-)
免疫系统(包括抗体、抗原)
免疫抑制剂
异构酶(E.C.5.-.-.-)
连接酶(E.C.6.-.-)
脂类运输
发光蛋白
裂解酶(E.C.4.-.-.-)
膜蛋白(无其他已知功能)
金属运输
马达蛋白
神经肽
癌蛋白
氧化还原酶(E.C.1.-.-.-)
氧气结合
氧气储存
氧气运输
光合作用
植物蛋白(无其他已知功能)
质子运输
蛋白质转运(一种参与转运其他蛋白质的蛋白质)
重组
复制
核糖体(仅在翻译不正确时使用;即使存在,也不要指定/RNA!)
核糖体蛋白
核糖核酸
信号蛋白(包括G蛋白)
拼接
结构基因组学(可能基因的产物)
结构蛋白
表面活性剂蛋白质
毒素(非抗生素,可使用例如水解酶抑制剂、毒素)
转移酶(E.C.2.-.-.-)
转录(DNA到RNA)
翻译(蛋白质合成;优于核糖体)
转锁酶(E.C.7.-.-.-)
转运蛋白(一种运输任何东西的蛋白质)
核蛋白(是否参与RNA/DNA结合或某种核加工尚不清楚)
病毒(用于整个病毒衣壳)
病毒蛋白(病毒衣壳中不涉及的病毒蛋白)
类病毒颗粒(用于聚集类病毒颗粒但不是标准病毒的情况)
当不知道其他功能时,请使用以下功能:
胆碱结合蛋白
细胞质蛋白质(一种功能尚不清楚但已知存在于细胞胞浆中的蛋白质。)
DNA结合蛋白
RNA结合蛋白
脂类结合蛋白
金属结合蛋白(如ZN、FE)
肽结合蛋白
蛋白质结合(意味着蛋白质与蛋白质的结合)
糖类结合蛋白xxx结合蛋白(如果以上均不适用,则适用于任何xxx配体,如血红素、维生素D、生物素)
蛋白质纤维
未知功能
B.结构系数格式
示例1包含用于细化的单个数据集的结构因子文件
数据_rxxxxsf#_审核修订id 1_0_审核创建日期?_audit.update_record“初始版本”##_条目id xxxx##_单元格输入id xxxx_电池长度a 118.8600_单元格长度_ b 155.0300_电池长度c 155.5400_单元格角度_阿尔法90.0000_单元格角度_贝塔90.0000_单元格角度_伽马90.0000#_对称条目id xxxx_对称性。国际表格编号20_symmetry.space_group_name_H-M“C 2 2 21”#回路__symmetry_equiv.id(对称_等效.id)_对称_equiv.pos_as_xyz1'X,Y,Z'2'-X、-Y、Z+1/2'3'X,-Y,-Z'4'-X、Y、-Z+1/2’5'X+1/2,Y+1/2,Z’6’-X+1/2,-Y+1/2,Z+1/2’7'X+1/2,-Y+1/2,-Z'8'-X+1/2,Y+1/2,-Z+1/2’##_衍射id 1_衍射晶体id 1_衍射细节?#_衍射_辐射_波长.id 1_差_辐射_波长_波长0.98100#_exptl_晶体.id 1#_reflns_scale.group_code(参考_刻度组代码1)#回路__refn.波长id_refn.crystal_id_refn.scale_group_code(参考缩放组代码)_refn.index_h参考号_refn.index_k参考号_refn.index_l参考号_refn.状态_参考。F_月份_单位_参考。F_meas_sigma_au公司_参考。计算(_C)_反射相位_refn.fom(参考号)_参考号pdbx_HL_A_iso_参考号pdbx_HL_B_iso_参考号pdbx_HL_C_iso_参考pdbx_HL_D_iso1 1 1 0 0 6 o 299.0 6.4 1306.2 0.0 0.32 0.33 0 0.00 0.001 1 1 0 0 10 o 726.8 15.0 1756.7 180.0 0.99 2.86 0 0.00 0.00...#反射结束
示例2包含最终细化和阶段化数据集的结构因子文件
#_审核修订id 1_0_审核创建日期?_audit.update_record“初始版本”##此文件包含两个数据集。第一个数据集用于细化。#第二个数据集用于定相。#_条目.id xxxx##_单元格输入id xxxx_电池长度a 108.7420_单元格长度b 61.6790_电池长度c 71.6520_单元格角度_α90.000_单元格角度β97.1510_单元格角度_伽马90.0000#_对称条目id xxxx_对称性。国际表格编号5_symmetry.space_group_name_H-M“C 1 2 1”#回路__symmetry_equiv.id(对称_等效.id)_对称_equiv.pos_as_xyz1'X,Y,Z'2'-X、Y、-Z'3’X+1/2,Y+1/2,Z’4’-X+1/2,Y+1/2,-Z’##_差异id 1_衍射晶体id 1_defrin.details“用于最终细化的数据”#_衍射_辐射_波长.id 1_衍射辐射波长1.0#_exptl_crystal.id 1#_reflns_scale.group_code(参考_刻度组代码1)#回路__refn.波长id_refn.crystal_id_refn.scale_group_code(参考缩放组代码)_refn.index_h参考号_refn.index_k参考号_refn.index_l参考号_refn.状态_参考。F_月份_单位_参考。F_meas_sigma_au公司1 1 1-50 0 1 x? ?1 1 1 49 5 1 o 37.7 9.41 1 1 50 0 x? ?...#结束数据_rxxxxAsf##这是阶段化的第二个数据集。###_cell.entry_id xxxx单元_单元格。CCP4_波长_id 1_单元格。CCP4_晶体_id 1_电池长度a 108.742_单元格长度b 61.689_电池长度c 71.652_单元格角度α90.000_单元格角度β91.151_单元格角度伽马90.000#_对称条目id xxxx_对称性。国际表格编号5_symmetry.space_group_name_H-M“C 1 2 1”#回路__symmetry_equiv.id(对称_等效.id)_对称_equiv.pos_as_xyz1'X,Y,Z'2'-X、Y、-Z'3’X+1/2,Y+1/2,Z’4’-X+1/2,Y+1/2,-Z’##_差异id 1_衍射晶体id 1_diffn.details“用于相位调整的数据”#_衍射_辐射_波长.id 1_衍射辐射波长0.98100#_条目id xxxx#_exptl_晶体.id 1#_reflns_scale.group_code(参考_刻度组代码1)#回路__refn.波长id_refn.crystal_id_refn.scale_group_code(参考缩放组代码)_refn.index_h参考号_refn.index_k参考号_refn.index_l参考号_refn.状态_参考。F_月份_单位_参考。F_meas_sigma_au公司_refn.pdbx命名差异_refl.pdbx命名差异西格玛1 1 1-50 0 1 x?1 1 1-50 0 2 x?...#反射结束
示例3包含用于最终细化和缩放未合并强度的数据集的结构因子文件
数据_rxxxxsf#回路__审核.修订_id_审核创建日期_审核.更新_记录1_0 2009-12-08“首次发布”1_1 2016-08-10“添加未合并数据”#_单元格输入id xxxx_电池长度a 44.0630_电池长度b 75.9360_电池长度c 85.9440_单元格角度_阿尔法90.0000_单元格角度_贝塔90.0000_单元格角度伽玛90.0000#_衍射id 1_衍射晶体id 1_衍射环境温度?_衍射晶体处理?_diffrin.details“用于细化的振幅”#_衍射_辐射_波长.id 1_衍射辐射波长1.5418#_衍射reflns.diffrn_id 1_衍射reflns.pdbx_d_res_high 1.769_衍射reflns.pdbx_d_res_low 19.546_衍射反射极限h最大24_衍射反射极限h最小值0_衍射反射极限k最大值42_衍射_reflns.limit_k_min 0_衍射反射极限最大值48_衍射反射极限最小值0_衍射参考号28813_衍射_反射.pdbx编号_obs 28799#_条目id xxxx#_exptl_晶体.id 1#_reflns_scale.group_code(参考_刻度组代码1)#_对称条目id xxxx_symmetry.space_group_name_H-M“P 21 21 21”_对称性。国际表格编号19#回路__symmetry_equiv.id(对称_等效.id)_对称_equiv.pos_as_xyz1'X,Y,Z'2'X+1/2,-Y+1/2,-Z'3'-X,Y+1/2,-Z+1/2'4'-X+1/2,-Y,Z+1/2'##回路__refn.crystal_id_refn.波长id_refn.scale_group_code(参考缩放组代码)_refn.index_h参考号_refn.index_k参考号_refn.index_l参考号_refn.状态_参考。F_月份_单位_参考。F_meas_sigma_au公司_refn.intensity_meas(反射强度_测量值)_反射强度_大小_参考号pdbx_I_plus_参考号pdbx_I_plus_sigma_refl.pdbx_I_分钟_refl.pdbx_I_分钟_西格玛_参考号pdbx_F_plus_回流.pdbx_F_plus_sigma_refl.pdbx_F_分钟_refl.pdbx_F_分钟_西格玛_refn.pdbx命名差异_refl.pdbx命名差异西格玛1 1 1 0 0 6 o 1376.410 15.01046 10408.40 226.5000 10408.40226.5000 104 08.40 266.5000 1376.4101 15.01046 1376.41015.01046 0.000000 0.0000001 1 1 0 0 8 o 217.3632 2.853964 259.2000 6.800000 259.2000 6800000 259.000 6.800000 217.36322.853964217.3632 2.859964 0.000000 0.0000001 1 1 0 0 10 o 106.2069 3.010540 62.000003.50000062.00000.35000062.000000.350000106.2069 3.0 10540 106.2069 30.10540 0.000000 0.0000001 1 1 0 0 12 o 429.4464 5.076715 1011.800 23.90000 1011.800 2390000 1011.8 00 23.9000 429.4465 5.076714 42.94464 5.076 715 0.000000 0.000000...#结束数据_rxxxxAsf#_条目id xxxx#_衍射id 1_衍射晶体id 1_差异详细信息;scalepack未融合强度;#_exptl_晶体.id 1#_衍射_辐射.diffrn _id 1_衍射_辐射.波长_id 1_衍射_辐射_波长.id 1_衍射辐射波长1.5418#回路__衍射_refn.衍射_id_衍射_refn.波长id_衍射反射标准代码_衍射refln.scale组代码_衍射refln.id_衍射反射指数_差异_参考编号_k_衍射refn.index_l_衍射反射强度_衍射_refn.intensity_sigma1 1 1 1 1 0 0 6 10072.3 319.81 1 1 1 2 0 0 -6 10746.7 320.81 1 1 1 3 0 0 8 253.1 9.51 1 1 4 0-8 265.6 9.8...#反射结束
1E.Krissinel和K.Henrick(2005年)。晶体中蛋白质集合的检测。收录:M.R.Berthold等人(编辑):CompLife 2005,LNBI 3695,第163-174页。柏林-海德堡施普林格-弗拉格。
2Henrick,K.和J.M.Thornton。1998年。PQS:蛋白质四元结构文件服务器。趋势。生物化学。科学。23:358-361.
三结构质量和目标参数。R.A.Engh和R.Huber。《国际结晶学表》(2006年)。第F卷,第18.3章,第382-392页。
4“精炼含核酸结构的新参数”,加里·帕金森(Gary Parkinson)、雅罗斯拉夫·沃伊特科夫斯基(Jaroslav Vojtechovsky)、莱斯特·克洛尼(Lester Clowney)、阿克塞尔·布伦格(Axel Brunger)*和海伦·M·伯曼(Helen M.Berman)。(1996)晶体学报。D 52,57-64号
5“PHI/PSI-霍乱学:拉马钱德兰重访”,GJ Kleywegt和TA Jones(1996)Structure 41395-1400。
6参见马修斯,B.W.1968。蛋白质晶体的溶剂含量。分子生物学杂志。33:491-497和网址:http://www.doe-mbi.ucla.edu/~sawaya/tutorials/Characterize/Characterize.html