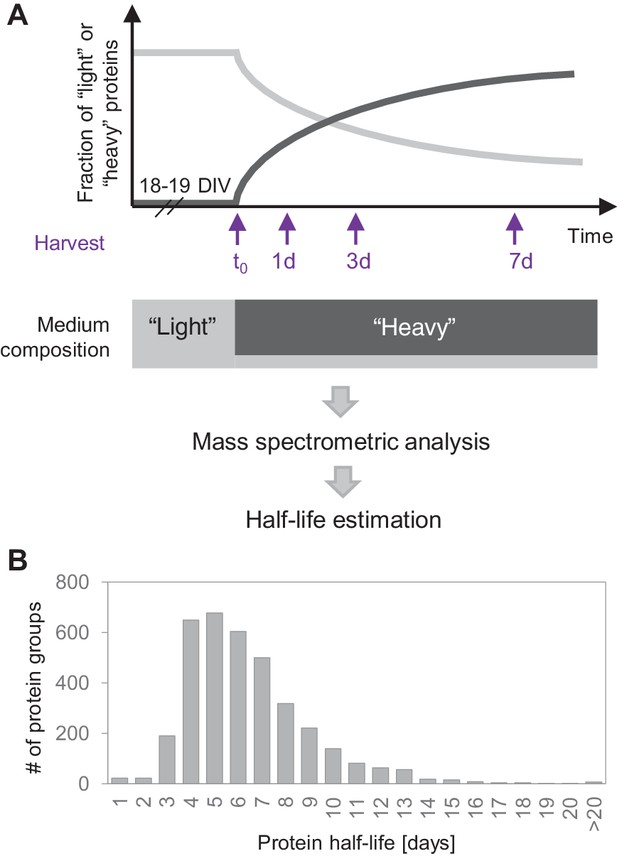
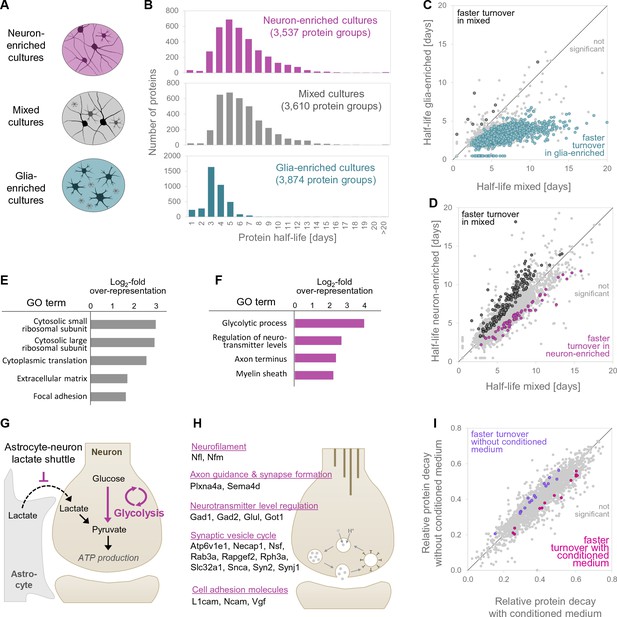
在这里,我们使用动态SILAC方法来描述不同细胞环境中神经元蛋白的周转率。总而言之,我们获得了5100多个蛋白质组的半衰期测量值,这些蛋白质组的半衰期范围从<1天到>20天,在混合培养物中的中位半衰期为5.4天。通过以下方法获得了类似的半衰期分布(中位数=4.2天)Cohen等人(2013)世卫组织测量了来自混合皮层培养物的2802个蛋白质组的周转率。最近的一项研究Mathieson等人(2018年)据报道,原代小鼠胚胎神经元的半衰期明显较短(中位数=1.9天)。这些较短的半衰期可能是由于培养物相对不成熟(从胚胎而非出生后的神经元制备)和相对较短的时间所致在体外(5天vs.18-19天)。我们进一步比较了由我们的在体外混合培养对小鼠脑蛋白半衰期的研究体内代谢标记。虽然蛋白质很明显很长寿体内(平均半衰期~9天)与在体外,基因比较显示蛋白质半衰期之间存在显著相关性(Spearman秩相关=0.62,p<0.001;图1——图补充3). 系统偏移可以通过不同的实验方法、物种(小鼠和大鼠)、细胞组成和环境以及所研究细胞的不同年龄来解释。Visscher等人(2016)最近报道的年轻细胞蛋白质半衰期缩短(秀丽隐杆线虫幼虫)与较老的细胞(成体秀丽隐杆线虫).
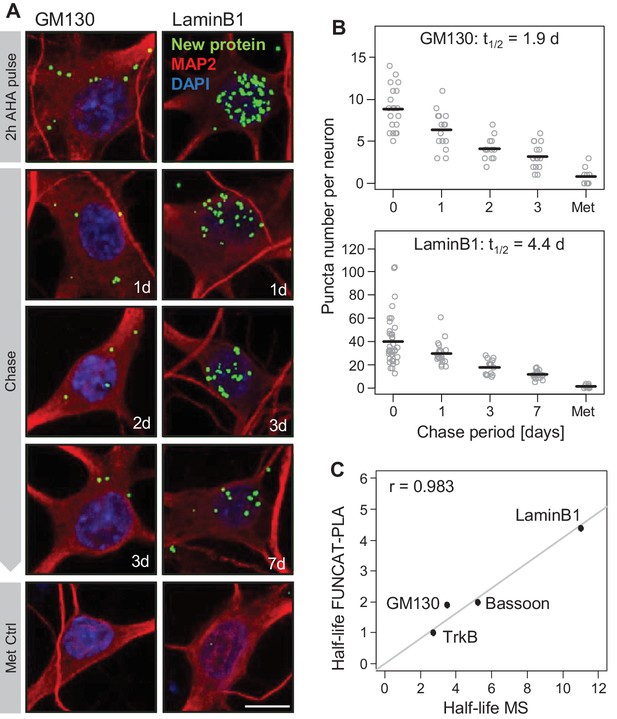
我们验证了使用动态SILAC和代谢标记和可视化策略FUNCAT-PLA测量的蛋白质周转率(汤姆·迪克等人,2015年). 我们发现,通过MS和FUNCAT-PLA测定的蛋白质的排序半衰期具有很好的相关性。蛋白质半衰期可视化就地然而,FUNCAT-PLA在系统上比MS测定的短。这种抵消的原因可能包括叠氮基或含AHA的蛋白质的相对不稳定,或增加AHA标记的灵敏度,以检测错误折叠蛋白的快速降解,而这些错误折叠蛋白被连续SILAC标记所遗漏(也在下文中讨论)。
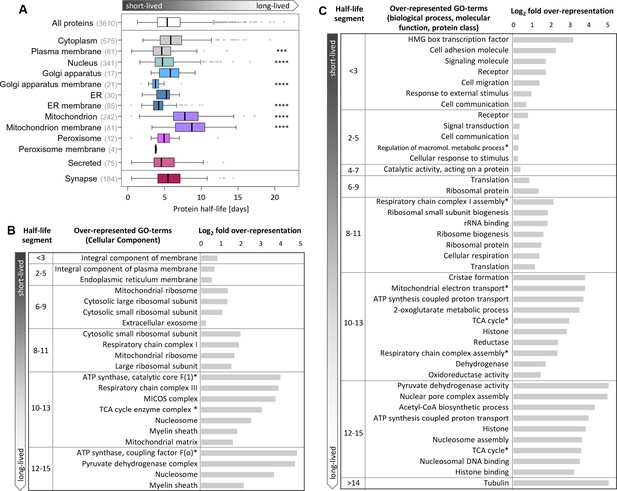
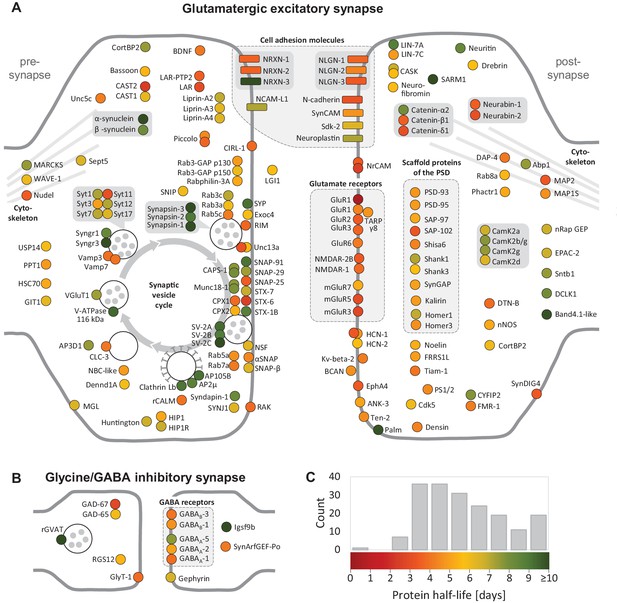
对我们的动态SILAC数据的分析表明,蛋白质周转受蛋白质的细胞位置影响。我们发现,膜蛋白,尤其是质膜、内质网和高尔基体的膜蛋白显示出明显短于平均半衰期的半衰期,而线粒体蛋白显示出显著更长的半衰周期。蛋白质周转的这些系统性差异可能是由于细胞溶质和膜蛋白的不同主要降解机制所致(Jin等人,2018年;Tai和Schuman,2008年)以及线粒体独特的分裂和融合质量控制机制(Cagalinec等人,2013年;戈麦斯和斯科拉诺,2008年). 有趣的是,突触蛋白群体的半衰期分布与普通群体相似。蛋白质半衰期也与蛋白质功能相关。例如,受体、信号分子和参与细胞通信的蛋白质在短寿命蛋白质组中过度表达。相比之下,其他功能类,如组蛋白和参与能量代谢的蛋白质,在长寿命蛋白质中的比例过高。这一发现支持了蛋白质寿命进化的观点,即参与内部和外部信号反应的蛋白质是短暂的,而在细胞中发挥更稳定功能的蛋白质寿命更长,以节省蛋白质合成和降解所需的能量。
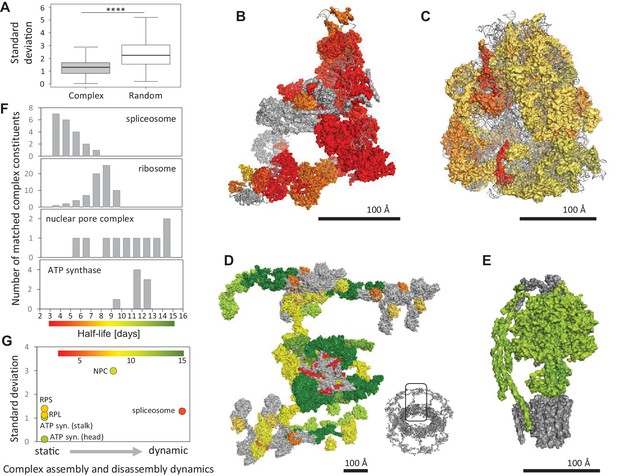
蛋白质通过设计相互作用,这种相互作用对细胞功能的执行和调节至关重要。Cohen等人(2013)研究表明,与初级皮层神经元中的非相互作用蛋白相比,相互作用蛋白具有更相似的半衰期。在此基础上,我们发现多蛋白复合物的成员显示出比随机选择的蛋白质更相似的半衰期。蛋白质复合物在其生物生成、组装和拆卸动力学方面差异很大,这些差异可能反映在复合物之间的不同平均半衰期或复合物内半衰期的变化上。我们详细研究了4种不同的多蛋白复合物的蛋白质成分的半衰期:ATP合酶、核糖体、核孔复合物和剪接体。对于我们检测的四种复合物,组装和拆卸方面最动态的是剪接体,该复合物中的蛋白质显示出最短的半衰期。核孔复合体显示出其相关蛋白质组分的高度可变半衰期,这与以前的研究一致(Beck和Hurt,2017年;Daigle等人,2001年;Mathieson等人,2018年). 令人惊讶的是,核糖体是一种典型的稳定细胞复合体,其中40S和60S亚基被认为是在细胞核和核仁中“终身”组装的,其成分的半衰期表现出很大的变异性,这表明一些核糖体蛋白可能被交换来修复(Pulk等人,2010年)甚至专门化核糖体(Xue和Barna,2012年)亚细胞隔室(霍尔特和舒曼,2013年).
对上述结果的解释必须注意,我们不知道我们测量的半衰期是否代表蛋白质的单一功能状态。最近对小鼠成纤维细胞的一项研究表明,约10%的蛋白质显示出非反应性衰变动力学,在合成后的最初几个小时内降解更快(McShane等人,2016年). 我们在这里使用的动态SILAC方法不允许我们准确区分具有不同稳定性的同一蛋白质的不同亚群。由于不稳定蛋白质在合成后很快被降解,因此它们代表了整个细胞裂解液中含量较低的亚群,与相应的稳定蛋白质群相比,它们对测量的MS信号的贡献较小。此外,我们没有在更换培养基后的第一个小时内采集样本,这对于准确解析具有不同衰变动力学的蛋白质种群是必要的。相比之下,在脉冲后短时间间隔内采样多个追踪时间点的脉冲相位实验中,可以更好地检测不同寿命的亚种群。在脉冲期间,所有子群体的蛋白质都被合成,在追逐过程中,这些子群体将以不同的时间常数衰变:短寿命蛋白质的部分将在短时间后消失,而长寿蛋白质的部分在最终降解之前将保持更长的时间。或者,在未来的实验中,纯化亚细胞隔室(例如突触体)或细胞器(例如核糖体),并独立测定特定细胞位置或功能群中蛋白质的半衰期,可以对具有不同半衰期的亚群提供新的见解。
已描述了氨基酸序列的内在因素,这些因素有助于蛋白质的稳定性;发现特定氨基酸位于蛋白质N末端时具有不稳定作用(Bachmair等人,1986年;Gibbs等人,2014年). 在我们的数据集中,与长寿命蛋白质相比,短寿命蛋白质组分中没有过度表达不稳定的N末端氨基酸,这表明N末端序列特性对原代海马培养物中的蛋白质寿命几乎没有影响。
通过比较胶质细胞或富含神经细胞的培养物中相同蛋白质的半衰期,我们进一步研究了细胞内环境是否对大脑蛋白质周转产生额外影响。我们发现,当蛋白在胶质细胞中表达时,其转换速度明显快于在神经元中表达时;这种差异不能用胶质细胞分裂来解释。其他人研究了不同脑细胞类型中核蛋白的周转率,并观察到与神经元相比,胶质细胞的蛋白质周转更快(富山等人,2013年). 我们还研究了细胞外环境对蛋白质周转的影响,比较了富含神经元的培养物和含有神经元和神经胶质的混合培养物中蛋白质的周转。我们发现,虽然大多数蛋白质半衰期在两种条件之间没有改变,但当胶质细胞存在于细胞外环境中时,一组重要的蛋白质表现出更快或较慢的转换。我们认为,半衰期的大部分变化可能是由于与胶质细胞的粘附性相互作用,而不是分泌因子,因为当我们比较经处理或未经处理的富含神经细胞的培养物与胶质条件培养液时,周转率没有大的差异。在没有胶质细胞的情况下表现出更快周转的蛋白质包括糖酵解酶和与囊泡循环相关的蛋白质。这种转换调节可能与各自蛋白质的活性状态有关。在没有神经胶质细胞的情况下,神经元缺乏神经胶质提供的乳酸(通过神经胶质-神经元乳酸穿梭器),因此需要增加糖酵解活性才能产生丙酮酸。糖酵解酶周转的增加可能反映了这种活性的增加。最近在酵母中证明了营业额和单个蛋白质活性之间的类似相关性(Martin-Perez和Villén,2017年). 综上所述,这些数据表明,细胞内(局部)和细胞外(全局)环境对蛋白质周转都有强大的影响。
在目前的实验中,我们依赖于神经元或胶质细胞在特殊培养条件下的相对富集。随着细胞类型特异性代谢标记的最新发展(Alvarez-Castelao等人,2017年),有可能结合此处使用的动态SILAC方法直接检查不同细胞类型的蛋白质周转。此外,了解可塑性如何调节和利用周转来修改大脑蛋白质组是未来研究的目标。