模型
描述
我们假设样本中的读取次数j个分配给基因的我可以用负二项(NB)分布建模,
(1)
它有两个参数,平均值μ伊吉以及方差.读取计数K(K)伊吉是非负整数。分布概率在补充注释A中给出。(所有补充注释都在附加文件中1.)当存在过度分散时,NB分布通常用于建模计数数据[12].
实际上,我们不知道参数μ伊吉和,我们需要根据数据进行估算。通常,重复次数很少,需要进行进一步的建模假设,以获得有用的估计。在本文中,我们开发了一种基于以下三个假设的方法。
首先,平均参数μ伊吉即基因的观察计数的期望值我在样品中j个,是条件相关的per-gene值的乘积q个我,ρ(j个)(其中ρ(j个)是样品的实验条件j个)和尺寸系数秒j个,
(2)
q个i、 ρ(j个)与基因片段真实(但未知)浓度的期望值成正比我在条件下ρ(j个). 尺寸系数秒j个表示库的覆盖范围或采样深度j个,我们将使用该术语普通比例尺对于数量,例如q个我,ρ(j个),通过除以进行覆盖调整秒j个.
第二,方差是a的总和散粒噪声项和a原始方差项,
(3)
第三,我们假设per-gene原始方差参数v(v)我,ρ是的平滑函数q个我,ρ,
(4)
这种假设是必要的,因为复制次数通常太少,无法精确估计基因的方差我根据这个基因的现有数据。这种假设使我们能够汇集来自表达强度相似的基因的数据,以进行方差估计。
方程中方差的分解(三)由以下层次模型驱动:我们假设基因片段的实际浓度我在样品中j个与随机变量成比例R(右)伊吉这样基因片段的比率我已排序为秒j个第页伊吉对于每个基因我和所有样品j个条件的ρ,的R(右)伊吉i.i.d.是平均值q个iρ和方差v(v)iρ因此,计数值K(K)伊吉,条件为R(右)伊吉=第页伊吉,是泊松分布率秒j个第页伊吉.的边际分布K(K)伊吉-当允许变化时R(右)伊吉-具有平均值μ伊吉和(根据总方差定律)方程式中给出的方差(三). 此外,如果R(右)伊吉根据伽马分布建模K(K)伊吉为NB(例如,参见[12],第4.2.2节)。
适合的
我们现在描述如何将模型拟合到数据中。这些数据是n个×米计数表,k个伊吉,其中我= 1,...,n个索引基因,以及j个= 1,...,米为样本编制索引。该模型有三组参数:
(i)米尺寸系数秒j个; 样本中所有计数的期望值j个与…成比例秒j个.
(ii)对于每个实验条件ρ,n个表达式强度参数q个iρ; 它们反映了基因片段的预期丰度我处于条件下ρ也就是基因计数的期望值我与…成比例q个iρ.
(iii)平滑功能v(v)ρ:ℝ+→ℝ+; 对于每个条件ρ,v(v)ρ对原始方差的相关性建模v(v)iρ关于期望均值q个iρ.
尺寸系数的目的秒j个是从不同样本中渲染计数,这些样本可能已按不同深度排序,具有可比性。因此,比率(K(K)伊吉)/(K(K)ij’)相同基因的预期计数我在不同的样品中j个和j’应等于尺寸比秒j个/秒j’if基因我没有差异表达或样本j个和j’是重复的。读取总数,∑我 k个伊吉,似乎是测序深度的一个很好的衡量标准,因此是秒j个然而,实际数据的经验表明,情况并非总是如此,因为一些高度差异表达的基因可能会对总读取计数产生强烈影响,导致总读取计数的比率不能很好地估计预期计数的比率。
因此,为了估计规模因素,我们取观察计数比率的中位数。将刚刚概述的程序推广到两个以上样本的情况,我们使用:
(5)
该表达式的分母可以解释为通过对样本取几何平均值获得的伪参考样本。因此,每个尺寸系数估计值计算为j个-第个样本的计数与伪参考的计数相同。(注:在审查这份手稿时,罗宾逊和奥什拉克[13]建议了类似的方法。)
估计q个iρ,我们使用样本计数的平均值j个与条件相对应ρ,转换为普通规模:
(6)
哪里米ρ是条件的重复次数ρ和在这些重复上运行。功能v(v)ρ,我们首先计算普通尺度上的样本方差
(7)
并定义
(8)
在附加文件的补充注释B中1我们证明了这一点w个iρ-z(z)iρ是原始方差参数的无偏估计量v(v)iρ方程式的(三)。
然而,对于少量重复,米ρ与应用程序中的典型情况一样w个iρ高度可变,并且w个iρ-z(z)iρ对于统计推断来说,这不是一个有用的方差估计量。相反,我们使用局部回归[14]关于图获得平滑函数w个ρ(q个),使用
(9)
作为我们对原始方差的估计。
需要注意避免局部回归中的估计偏差。w个iρ是平方随机变量和残差的总和倾斜。以下参考文献[15],第8章和[14],第9.1.2节,我们使用伽马族的广义线性模型进行局部回归,使用位置匹配包装[16].
差异表达测试
假设我们有米A类复制生物条件A和米B类条件B的样本。每个基因我,我们想权衡数据中该基因在两种条件下差异表达的证据。特别是,我们想测试虚假设q个国际机场=q个国际银行,其中q个国际机场是条件A和q的样本的表达式强度参数国际银行对于条件B,为此,我们将每个条件中的总计数定义为测试统计,
(10)
和他们的总金额K(K)是=K(K)国际机场+K(K)国际银行根据上一节中描述的错误模型,我们在下面显示,在零假设下,我们可以计算事件的概率K(K)国际机场=一和K(K)iB公司=b条对于任何一对数字一和b条。我们将此概率表示为第页(一,b条). 这个P(P)一对观测计数和的值(k个国际机场,k个国际银行)那么所有概率的总和小于或等于第页(k个国际机场,k个国际银行),假设总金额为k个是:
(11)
变量一和b条在上述总和中,取0,。。。,k个我秒到目前为止,提出的方法遵循了罗宾逊和斯迈思的方法[11]与其他条件测试类似,如Fisher精确测试。(参见参考[17],第3章,讨论测试中条件反射的优点。)
计算 第页(一,b条). 首先,假设在零假设下,不同样本的计数是独立的。然后,第页(一,b条)=优先级(K(K)我A类=一)优先级(K(K)我B类=b条). 因此,问题是计算事件的概率K(K)我A类=一类似地,和K(K)我B类=b条.随机变量K(K)我A类是以下各项的总和米A类
NB-分布随机变量。我们通过NB分布来近似其分布,NB分布的参数我们从K(K)伊吉为此,我们首先根据两种条件的计数计算合并平均值估计值,
(12)
这说明了虚假设规定q个我A类=q个我B类条件A的平均值和方差之和为
(13)
(14)
附加文件中的补充注释C1描述了NB的分布参数K(K)我A类可以根据和(为了避免偏差,我们不直接匹配力矩,而是匹配不同的一对分布统计数据。)K(K)我B类通过类比获得。
附加文件中的补充注释D1解释了我们如何计算方程式中的总和(11)。
应用
数据集
我们根据以下数据集给出了结果:
飞行胚胎中的RNA-Seq
B.Wilczynski、Y.-H.Liu、N.Delhomme和E.Furlong在苍蝇胚胎中进行了RNA-Seq实验,并在发表之前与我们分享了部分数据。在这个数据集的每个样本中,都有一个基因被设计成过表达,我们比较了两个生物复制品,每一个都是这样的条件,以下分别表示为“a”和“B”。
神经干细胞的标记序列
Engström公司等。[18]执行Tag-Seq[19]用于神经细胞的组织培养,包括四个来自胶质母细胞瘤衍生神经干细胞(GNS)和两个来自非癌神经干细胞。由于每种组织培养物来源于不同的受试者,因此具有不同的基因型,这些数据显示出高度的可变性。
酵母的RNA-Seq
那加拉克什米等。[1]对酿酒酵母文化。他们测试了两种库准备协议,数据传输和右侧,并对每个方案进行了三次测序,因此对于每个方案的第一次运行,他们有一个进一步的技术复制(相同的培养物,复制的文库制备)和一个进一步生物复制(不同的培养物)。
HapMap样本的ChIP-Seq
卡索夫斯基等。[20]通过ChIP-Seq比较了10名人类个体DNA区域的蛋白质占位。他们编制了聚合酶II和NF-κB的区域列表,并计算了每个样本对应于每个区域的读取数。这项研究的目的是调查不同地区的职业在个人之间的差异。
方差估计
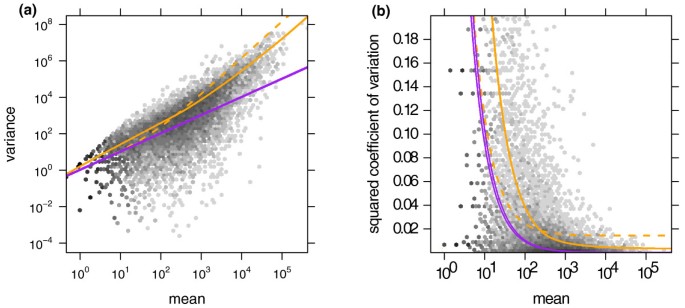
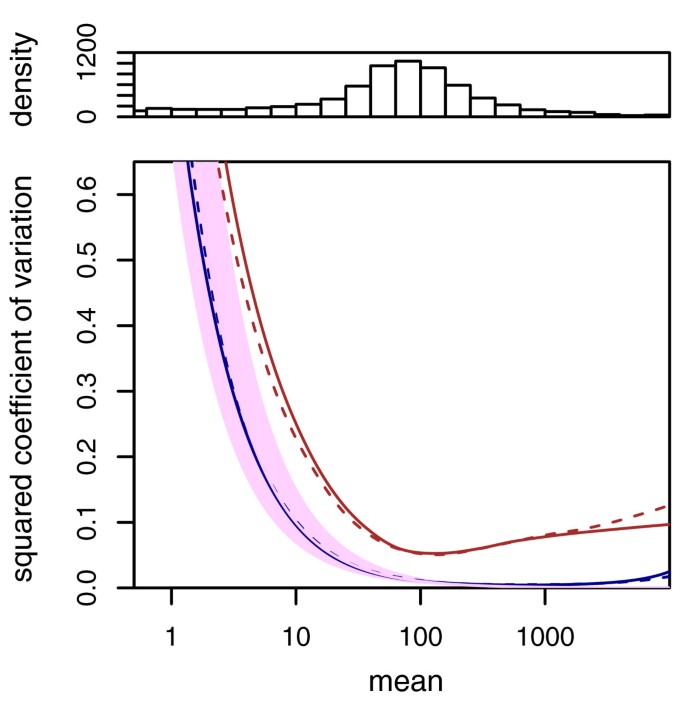
我们首先演示方差估计。图1a个显示了样本方差w个iρ(方程式(7))根据平均值绘制(方程式(6))对于条件A类在飞行RNA-Seq数据中。图中还显示了局部回归拟合w个ρ(q个)和散粒噪音.在图中1亿,我们绘制了平方变异系数(SCV),即方差与均方的比率。在该图中,橙色线和紫色线之间的距离是由于生物采样引起的噪声的SCV(参见等式(三)).
图中的许多数据点1亿这远远高于拟合的橙色曲线,可能会导致局部回归拟合效果不佳。然而,预计残差分布会出现强烈的偏差。参见附加文件中的补充注释E1以了解适用于验证匹配性的诊断的详细信息和讨论。
测试
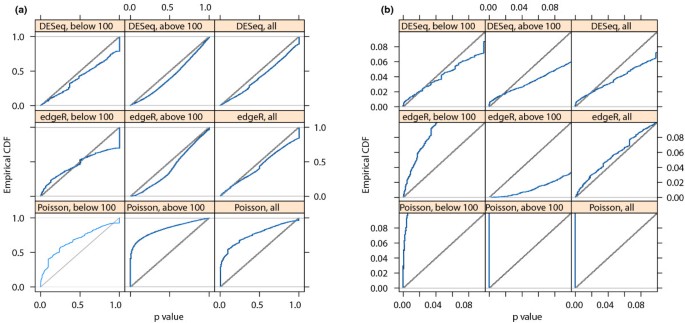
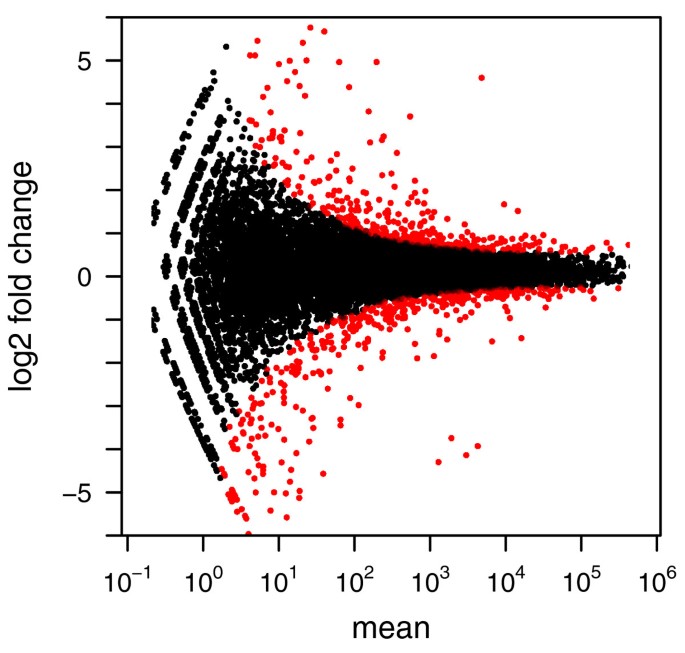
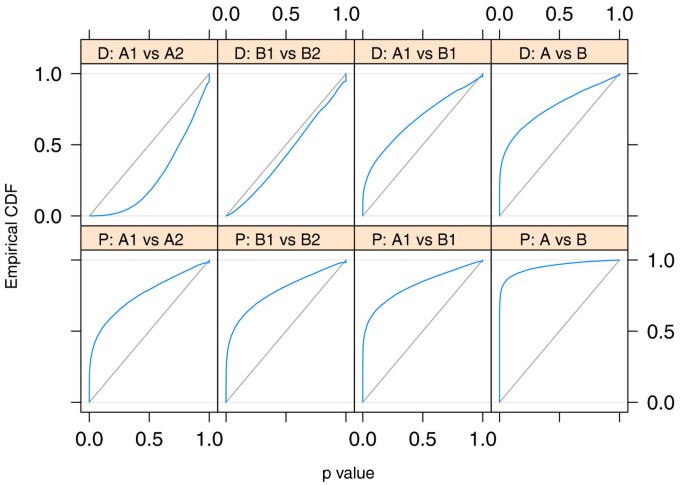
为了验证DESeq公司保持对I型错误的控制,我们对比了其中一个复制品的情况A类在对另一个样本的飞行数据中,对两个样本使用从两个重复中估计的方差函数。图2显示了P(P)通过比较获得的值。为了控制I类错误P(P)值低于阈值α必须≤α也就是说,ECDF曲线(蓝线)不应超过对角线(灰线)。如图所示,I类错误由边缘R和DESeq公司,但不是基于泊松的χ2测试。后者低估了数据的可变性,因此会导致许多假阳性拒绝。除了对真实数据进行评估外,我们还验证了DESeq公司对上述错误模型生成的模拟数据进行I类错误控制;参见附加文件中的补充注释G1接下来,我们对比了两者A类两个样本的对比B类样本。使用上一节中描述的过程,我们计算了P(P)每个基因的值。图三显示获得的褶皱变化和P(P)值。12%的P值低于5%。使用Benjamini和Hochberg程序进行多次试验的调整[21]864个基因(17605个)的错误发现率(FDR)为10%,差异表达显著。图中用红色标记三演示了检测差异表达的能力如何依赖于总计数。具体来说,低计数的强散粒噪声导致测试过程只调用非常高的折叠变化。还可以看出,对于大约100以下的计数,即使计数水平略有增加,也会降低散粒噪声的影响,从而降低折叠式变换的要求,而在较高计数时,当散粒噪声变得不重要时(参见图1亿),折叠式截止线仅弱依赖于计数水平。这些图有助于指导实验设计:对于弱表达基因,在散粒噪声很重要的区域,可以通过更深入的测序来增加功率,而对于更高计数状态,只有通过进一步的生物复制才能实现功率的增加。
与edgeR的比较
我们还分析了数据边缘R(版本1.6.0[8,10,11]). 我们跑了边缘R有四种不同的设置,即在通用分散模式和分段分散模式下,或者使用根据DESeq公司或获取顺序读取的总数。结果与这些选择没有太大关系,这里我们报告了标签分散模式的结果DESeq公司-估计的规模因素。(复制本文中报告的所有分析、数字和数字所需的R代码在附加文件中提供2; 此外,本补充提供了以下其他设置的结果边缘R。原始数据可以在附加文件中找到三)
回到图1我们看到了边缘R的方差的单值离散估计值低于DESeq公司弱表达基因和强表达基因的表达量较高。因此,如图所示2边缘R对于低表达基因是反保守的。然而,它通过对强表达基因更加保守来弥补这一不足,因此,平均而言,I型错误控制得以维持。
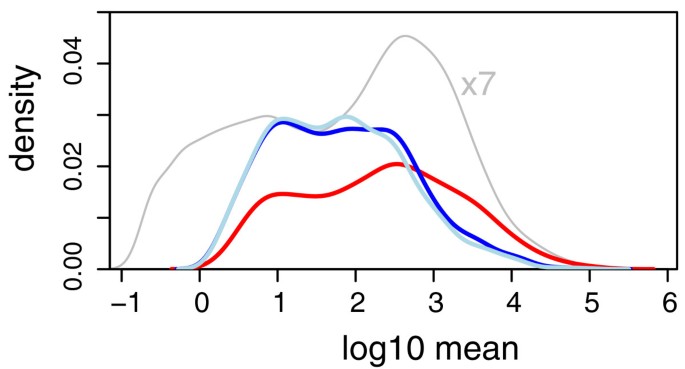
然而,在不同条件之间的测试中,这种行为可能会导致发现列表中出现偏差;对于当前数据,如图4显示,弱表达基因似乎过表达,而很少有平均水平高的基因被称为差异表达边缘R虽然两种方法的总体敏感性似乎相当(DESeq公司报告了864次点击,边缘R1127次点击),DESeq公司产生的结果在动态范围内更加平衡。
神经干细胞数据也获得了类似的结果,该数据集具有不同的生物背景和不同的噪声特性(参见附加文件中的补充注释F1). 本研究中提出的方差估计方案的灵活性似乎在一系列应用中比现有方法提供了真正的优势。
在没有复制的情况下工作
DESeq公司允许在一种或两种条件下分析没有生物复制的实验。虽然人们可能不想从这样的分析中得出强有力的结论,但它可能仍然对探索和假设生成有用。
如果复制仅适用于其中一种条件,则可以选择假设根据该条件的数据估计的方差-均值相关性也适用于未复制的条件。
如果这两种情况都没有重复,我们仍然可以基于以下假设进行分析:对于大多数基因,没有真正的差异表达,并且可以通过将两个样本视为重复来估计有效的均值-方差关系。少数差异丰富的基因将充当异常值;然而,它们不会对gamma-family GLM拟合产生严重影响,因为形状参数低值的gamma分布具有很重的右尾。预计可能会高估方差,这将使该方法变得保守。
我们使用飞行RNA-Seq和神经细胞Tag-Seq数据进行了这样的分析,将这两个数据集限制为两个样本,每个条件一个样本。对于神经细胞数据,正如预期的那样,估计的方差函数略高于从全球导航系统和NS公司复制。
使用它来测试差异表达,在FDR=10%时仍然发现269次点击,其中202次是所有可用样本的更可靠分析的612次点击中的一次。然而,就飞行RNA-Seq数据而言,862次命中中只有90次(11%)被恢复(有两次新命中)。这些观察结果的解释是,在神经细胞数据中,重复之间的变异性并不比条件之间的变异小得多,这使得后者成为前者的可用替代品。另一方面,对于苍蝇数据,复制之间的变异性远小于条件之间的变异,这表明复制提供了有关数据中实验变异的重要信息,但在其他方面没有可用信息(另见下一节)。
方差稳定变换
给定方差-均值依赖性,方差稳定变换(VST)是一个单调映射,使得对于变换后的值,方差(近似)与均值无关。使用方差-均值相关性w个(q个)估算人DESeq公司,VST由下式给出
(15)
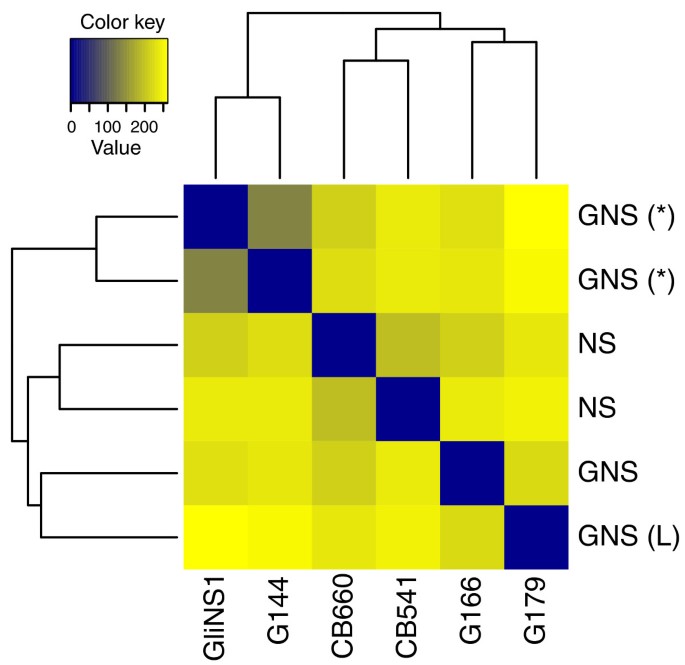
应用转换τ到通用刻度计数数据,k个伊吉/秒j个,生成在整个动态范围内方差大致相同的值。VST的一个应用是样本聚类,如图所示5; 这种方法比在未转换的计数数据上定义合适的距离度量更简单,其选择并不明显,并且可能不容易与可用的聚类或分类算法结合(这些算法往往是为具有类似分布特性的变量设计的)。
ChIP-Seq公司
DESeq公司也可用于分析比较ChIP-Seq分析。卡索夫斯基等。[20]分析HapMap个体的转录因子结合,并计算每个样本中映射到预定结合区域的读取次数。我们从他们的数据集中考虑了两个个体,即HapMap ID GM12878和GM12891,这两个个体至少进行了四次重复,并测试了这两个区域的不同职业。图的左上两个面板6显示同一个体内的比较,表明I型错误由以下因素控制DESeq公司。使用Benjamini-Hochberg调整,在10%FDR时无显著区域。然而,当对比这两个个体时,发现了不同的职业,在19028个区域中,有4460个在仅使用两个重复时显著,而在使用四个重复时则有8442个显著(右上方两个面板)。
使用另一种方法,卡索夫斯基等拟合泊松族的广义线性模型(GLM)。这(图的下一行6)导致了P(P)即使是在同一个体内进行比较时,也会出现值,这表明泊松GLM低估了方差,而P值的直接使用会导致反保守(过度乐观)偏差。卡索夫斯基等解决了这一问题,并通过使用其他标准来调用差异职业来调整偏差。