摘要
收稿日期:2004年9月22日;2004年10月5日接受
简介
蛋白质结构域是分子进化的不同单元,通常与分子功能的特定方面有关,如催化或结合。通常,它们代表三维(3D)结构的离散单元。蛋白质序列中功能特征域的鉴定可能为其分子和细胞功能提供第一线索。
蛋白质结构域属于家族。令人眼花缭乱的功能多样性阵列,以及由明显的序列相似性分组的大量簇,可以减少到几百到几千个域超家族之间的任何地方,这取决于一个基于3D结构和/或功能相似性分组簇的积极性。在许多情况下,一个或几个搜索模型就足以唯一地识别序列数据库中一个大型、多样化超家族的所有成员。事实上,用几千个结构域模型就可以在三分之二以上的已知蛋白质序列中识别和标记结构域,如全面收集的Pfam(1). 然而,即使是像Pfam这样的紧凑集合也无法帮助为真正同源的家族创建单独的模型。蛋白质序列中重叠的区域有时会被多个模型注释。保护域数据库(CDD)还镜像了其他集合,这些集合在很大程度上与Pfam:SMART冗余(2)和COG(三). 当然,这加剧了注释问题。CD-Search资源的用户(4)可能面临多个重叠注释,有时分数非常相似,但功能关联不同。这种经常混淆的冗余是多源集合(如CDD)的必要属性,但不是所需属性。
可以采取某些明显的步骤来减少冗余,这就是我们在CDD v2.00版中开始做的。搜索模型基于蛋白质数据库中的重叠点击数进行聚类。不会显著增加集群总覆盖范围的集群成员将从Entrez的默认CDD集合中删除。我们还删除了搜索模型,其中注释了很少或没有序列,以及搜索模型似乎只针对在狭窄的系统发育谱系中发现的蛋白质和/或结构域。
然而,如果冗余能够提供更具体的功能注释,并且相关模型之间的关系清晰并向用户解释清楚,那么冗余可能是一件好事。细分领域超家族存在实际限制:大量的领域模型会影响数据库搜索时间,并且在许多情况下,实验支持的函数注释是稀疏的。对于CDD,我们采用了一个原则,即只为存在于不同生物中的古老保守结构域创建亚科。只有当成员序列的系统发育分布通过过去或更早发生的约0.5 Byr的基因重复表明域“同源”群的起源时,我们才创建亚家族。这一原则有助于我们保持我们希望的统一和可理解的粒度级别。在子家族中,我们试图从序列注释和已发表的文献中识别功能。对齐模型在整个超系列层次结构中保持一致。子家族比对中的核心模型可以映射到“父”模型中通常不太广泛的比对上,这极大地促进了更新,以包括新的代表性结构和序列。
为了识别古老的亚科以分离个体搜索模型,我们对多重序列比对进行系统发育分析并构建序列树。此过程需要相当准确的路线,我们经常会修改从外部来源导入的路线模型。在对齐管理中,我们考虑来自3D结构和结构叠加的信息,在可能的情况下,定义结构保守的核,准确地划定域边界,并解决基于序列的对齐方法和结构叠加之间的冲突(5). NCBI管理的路线符合简单的块结构,均匀对齐、无间隙、结构保守的块由未对齐区域分隔,捕获长度变化。
来自策划集和导入集的对齐模型被转换为位置特定的评分矩阵,后者被组装到搜索数据库中,用于RPS-BLAST(6).
客户尽职调查内容和访问
CDD可通过Entrez数据检索系统访问(7),并且可以作为Entrez的“域名”数据库进行查询。对功能描述中的域名和术语进行索引,并通过与其他Entrez资源(如NCBI分类数据库、PubMed®和Entrez的蛋白质数据库)的相互链接提供额外的搜索功能。预先计算的CD-蛋白质搜索记录在CDART数据库中(8),它提供了Entrez中所有蛋白质的结构域概要。预先计算的搜索结果很容易访问,并基于相似的域结构和基于重叠的hit-list的域-域链接为蛋白质-域链接、蛋白质-蛋白质链接提供数据。
CDD中的大多数域模型都是从两个外部来源(Pfam和COG)导入的。CDD还包含来自SMART的模型,以及数百个NCBI-curated域模型,可通过以“cd”开头的加入进行识别。虽然CD-Search继续将Pfam版本11.0、SMART版本4.0和COG作为单独的搜索集进行镜像,但Entrez中提供的默认“非冗余”CDD v2.01目前仅保留7255个Pfam模型中的5252个、663个SMART模型中的575个和4873个COG模型中的4101个。其余部分因冗余、无效或血统特异性而被删除。
搜索模型可用于本地RPS-BLAST安装以及CDD校准,网址为ftp://ftp.ncbi.nlm.nih.gov/pub/mdb/cdd文件/。RPS-BLAST的源代码是NCBI工具包发行版的一部分,可访问ftp://ftp.ncbi.nlm.nih.gov/工具箱.
在ENTREZ中查找域
当提交蛋白质查询进行蛋白质–蛋白质BLAST®搜索时,默认情况下会将其提交到CD-Search,并且生成的域注释以图形方式显示在中间的BLAST™结果页面上。用户可以启动包含详细结果的浏览器窗口。预先计算好的CD-Search结果也可以通过[Domains]链接在Entrez中找到蛋白质。
例如,人们可以研究植物驱动蛋白家族,例如来自Zea mays公司.CD-Search生成图形显示,如图1。查询蛋白的两个区域会收到多个看似多余的点击。中心线圈区域与CDD未固化子集中包含的各种线圈模型的得分很高。在NCBI策划的几个模型中,C端运动域得分很高。
图1。
预先计算或实时CD-Search结果可用于Entrez中的蛋白质序列。单击彩色条将启动对齐显示,将查询合并到域对齐模型中,以进行进一步分析。具有相同颜色的域注释栏被分组为多组“相关”域,这表明它们共享了许多重要的序列间隔E类-值。灰色的注释栏被归类为假定的多域模型,并被排除在域-域相邻区域之外。图的下半部分显示了域族层次结构的图形表示,给出了一个特定成员的摘要(cd01366)。
人们可以通过链接找到最佳得分匹配项“cd01366 or KISc_C_terminal”,以查看其多序列对齐中嵌入的查询序列。显而易见的是,cd01366是相关领域模型更大层次结构中的一个终端节点,总结了本例中的驱动蛋白和肌球蛋白运动领域。gi |10130006的C末端区域的次优、第三得分命中率等都是该层次结构中的其他节点。人们可能想比较分数和E类-值以了解查询序列在某个特定子组中的得分是否显著提高。
在每个子组的级别上,可以检查查询序列与该家族其他成员的相似性。我们在CD比对模型中记录了保守特征,例如活性位点或结合界面,它们的位置和残基保守模式可以在查询的上下文中进行检查。我们提供了额外的注释,例如PubMed中的文献链接和Entrez中的教科书链接,以便用户可以了解更多关于各自家族的生物学。
构建域家族层次结构和记录保守特征是NCBI管理工作的主要目标。我们将保守特征与证据一起记录下来,例如“结构证据”、举例说明结合的特定3D复合体或文献引用。我们还记录了用于决策子家族分裂的序列树,作为域家族层次结构的证据。
未来发展
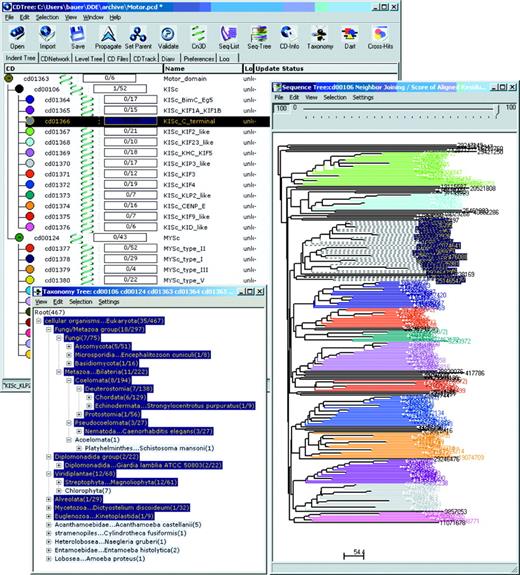
从2005年开始,我们计划分发用于构建和维护这些层次结构的软件,作为网络浏览器的辅助应用程序,使用户能够可视化CD系列层次结构、序列树和序列树中跨节点的分类多样性。图2显示了为肌球蛋白/激动素运动域家族计算的序列树,该家族在图1.
图2。
肌球蛋白/动球蛋白运动域的亚家族层次结构,相应的序列树和分类显示。突出显示了一个子家族(KISc_C_terminal),突出显示反映在序列树和分类视图中。很明显,这个子家族的成员在这个树中形成了一个由邻接算法计算的独特节点。同样明显的是,这个亚科的成员跨越了多种分类群,这表明这种特殊类型的结构域已经存在于他们共同祖先的基因组中。
如果用户希望进行调查,可以访问NCBI馆长使用的校准数据和分析算法,这将使层次结构编辑过程更加透明。层次结构编辑器和Cn3D的感兴趣用户(9)关联的基于结构的对齐编辑器将能够导入其他序列并在系统发育聚类中检查它们的行为。
我们感谢Pfam、SMART和COG的作者,他们创造了宝贵的资源,并帮助访问数据。我们也感谢美国国立卫生研究院院内研究计划的支持。我们感谢NCBI BLAST小组开发RPS-BLAST并提供持续支持。欢迎提出意见、建议和问题,请发送至info@ncbi.nlm.nih.gov.
参考文献
1贝特曼,A.,科恩,L.,杜宾,R.,芬恩,R.D.,霍利希,V.,格里菲斯·琼斯,S.,坎纳,A.,马歇尔,M.,莫克森,S(
2004
)Pfam蛋白质家族数据库。核酸研究。
,32
,138
–141. 2Letunic,I.、Copley,R.R.、Schmidt,S.、Ciccarelli,F.D.、Doerks,T.、Schultz,J.、Ponting,C.P.和Bork,P(
2004
)SMART 4.0:走向基因组数据集成。核酸研究。
,32
,142
–144. 三。Tatusov,R.L.,Fedorova,N.D.,Jackson,J.D.,Jacobs,A.R.,Kiryutin,B.,Koonin,E.V.,Krylov,D.M.,Mazumder,R.,Mekhedov,S.L.,Nikolskaya,A.N.,Rao,B.S.,Smirnov,S.,Sverdlov,A.V.,Vasudevan,S(
2003
)COG数据库:更新版本包括真核生物。BMC生物信息学
,4
,41
. 4Marchler-Bauer,A.和Bryant,S.H(
2004
)CD-Search:蛋白质域动态注释。核酸研究。
,32
,第327页
–W331。 5Marchler-Bauer,A.,Panchenko,A.R.,Ariel,N.和Bryant,S.H(
2002
)蛋白质结构域序列和结构比对的比较。蛋白质
,48
,439
–446. 6Marchler-Bauer,A.,Panchenko,A.R.,Shoemaker,B.A.,Thiessen,P.A.,Geer,L.Y.和Bryant,S.H(
2002
)CDD:一个保守的领域比对数据库,链接到领域三维结构。核酸研究。
,30
,281
–283. 7Wheeler,D.L.,Church,D.M.,Edgar,R.,Federhen,S.,Helmberg,W.,Madden,T.L.,Pontius,J.U.,Schuler,G.D.,Schriml,L.M.,Sequeira,E.,Suzek,T.O.,Tatusova,T.A.和Wagner,L(
2004
)国家生物技术信息中心数据库资源:更新。核酸研究。
,32
,35
–40. 8Geer,L.Y.、Domrachev,M.、Lipman,D.J.和Bryant,S.H(
2002
)CDART:通过结构域构建的蛋白质同源性。基因组研究。
,12
,1619
–1623. 9Wang,Y.、Geer,L.Y.、Chappey,C.、Kans,J.A.和Bryant,S.H(
2000
)Cn3D:Entrez的序列和结构视图。生物化学趋势。科学。
,25
,300
–302.
©2005,作者
核酸研究,第33卷,数据库问题©牛津大学出版社2005;保留所有权利