1引言
药物代谢影响药物分子的药代动力学和药效学,同时改变其药理活性和毒性(Kirchmair等人,2015年;马尼坎丹和纳吉尼,2018年). 在药物研发中确定药物代谢对于生产安全有效的药物至关重要。由于高分辨率质谱(HRMS)等仪器的进步,最近检测痕量代谢物的能力显著提高(Zhu等人,2011年).
然而,这些原始数据并没有直接促进药物开发。代谢物结构必须以人类可读和易于共享/搜索的方式进行阐明和处理数据记录。
仪器仪表的进步给原始数据处理带来了挑战。传统的代谢识别(MetID)工具,如手动专家系统,已不足以满足日益复杂的化学信息学领域的需求(Cuyckens,2018年;盖欣和霍尔曼,2021年). 此外,MetID科学家可能是结构解释小组的一员,这意味着软件需求经常受到相互竞争的考虑。
代谢物预测工具可能使用不同的计算方法,但它们都受到可用数据的质量和数量的限制。因此,MetID团队的效率取决于其软件工具、实验设备和数据管理策略。本文总结了MetID软件的现状,包括可用商业应用程序的概述和对未来创新的展望。
2基于软件的技术概述
MetID软件用于两项主要活动,尽管存在重叠,但许多程序同时执行这两项任务:
1.结构代谢物的实验前生成
2.数据的实验后分析
市面上有几个MetID软件包,大大提高了手动技术的效率和准确性(Kirchmair等人,2012年;Kazmi等人,2019年).
虽然这些软件工具具有独特的功能,但大多数应用程序预测非哺乳动物代谢物的能力有限。处理后的代谢数据偏向哺乳动物,这意味着该软件无法可靠地预测植物、昆虫或细菌产生的代谢产物。这对农药开发和环境毒理学有影响(Hoagland等人,2000年;Hatzios等人,2001年). 研究机构可以建立由专家知识支持的内部生物转化数据库,以提高MetID软件在专业领域的性能。
2.1实验前代谢物生成
实验前代谢物生成工具预测代谢物从头开始基于结构。实验前预测的三种最常见方法是基于规则的、基于机器学习的和基于停靠的。
这些计算策略并不相互排斥,因为它们处理代谢物预测的不同方面。只有基于规则的方法才能生成潜在代谢物的结构。其他技术评估了新陈代谢的首选部位(SoM)。许多软件包使用多种方法覆盖整个代谢物预测工作流。
2.1.1基于规则的实验前代谢物生成
顾名思义,基于规则的预测软件使用经验推导的规则来预测给定分子的生物转化。该软件通过将分子与代谢反应的实验数据库进行比较来发现可能的代谢物。或者,该算法可以识别满足不同反应类型的SoM标准的子结构,然后评估将发生的转换。重复此过程以预测下一代代谢物。
基于规则的系统提供了可以与实验观察结果进行合理比较的预测优势。研究人员可以评估特定代谢物的预测方式,让专家运用他们的知识。由于这些工具受到可用规则集的限制,因此需要软件更新和内部数据来改善这一限制。这可能是一个耗时的过程。
基于规则的MetID软件示例包括
•Nexus Meteor:一个基于知识的系统,使用以通用反应描述表示的生物转化字典。使用推理规则将这些生物变换应用于结构(Marchant等人,2008年).
•BioTransformer:预测多个系统中外源代谢的混合软件工具(Djoumbou-Feunang等人,2019年). 它使用生物转化数据库(MetXBioDB)、反应知识库和包含机器学习算法(如CypReact)的推理引擎(Tian等人,2018年)预测酶的选择性。
•GLORYx:第一阶段和第二阶段代谢物预测软件。GLORYx采用了一种混合方法,其中包括用于SoM预测的基于随机森林的机器学习算法,以及使用SMIRKS符号编码的生物转化规则的文献衍生数据库(de Bruyn Kops等人,2019年;de Bruyn Kops等人,2021年).
2.1.2基于机器学习的实验前代谢物生成
机器学习是一种基于现有知识构建预测算法的计算策略。该模型对训练数据进行处理,以找到算法中捕获的模式。初始训练完成后,可以对算法进行改进,以考虑代谢物预测的新数据(Finkelmann等人,2018;Göller等人,2020年). 此更新过程可能是自动化的,但即使在无监督模式下,也可能需要大量时间进行统计分析。
机器学习模型需要原子表示来捕获潜在反应位点的决定反应性的特征(Rydberg等人,2010年;Matlock等人,2015年). 这种计算策略在几个方面与基于规则的模型不同。机器学习模型不受预先确定的规则的限制,允许它们考虑更广泛的代谢途径。部署基于机器学习的预测软件通常需要来自先前实验MetID研究的数据,这需要资源来收集和管理。
大多数机器学习软件并没有将其计算逻辑组织成人类可读的规则,意义解释具有挑战性(Kirchmair等人,2015年;Kazmi等人,2019年). 这与MetID研究人员有关,因为代谢产物预测可能涉及研究决策和监管文件。
使用机器学习模型的MetID软件示例包括
•XenoSite服务器:提供用于可视化原子的工具,最有可能是几个重要细胞色素P450(CYP450)的代谢位点。XenoSite服务器使用神经网络机器学习模型(Matlock等人,2015年).
•MetScore:使用基于随机森林的方法预测第一和第二阶段代谢。使用量子化学衍生的分子表示进行反应性预测(Finkelmann等人,2017年).
•SMARTCyp:采用基于配体的CYP450 SoM预测方法,使用预先计算的量子机械活化能来估计位点反应性。预测会根据站点可访问性进行调整(Rydberg等人,2010年;Olsen等人,2019年).
2.1.3基于对接的实验前代谢物生成
实验前计算策略的基于对接的方法使用药物分子的3D结构信息来预测它们如何与CYP450相互作用。可以使用母体化合物进行对接,在这种情况下,最合适的姿势将指示首选的SoM(Li等人,2011年). 或者,代谢物结构可以使用混合对接/基于规则的方法对接(Tarcsay等人,2010年):
1.基于规则的代谢物生成
2.在CYP450反应位点对接代谢物
3.基于互补性选择可能的代谢物
添加的结构上下文可能会提高基于停靠的预测模型的准确性,尽管这些信息可能不可用。基于对接的模型通常仅限于CYP450,不涵盖人类肝微粒体(HLM)的其他活性。
大多数基于停靠的代谢物预测研究都没有使用单一的软件包。相反,他们使用多种工具来完成分析(Tarcsay等人,2010年;Moors等人,2011年). 这提供了灵活性,但可能会阻碍用户体验和生产力。
基于停靠的软件示例包括
•IDSite:评估蛋白质-甘氨酸复合物的能量,并使用对接工具(GLIDE)将配体放入活性位点。这与结构建模程序(PLOP)相结合,以确定结合方向并预测SoM(Li等人,2011年).
•MetaSite:采用有时称为伪锁的方法(Tyzask和Kirchmair,2019年). 该软件通过将配体结构与编码细胞色素酶活性位点“指纹”的GRID分子相互作用场对齐,预测潜在的SoM(Cruciani等人,2005年).
2.2实验后MetID工具
实验后MetID软件使用预测数据和实验数据来识别和验证代谢产物。如上所述,母体化合物的结构用于预测代谢物结构。然后应用程序评估分析数据,以确定存在哪些理论化学品。
使用实验后预测可以显著提高MetID研究的准确性和可靠性。这种精度的提高是以需要实验数据为代价的。由于实验前MetID软件的主要目标是避免不必要的代谢实验,因此实验后预测软件不应被视为替代品。实验后预测软件旨在加速代谢物数据分析并提供额外的验证水平。
•质谱:使用液相色谱-质谱、UV、荧光和放射色谱数据自动识别小分子和肽的代谢物。根据MS和MS/MS裂解模式将化学结构分配给色谱峰(特伦泽等人,2009年).
•MZmine:具有代谢物识别功能的质谱分析数据处理工具(Pluskal等人,2020年). 代谢物结构是通过复合数据库搜索确定的,该搜索可能涉及机器学习算法(SIRIUS/CSI:FingerID)的预测(杜尔科普等人,2019年).
3 MetaSense操作和功能
MetaSense(元感知)®是由ACD/Labs开发的代谢产物预测软件包,它采用基于规则和机器学习来执行实验前和实验后预测,简化了专家的审查过程。了解其功能为了解MetID软件的运行方式提供了一个有益的示例。
3.1实验前代谢物生成
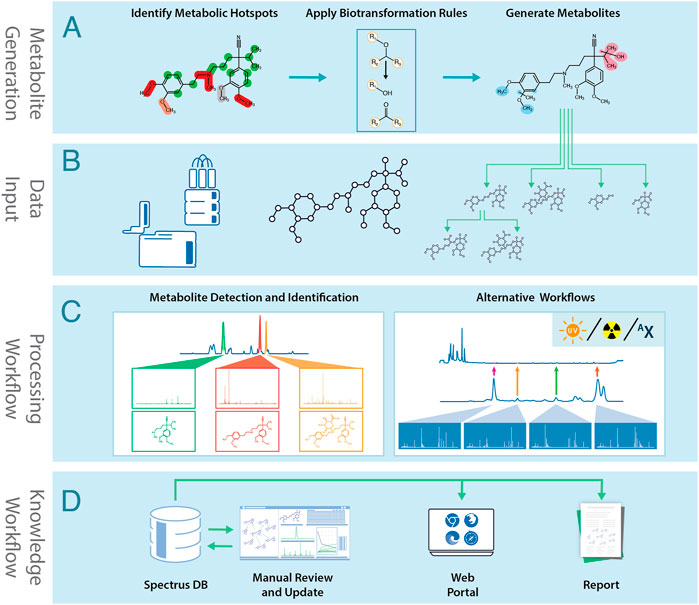
MetaSense的代谢物生成过程总结如下图1A。发动机由两个主要部件组成:
1.生物转化规则数据库将潜在SoM的化学环境映射到预期反应产物列表。生物转化规则集是从多篇评论出版物中编译而成的(Testa和Krämer,2007年a;Testa和Krämer,2007年b;Testa和Krämer,2008年;Dalvie等人,2002年)并通过分析CYP450底物及其代谢产物的内部数据库进行进一步扩展。规则根据这些反应类型进行分组:
•第一阶段水解:活性官能团的自发或酶水解裂解
•I相氧化还原反应,包括羟基化、脱烷基化、杂原子氧化、环氧化物形成和环去饱和以及随后的芳构化
•第二阶段-共轭反应,如磺化、葡萄糖醛酸化、添加谷胱甘肽和各种氨基酸
2.软点排序算法估计特定SoM的代谢反应可能性。使用的评分函数取决于反应类型。
水解和第二阶段评分基于简单的启发式,反映了SoM的整体不稳定性和基于母体化合物的物理化学特征的共轭敏感性。例如,通过预测对数来评估亲脂性降低共轭物(例如磺化、葡萄糖醛酸化)的可能性D类7.4值,对固有极性和水溶性分子的评分较低。相反,谷胱甘肽的添加纯粹是基于规则的——其靶位点是通过对一组亲电片段的亚结构搜索来确定的。
对于第一阶段的氧化还原反应,MetaSense使用基于机器学习的区域选择性预测模型,该模型与ACD/Labs Percepta平台共享。这些模型确定了由CYP450和HLM中表达的其他代谢酶催化的五种常见氧化还原反应类型的软点。
模型使用GALAS(全局,根据相似性进行局部调整)方法,该方法可以描述为两个过程的组合:
•预测感兴趣财产的分段基线QSAR模型
•基于相似性的例程(或本地模型),根据对训练集中确定的最相似化合物的基线模型性能的分析,引入额外的修正。
已经开发出一种专门的结构裂解方法,以在原子对原子的基础上解释酶的区域选择性。与使用整个分子的一张“数字图像”的传统碎裂技术不同,这种区域选择性模型根据所选的中心原子使用了几种独特的分子表示。这种碎裂方法和围绕反应中心的原子称重方案包括用于原子中心预测的GALAS方法变化(Dapkunas等人,2009年).
该模型的输出是特定原子成为HLM酶目标的概率,以及可靠性指数——基于局部相似性校正步骤的预测置信度的定量度量。可靠性指数通过证明该值与两个定量的公认模型精度指标之间的直接相关性,一致地预测质量估计(Sazonovas等人,2010年)(如MAE、RMSE)和定性(如敏感性、特异性)模型(Dapkunas等人,2009年;Didziapetris等人,2010年). 最后,将GALAS HLM区域选择性模型的两个输出进行组合,以产生范围为0到1的总体SoM得分。
一旦计算出分子中所有可能的反应位点的SoM分数,就会根据分数阈值对其进行过滤。应用生物转化规则生成拟用代谢物结构列表。可以重复此过程以生成生物转化图(BTM)。产品可以根据分子量、反应类型或自定义生物转化规则进行过滤。
3.2实验后代谢产物鉴定
MetaSense实验后工作流程总结如下图1B–D该过程从将实验数据文件和相应的结构导入处理环境开始。这可能包括来自LC/MS/MS、放射性追踪、紫外线追踪或同位素富集工作流的数据。该软件可以使用大多数主要仪器供应商的数据和代谢物预测软件,如Meteor Nexus(拉萨有限公司)、MetaSite(分子发现)或用户创建的SDFiles。
处理后,解释光谱上传至中央数据库,并自动创建BTM。科学家可以审查整个项目,并根据专家知识添加缺失的代谢物。
LC/MS痕量分离为提取离子色谱图(XIC)。预测的代谢物通过准确的质量和同位素模式与峰值相匹配。由于每个XIC可能包含来自多个代谢物的峰,通过应用碎片规则和碎片离子质量转移,利用MS/MS光谱定位生物转化位点。如果反应位点不明确,则使用Markush符号表示代谢物结构。该软件还支持数据相关采集、全离子碎片化和MSE类.
通过对照样品比较和分数质量差确定意外代谢物。由于所有数据和解释都链接在一起并存储在一起,用户可以查看原始色谱图和光谱,并将其发送到其他软件工具进行结构解释。
MetaSense提供了两个显著的功能
•自动创建BTM,手动创建很耗时。BTM和动力学图是根据结构和实验数据生成的。MetaSense使用化学智能来完善BTM,排除化学上不可行的步骤。
•在可搜索的数据库中存储分析和化学数据,包括峰面积、元数据、地图和绘图。如果发现新的代谢物,可以重新处理和更新分析数据。
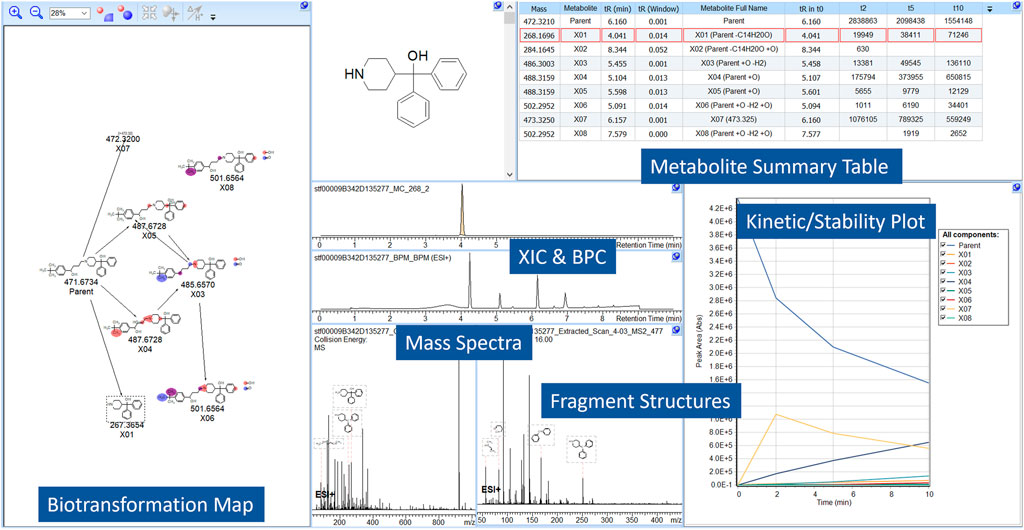
3.3 MetaSense特非那定示例
特非那定的时间过程代谢物研究如所示图2。在数据集中设置实验前代谢预测反应(阶段I/II),并使用实验后ID进行过滤。通过预测碎片的光谱分配来评估这些结构的可行性。父代或代谢物区域可视化,允许用户评估整个研究中代谢物的形成。
4讨论
MetID科学家获得的数据比以往任何时候都多。这包括高分辨率仪器的实验数据和计算机模型生成的电子数据。有效的数据处理是一项重大挑战。我们预测,生产力的突破将归功于数据管理的改进。
4.1数据的互联表示
新陈代谢研究的数据高度相互关联,但它们可以分为四个层次:原始数据、元数据、处理数据和解释。
•仪器检测器的原始数据是代谢物数据的基础。
•原始数据与元数据相关联,包括仪器条件、化学结构、模型生物、样品类型和样品制备方法。
•然后将原始数据和元数据处理成离子轨迹和综合峰值。
•然后将处理后的数据抽象为解释层,包括BTM、动力学图和其他可视化。
MetID专家必须了解这些数据层和解释层之间的联系。从原始数据自动生成BTM和动力学图可以节省时间。更高级别的抽象必须与原始数据相连接,以支持严格的分析,并确定用于报告和监管审查的证据链。
这四层数据及其连接应可供审查。大多数MetID软件不符合此标准,因为这些工具不允许在实验设计处理的数据-原始数据之间进行超链接。数字工具的创新将克服这些限制,使研究人员能够跟踪多个数据层之间的连接。
4.2分析数据管理策略
MetID软件的生产率与可用数据的数量和质量直接相关。这最终取决于研究组织的数据管理策略。因此,分析数据管理系统应被视为任何MetID项目的关键。
必须设计一个分析数据管理系统,以满足整个研究组织的需要。这需要在特定研究人员的功能需求和实际考虑(如费用、部署时间或前向兼容性)之间取得平衡。一些最相关的问题包括:
•数据存储策略:虽然从理论上来说,始终存储每一条数据是理想的,但这并不实际,也不划算。需要存储哪些功能来为未来的数据挖掘和机器学习做准备?
•可查找性:可查找数据包括足够的元数据,可以随时检索。丢失的数据通常需要重复实验,从而增加了时间和成本。通过围绕元数据管理的稳健业务实践,可以提高可查找性。
•同质文件格式:数据必须以长期可用的格式保存。数据应以一致的供应商中性格式存储,以减少访问障碍,促进互操作性,并简化数据比较。
•当前需求与未来扩展:设计用于满足单个实验室需求的本地数据库可以以最小的开销快速部署。这样的分散系统可能不利于机器学习项目或实验室间协作。企业系统需要更多的部署和管理工作,但可以设计为促进数据科学项目。
•遗留数据的作用:研究机构积累了大量数据。根据当前最佳实践,遗留数据可能尚未处理或基于数据。该数据应该单独管理,还是可以与新数据一起使用?是否需要重新格式化、重新处理或其他形式的维护?
每个研究功能都有其优先考虑的特定数据库功能。MetID科学家受益于包含BTM和汇总表的可搜索数据库。
5展望未来
由于仪器和软件的改进,代谢研究的准确性和效率显著提高。下一个挑战是缩放处理和预测工具,以管理现代分析设备生成的数据量。
应开发应用程序来表示原始数据、元数据、处理数据和解释之间的联系。我们需要专用工具来了解这四个数据层之间的关系。
机器解释依赖于有效地整理和管理数据。研究机构需要投资于支持成功MetID计算机模型的系统。药物代谢预测和鉴定方面的创新将指导科学家开发更安全、更有效的药物。
数据可用性声明
文章中包含了研究中提出的原始贡献,可以向通讯作者进行进一步的询问。
作者贡献
列出的所有作者都对这部作品做出了实质性、直接和智力上的贡献,并批准出版。
利益冲突
作者受雇于化学软件公司ACD/Labs。ACD/Labs是MetaSense和Percepta的开发人员。ACD/Labs还提供分析数据管理服务。
出版商备注
本文中表达的所有声明仅为作者的声明,不一定代表其附属组织的声明,也不一定代表出版商、编辑和审稿人的声明。任何可能在本文中进行评估的产品,或制造商可能提出的索赔,都不受出版商的保证或认可。
致谢
我们要感谢罗伯特·霍夫曼(Robert Hoffmann)为说明我们的数据所做的工作。
工具书类
Cruciani,G.、Carosati,E.、De Boeck,B.、Ethirajulu,K.、Mackie,C.、Howe,T.等人(2005年)。代谢位点:从化学家的角度理解人类细胞色素的代谢。医学化学杂志。48 (22), 6970–6979. doi:10.1021/jm050529c
PubMed摘要|CrossRef全文|谷歌学者
Cuyckens,F.(2018年)。药物代谢和药代动力学中的质谱分析:当前趋势和未来展望。快速通讯。质谱。33(S3),90-95。doi:10.1002/rcm.8235
PubMed摘要|CrossRef全文|谷歌学者
Dalvie,D.K.、Kalgutkar,A.S.、Khojasteh-Bakht,S.C.、Obach,R.S.和O'Donnell,J.P.(2002)。五元芳香杂环的生物转化反应。化学。研究毒物。15 (3), 269–299. 数字对象标识代码:10.1021/tx015574b
PubMed摘要|CrossRef全文|谷歌学者
Dapkunas,J.、Sazonovas,A.和Japertas,P.(2009)。人类CYP3A4和CYP2D6代谢位点的概率预测。薪酬福利6 (11), 2101–2106. doi:10.1002/cbdv.200900078
PubMed摘要|CrossRef全文|谷歌学者
de Bruyn Kops,C.、Šícho,M.、Mazzolari,A.和Kirchmair,J.(2021)。GLORYx:预测异种生物第一阶段和第二阶段生物转化产生的代谢物。化学。研究毒物。34 (2), 402–299. doi:10.1021/acs.chemrestox.0c00224
CrossRef全文|谷歌学者
de Bruyn Kops,C.,Stork,C.,Šícho,M.,Kochev,N.,Svozil,D.,Jeliazkova,N.等人(2019年)。GLORY:基于预测代谢位点的可能细胞色素P450代谢物结构生成器。前面。化学。7, 402. doi:10.3389/fchem.2019.00402
PubMed摘要|CrossRef全文|谷歌学者
Didziapetris,R.、Dapkunas,J.、Sazonovas,A.和Japertas,P.(2010年)。CYP3A4抑制剂虚拟筛选的可训练构效关系模型。J.计算。辅助分子设计。24 (11), 891–906. doi:10.1007/s10822-010-9381-1
PubMed摘要|CrossRef全文|谷歌学者
Djoumbou-Feunang,Y.、Fiamoncini,J.、Gil-de-la-Funte,A.、Greiner,R.、Manach,C.和Wishart,D.S.(2019年)。生物变压器:小分子代谢预测和代谢物鉴定的综合计算工具。J.化学信息11, 2. doi:10.1186/s13321-018-0324-5
PubMed摘要|CrossRef全文|谷歌学者
Dührkop,K.、Fleischauer,M.、Ludwig,M.、Aksenov,A.A.、Melnik,A.V.、Meusel,M.等人(2019)。SIRIUS 4:将串联质谱转换为代谢物结构信息的快速工具。自然方法16 (4), 299–302. doi:10.1038/s41592-019-0344-8
PubMed摘要|CrossRef全文|谷歌学者
Finkelmann,A.R.、Goldmann,D.、Schneider,G.和Göller,A.H.(2018)。MetScore:细胞色素P450酶以外的代谢预测位点。化学医药化学13 (21), 2281–2289. doi:10.1002/cmdc.201800309
PubMed摘要|CrossRef全文|谷歌学者
Finkelmann,A.R.、Göller,A.H.、Schneider,G.和Göllar,A.H。(2017年)。基于从头算衍生原子表示的代谢位点预测。化学医药化学12 (8), 606–612. doi:10.1002/cmdc.201700097
PubMed摘要|CrossRef全文|谷歌学者
Géhin,C.和Holman,S.W.(2021)。2020年药物高分辨率质谱应用进展:一个全新的信息时代。分析。科学。副词。2, 142–156. doi:10.1002/ansa.2002000149
CrossRef全文|谷歌学者
Göller,A.H.、Kuhnke,L.、Montanari,F.、Bonin,A.、Schnecker,S.、ter Laak,A.等人(2020年)。拜耳的In Silico ADMET平台:过去二十年的机器学习之旅。药物研发。今天25 (9), 1702–1709. doi:10.1016/j.drudis.2020.07.001
PubMed摘要|CrossRef全文|谷歌学者
Hatzios,K.K.、Hall,J.C.、Hoagland,R.E.和Zablotowicz,R.M.(2001)。农药在植物和微生物中的生物转化:异同美国华盛顿:美国化学学会.doi:10.1021/bk-2001-0777.fw001
CrossRef全文|谷歌学者
Hoagland,R.E.、Zablotowicz,R.M.和Hall,J.C.(2000)。《植物和微生物中的农药代谢:概述》植物和微生物中的农药生物转化:相似性和差异性编辑J.C.Hall、R.E.Hoagland和R.M.Zablotowocz(美国华盛顿:美国化学学会), 2–27. doi:10.1021/bk-2001-0777.ch001
CrossRef全文|谷歌学者
Kazmi,S.R.、Jun,R.、Yu,M.S.、Jung,C.和Na,D.(2019年)。用于预测药物代谢和命运的硅胶方法和工具:综述。计算。生物医学。106, 54–64. doi:10.1016/j.compbiomed.2019.01.008
PubMed摘要|CrossRef全文|谷歌学者
Kirchmair,J.、Göller,A.H.、Lang,D.、Kunze,J.和Testa,B.、Wilson,I.D.等人(2015年)。预测药物代谢:实验和/或计算?Nat.Rev.药物发现。14 (5), 387–404. doi:10.1038/nrd4581
PubMed摘要|CrossRef全文|谷歌学者
Kirchmair,J.、Williamson,M.J.、Tyzack,J.D.、Tan,L.、Bond,P.J.、Bender,A.等人(2012年)。代谢的计算预测:位点、产物、SAR、P450酶动力学和机制。化学杂志。信息模型。52 (3), 617–648. doi:10.1021/ci200542m
PubMed摘要|CrossRef全文|谷歌学者
Li,J.,Schneebeli,S.T.,Bylund,J.、Farid,R.和Friesner,R.A.(2011年)。IDSite:预测P450介导药物代谢的准确方法。化学杂志。理论计算。7 (11), 3829–3845. doi:10.1021/ct200462q
PubMed摘要|CrossRef全文|谷歌学者
Manikandan,P.和Nagini,S.(2018年)。细胞色素P450的结构、功能及临床意义。Cdt公司19 (1), 38–54. doi:10.2174/1389450118666170125144557
PubMed摘要|CrossRef全文|谷歌学者
Marchant,C.A.、Briggs,K.A.和Long,A.(2008年)。在用于共享毒性和新陈代谢数据和知识的硅胶工具中:Derek用于Windows、Meteor和Vitic。毒物。机械。方法18, 177–187. 网址:10.1080/15376510701857320
PubMed摘要|CrossRef全文|谷歌学者
Matlock,M.K.、Hughes,T.B.和Swamidass,S.J.(2015)。XenoSite服务器:代谢预测工具的Web可用站点。生物信息学31 (7), 1136–1137. doi:10.1093/bioinformatics/btu761
PubMed摘要|CrossRef全文|谷歌学者
Moors,S.L.C.、Vos,A.M.、Cummings,M.D.、Van Vlijmen,H.和Ceulemans,A.(2011年)。基于结构的细胞色素P450 2D6代谢预测位点。医学化学杂志。54, 6098–6105. doi:10.1021/jm2006468
PubMed摘要|CrossRef全文|谷歌学者
Olsen,L.、Montefiori,M.、Tran,K.P.和Jörgensen,F.S.(2019年)。SMARTCyp 3.0:增强型细胞色素P450代谢位点预测服务器。生物信息学35 (17), 3174–3175. doi:10.1093/bioinformatics/btz037
PubMed摘要|CrossRef全文|谷歌学者
Pluskal,T.、Korf,A.、Smirnov,A.、Schmid,R.、Fallon,T.R.、Du,X.等人(2020年)。第7章。使用MZmine进行代谢组学数据分析。雷达散射截面8, 232–254. doi:10.1039/9781788019880-00232
CrossRef全文|谷歌学者
Rydberg,P.、Gloriam,D.E.、Zaretzki,J.、Breeman,C.和Olsen,L.(2010年)。SMARTCyp:一种预测细胞色素P450介导的药物代谢的2D方法。ACS医学化学。莱特。1 (3), 96–100. 数字对象标识码:10.1021/ml100016x
PubMed摘要|CrossRef全文|谷歌学者
Sazonovas,A.、Japertas,P.和Didziapetris,R.(2010)。急性毒性(LD50)分析中预测的可靠性估计和模型适用范围评估。SAR QSAR环境。物件。21 (1–2), 127–148. doi:10.1080/10629360903568671
PubMed摘要|CrossRef全文|谷歌学者
阿拉巴马州塔尔西。,Kiss,R.和Keserü,G.M.(2010年)。细胞色素P450 2C9代谢预测位点:基于知识的对接方法。J.计算。辅助分子设计。24 (5), 399–408. 数字对象标识代码:10.1007/s10822-010-9347-3
PubMed摘要|CrossRef全文|谷歌学者
Testa,B.和Krämer,S.D.(2007a)。药物代谢生物化学导论。化学。生物多样性。4 (3), 257–405. doi:10.1002/cbdv.200790032
PubMed摘要|CrossRef全文|谷歌学者
Testa,B.和Krämer,S.D.(2007b)。药物代谢生物化学导论。薪酬福利4 (9), 2031–2122. doi:10.1002/cbdv.200790169
CrossRef全文|谷歌学者
Testa,B.和Krämer,S.D.(2008年)。药物代谢的生物化学——导论。薪酬福利5 (11), 2171–2336. doi:10.1002/cbdv.200890199
CrossRef全文|谷歌学者
Tian,S.、Djoumbou-Feunang,Y.、Greiner,R.和Wishart,D.S.(2018年)。CypReact:用于预测人类细胞色素P450酶的硅内反应物的软件工具。化学杂志。信息模型。58, 1282–1291. doi:10.1021/acs.jcim.8b00035
PubMed摘要|CrossRef全文|谷歌学者
Trunzer,M.、Faller,B.和Zimmerlin,A.(2009年)。使用MetaSite和LC-MS/MS验证在早期发现阶段进行代谢软点识别和化合物优化。医学化学杂志。52 (2), 329–335. doi:10.1021/jm8008663
PubMed摘要|CrossRef全文|谷歌学者
Tyzack,J.D.和Kirchmair,J.(2019年)。预测细胞色素P450代谢用于药物发现的计算方法和工具。化学。生物药物设计。93 (4), 377–386. doi:10.1111/cbdd.13445
PubMed摘要|CrossRef全文|谷歌学者
Zhu,M.、Zhang,H.和Humphreys,W.G.(2011)。药物代谢产物的高分辨率质谱分析和鉴定。生物学杂志。化学。286 (29), 25419–25425. doi:10.1074/jbc.r110.200055
PubMed摘要|CrossRef全文|谷歌学者