数字
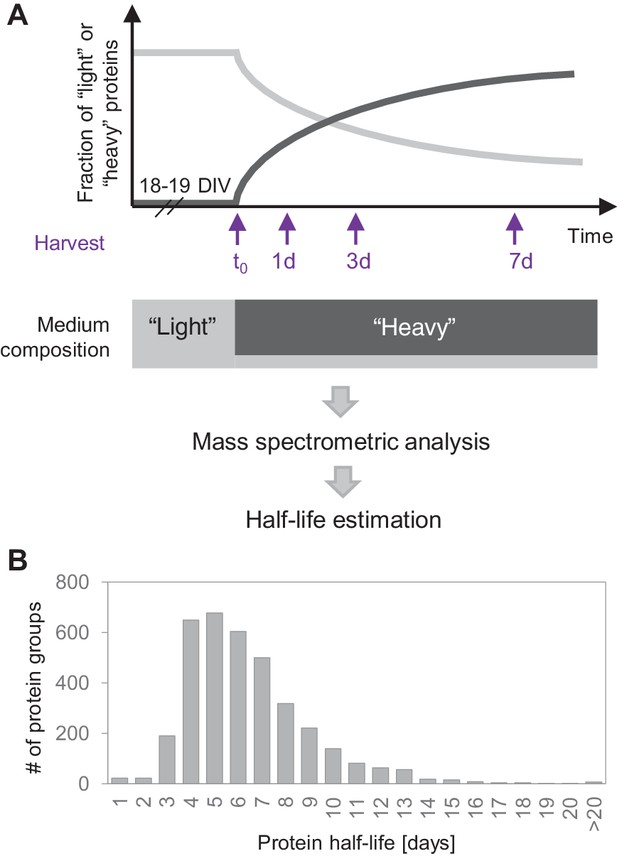
神经元中的蛋白质周转。
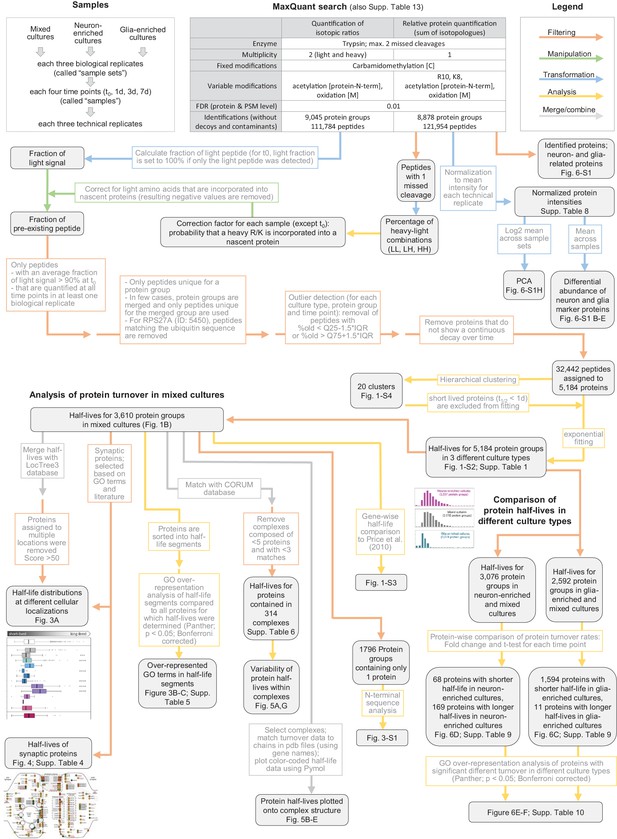
动态SILAC样本的数据处理工作流。
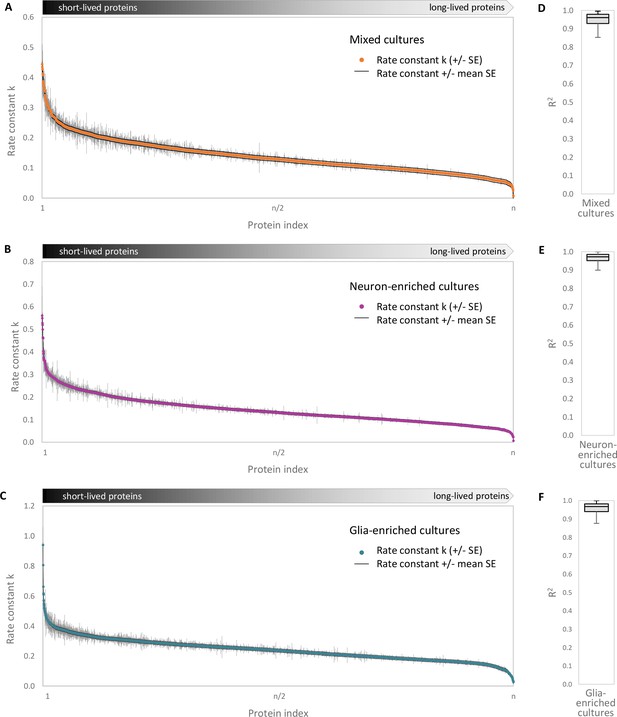
营业额估算的准确性。
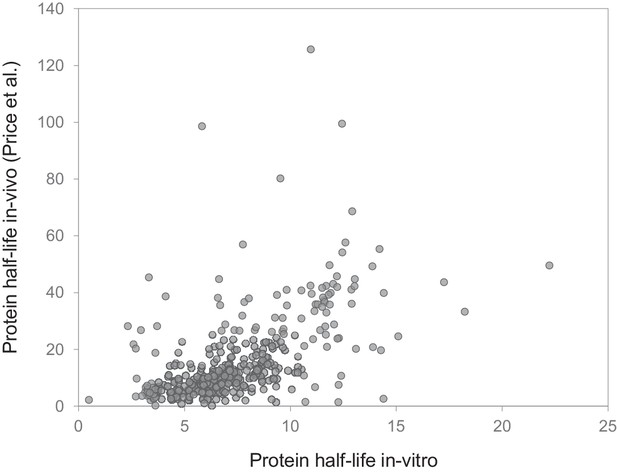
与的比较 体内 研究。
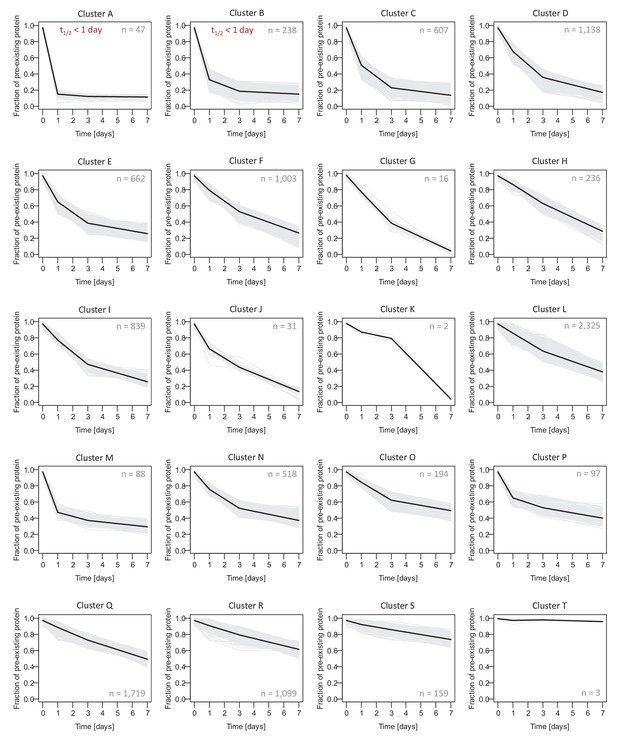
蛋白质衰变簇。
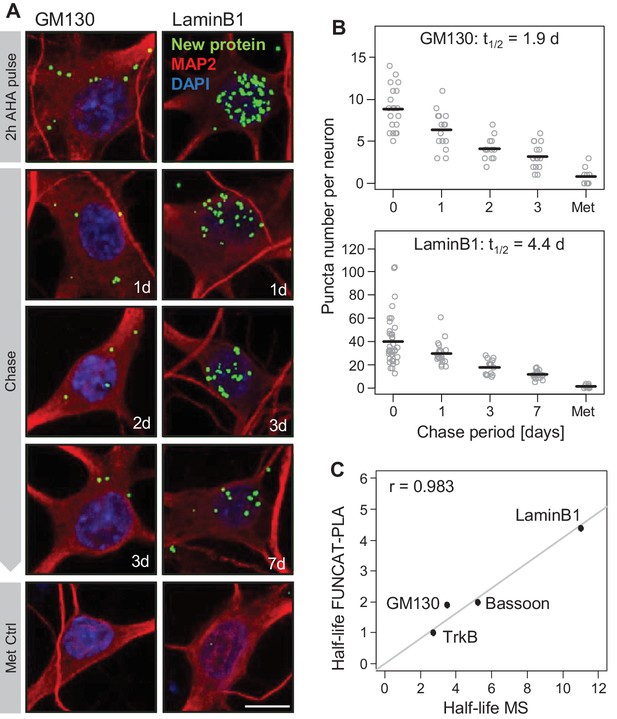
通过代谢标记和可视化验证质谱结果。
-
图2-源数据1 FUNCAT-PLA结果。 -
https://doi.org/10.7554/eLife.34202.008
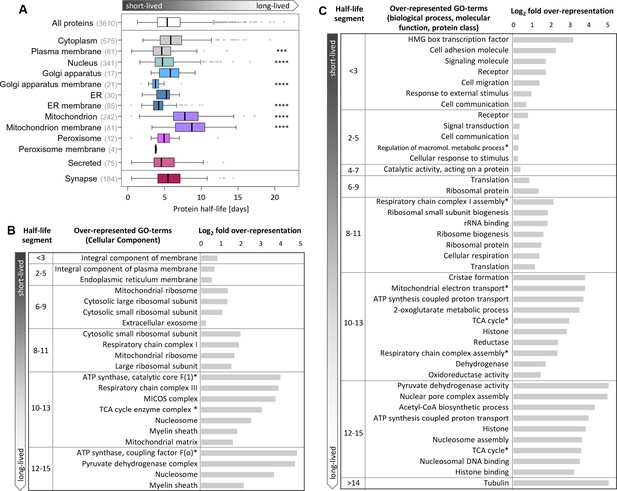
具有不同细胞位置和功能的蛋白质的半衰期。
-
图3-源数据1 蛋白质在不同亚细胞定位下的半衰期。 -
https://doi.org/10.7554/eLife.34202.011 -
图3-源数据2 半衰期段的GO分析。 -
https://doi.org/10.7554/eLife.34202.012
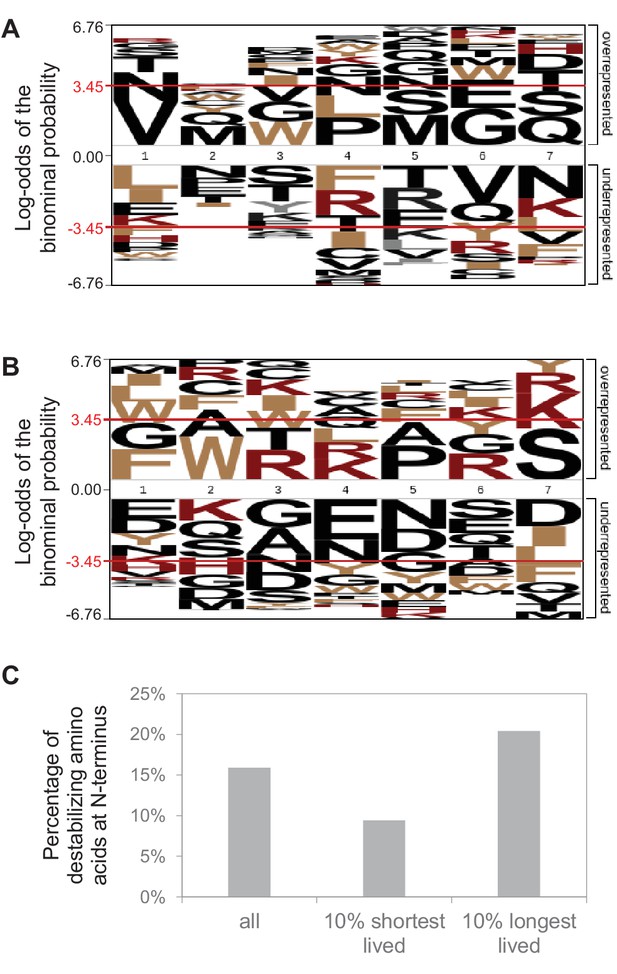
N末端序列分析。
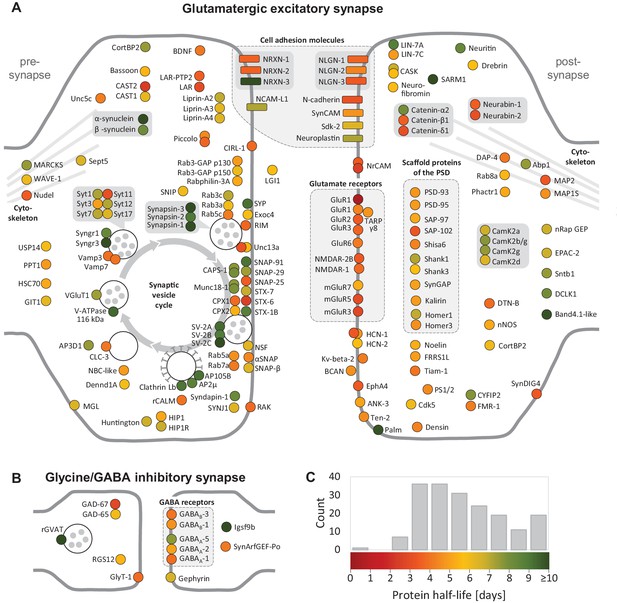
突触蛋白的半衰期。
-
图4——源数据1 突触蛋白的半衰期。 -
https://doi.org/10.7554/eLife.34202.014
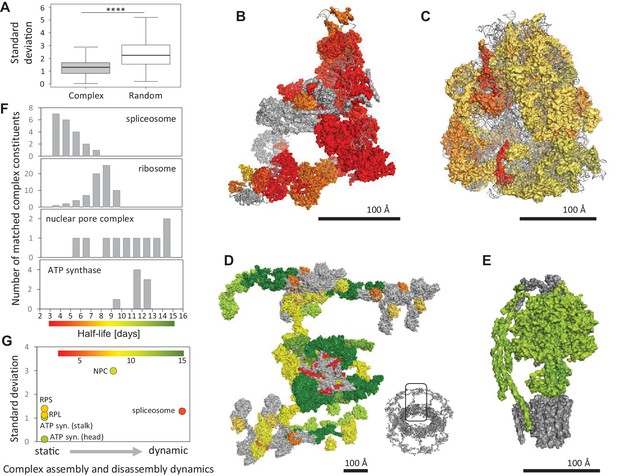
多蛋白复合物中的蛋白质半衰期。
-
图5-源数据1 蛋白质复合物成分的半衰期。 -
https://doi.org/10.7554/eLife.34202.017 -
图5-源数据2 核糖体、ATP合成酶、剪接体和核孔复合体成分的半衰期。 -
https://doi.org/10.7554/eLife.34202.018
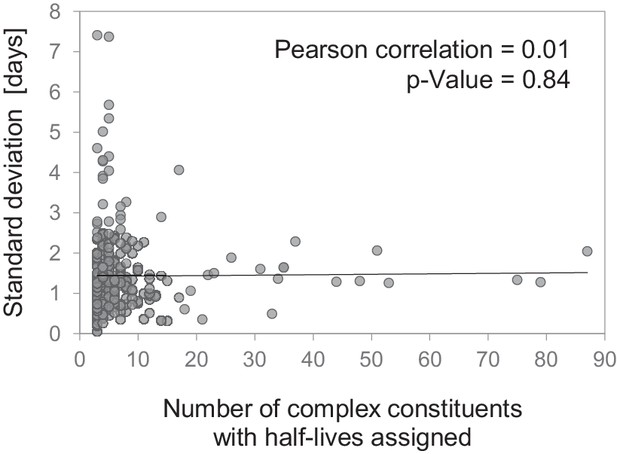
蛋白质复合体内半衰期变异性与复合体大小的相关性。
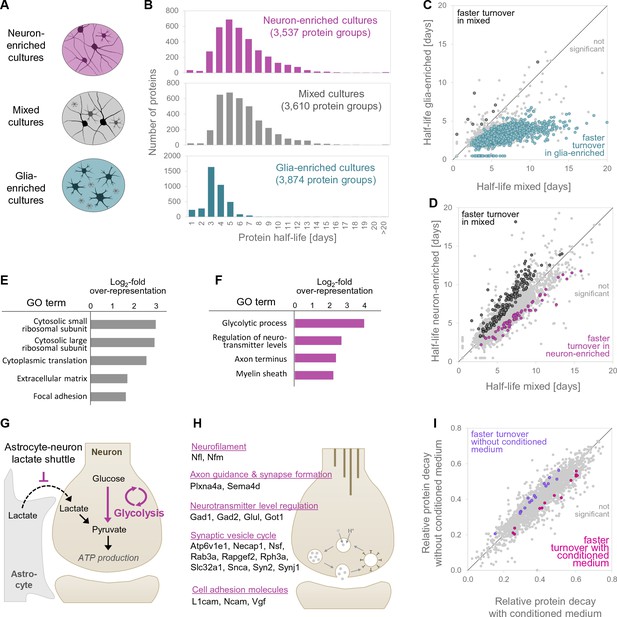
不同细胞类型在不同细胞环境中的蛋白质周转率。
-
图6——源数据1 不同培养类型的相对蛋白质丰度。 -
https://doi.org/10.7554/eLife.34202.025 -
图6——源数据2 不同培养类型之间的蛋白质半衰期比较。 -
https://doi.org/10.7554/eLife.34202.026 -
图6——源数据3 混合培养物和富含神经元培养物中不同周转率蛋白质的GO分析。 -
https://doi.org/10.7554/eLife.34202.027 -
图6——源数据4 在有条件培养基和无条件培养基的富含神经的培养物中具有不同周转率的蛋白质。 -
https://doi.org/10.7554/eLife.34202.028
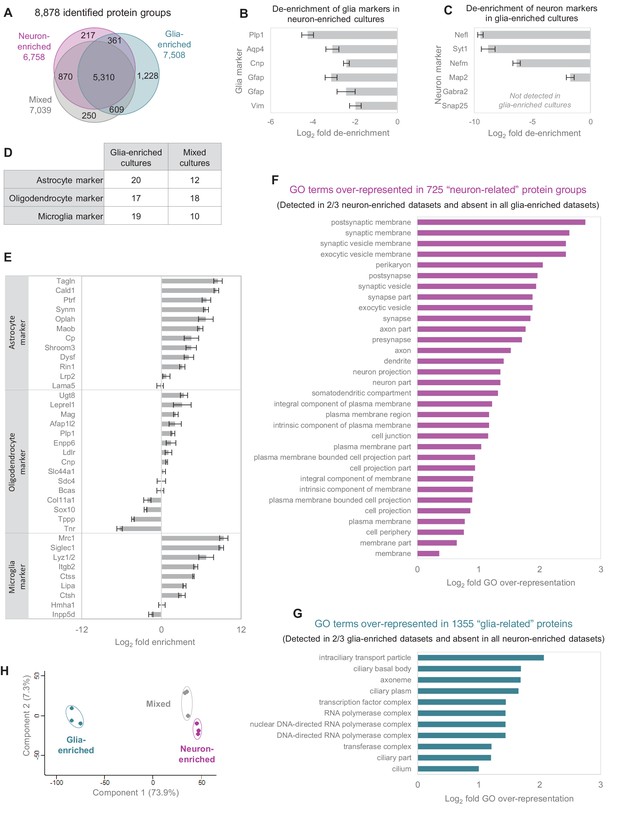
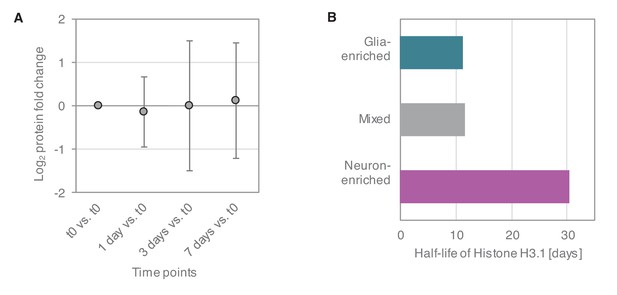
不同文化类型的比较。
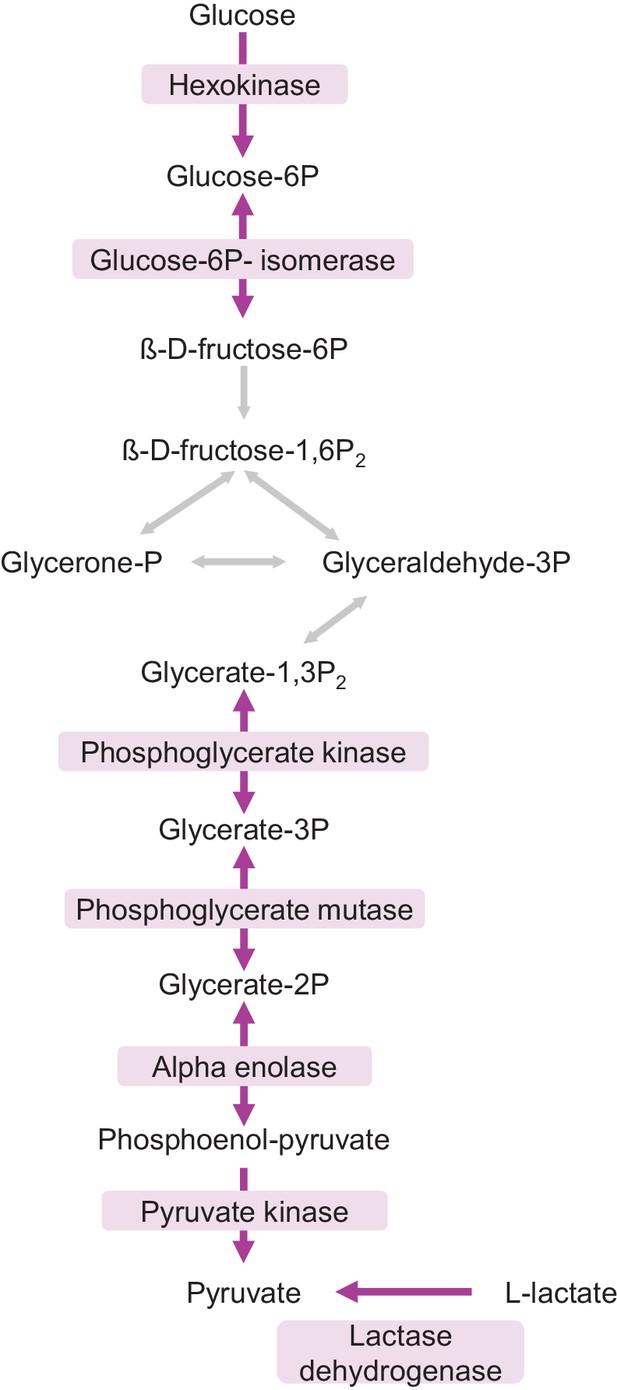
在富含神经元的培养物中,糖酵解酶的蛋白质周转更快。
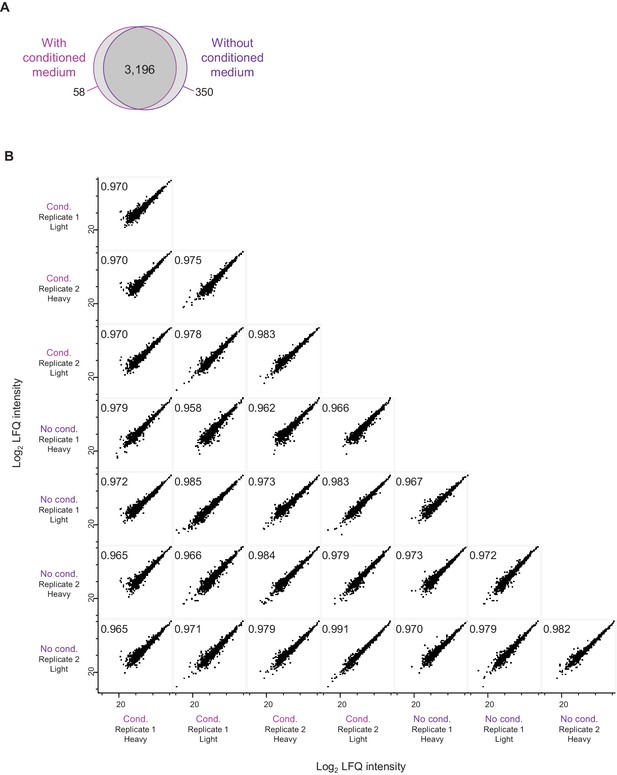
有条件培养基和无条件培养基的富含神经细胞培养物的比较。
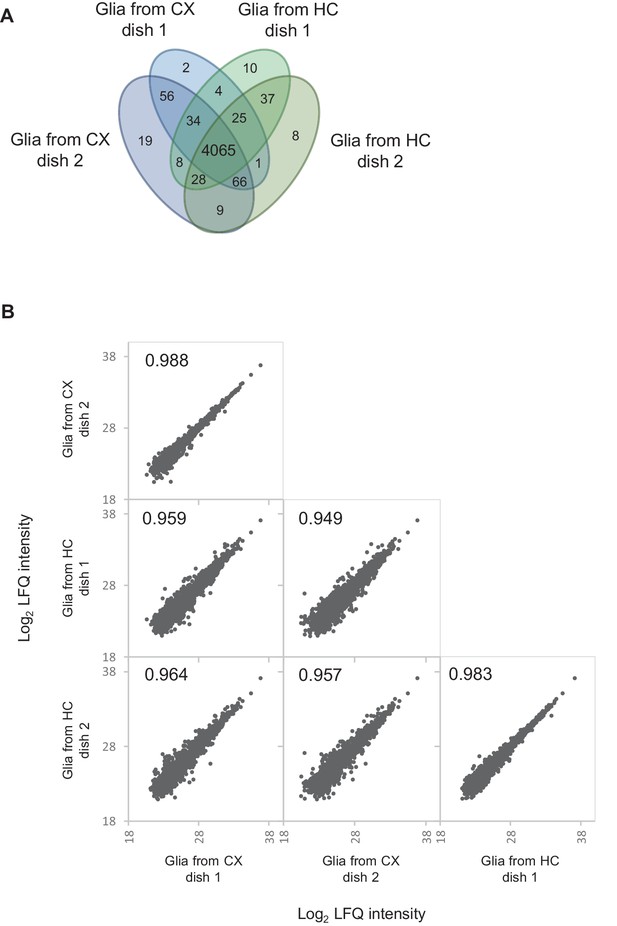
从皮层和海马制备的富含胶质细胞培养物的蛋白质组比较。
原代培养中细胞分裂的估计。
其他文件
-
补充文件1 所有培养类型中所有蛋白质的半衰期、速率常数、标准误差和测定系数。 -
https://doi.org/10.7554/eLife.34202.029 -
补充文件2 MS参数。 -
https://doi.org/10.7554/eLife.34202.030 -
补充文件3 示例表和MaxQuant参数集。 -
https://doi.org/10.7554/eLife.34202.031 -
透明的报告表 -
https://doi.org/10.7554/eLife.34202.032





