1引言
由于癌症是一种异质性疾病,正确识别癌症亚型对准确预后和改善治疗至关重要。随着高通量平台的发展,子类型方法已转向多组学集成,以便从整体角度区分亚型,并考虑不同分子水平(mRNA、甲基化等)的现象。公共存储库中积累了大量的分子数据,包括癌症基因组图谱数据集(TCGA)(1),基因组数据共享数据门户(GDC)(2),国际乳腺癌联合会(METABRIC)分子分类学(三)和英国生物银行(4). 这就需要强大而快速的分析方法来利用大型多组学数据集进行更准确的子类型发现。
目前用于多组学整合和癌症分型的方法可以根据其整合策略分为四类。第一种策略是将不同类型的数据连接到单个矩阵中,然后使用连接的数据对患者进行分区。例如,用户可以将多个数据类型(例如mRNA、甲基化、miRNA等)标准化并连接到一个矩阵中,然后应用为单组分分析开发的众所周知的方法,例如ConsensusClusterPlus(5),以确定子类型。这种方法简单且计算效率高。然而,它们并没有考虑到数据的异质性,例如,不同的数据类型可能具有不同的规模、维度,并且可能需要不同的规范化过程。
第二种策略是将多元经济数据建模为统计模型的混合物。此类别中的方法包括LRACluster(6)、rMKL-LPP(7)、iClusterPlus(8)、iClusterBayes(9),其他(10)、SBC(11),密件抄送(12),中频(13)、JIVE(14)、MCIA(15),moCluster(16)和sMBPLS(17). 这些方法通常最大化联合似然函数以确定模型参数和子类型。虽然这些方法在统计上是合理的,但它们需要估计大量的参数,而这些参数往往会导致过拟合和高计算复杂度。因此,在进行统计分析之前,通常会添加一个基因过滤或数据转换步骤。
第三种策略是将所有数据类型投影到联合潜在空间中。用于该策略的一种常见技术是非负矩阵分解。此类方法包括MvNMF(18),多NMF(19),国际NMF(20),国际NMF(21),加入NMF(22). 另一种方法是MCCA(23)它执行相关性分析,然后将相关性矩阵连接成一个单独的矩阵。将数据投影到关节空间后,进行聚类分析以确定最终的子类型。与第二种策略类似,这类方法通常具有过度的计算复杂性,无法应用于整个基因组规模。因此,基因过滤是数据处理中必不可少的一步。
第四种策略也称为基于相似性的策略。这类方法包括SNF(24)、PSDF(25)、PFA(26),IS-K含义(27)、NEMO(28),PIN码(29,30)、SCFA(31)和CIMLR(32). 这些方法首先计算每个数据类型的成对连通矩阵,该矩阵表示患者之间的相似性/连通性。然后将连通矩阵融合到一个可用于最终聚类的相似矩阵上。尽管相似矩阵功能强大,但它需要一个二次存储空间。当样本数量增加时,这是有问题的。正如我们将在分析中演示的那样,这些方法无法分析成千上万个样本的数据。
在这里,我们介绍了使用随机变换(SMRT)进行多组分型,这是一种用于癌症分型和大数据分析的新方法。与现有软件相比,该方法具有重要优势:(i)它允许研究人员在几分钟内分析数十万个样本,(ii)它可以将不同数量的患者的数据类型集成,(iii)集成和分析不同类型的不匹配数据的能力,以及(iv)web应用程序提供了一个方便的数据分析管道。我们还提高了基于集成的扰动聚类的效率,以支持对具有内存约束的机器的分析。我们对37个TCGA和两个METABRIC数据集的广泛分析表明,SMRT在识别具有显著不同生存特征的亚型方面比最先进的亚型方法更准确。此外,我们用大数据进行的模拟表明,SMRT比现有方法更快,可扩展性更高。具体来说,SMRT能够在几分钟内分析数十万个样本。
2材料和方法
2.1 SMRT管道
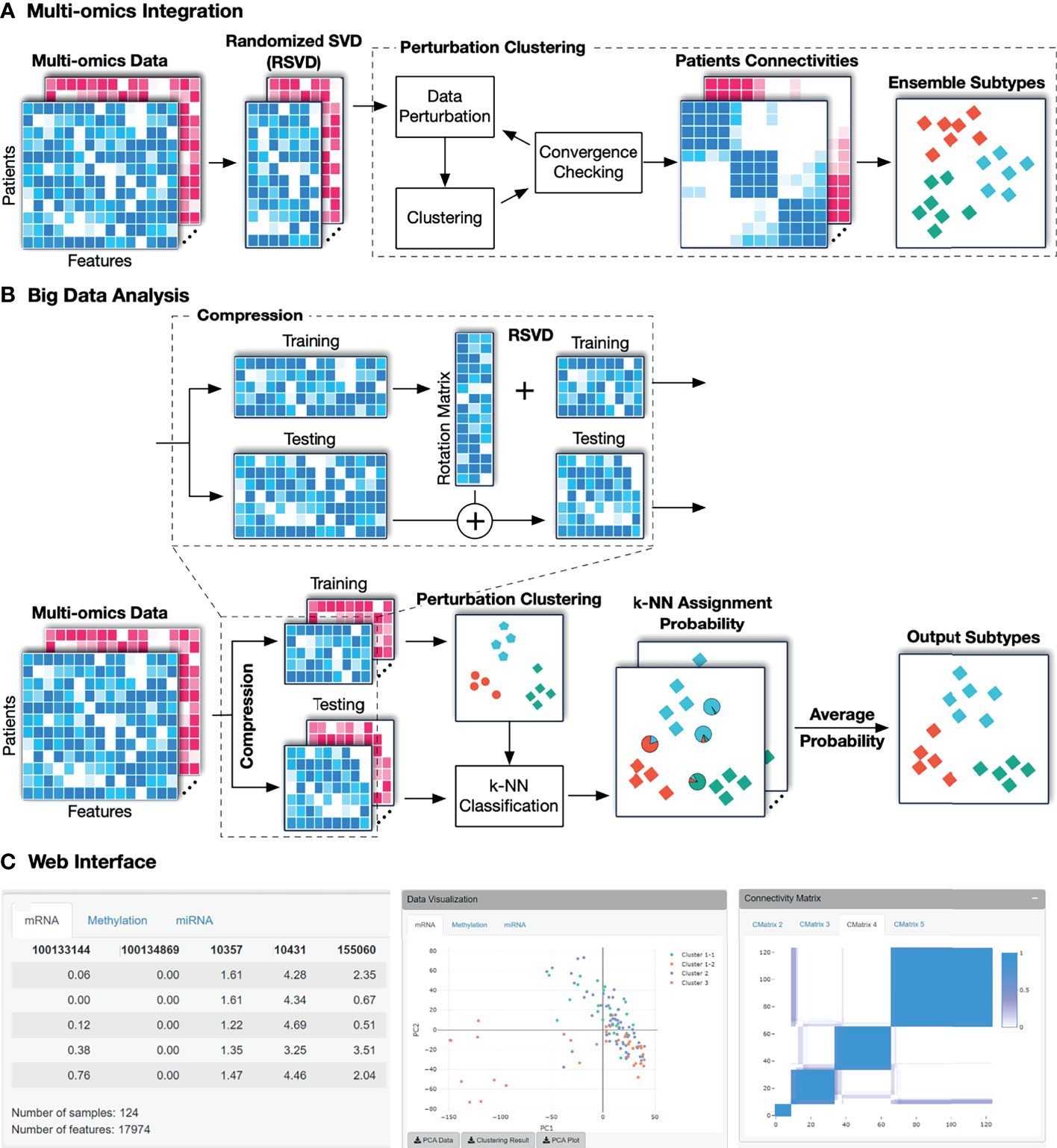
SMRT的总体工作流程如所示图1。此工作流为大数据和中等大小的数据提供了两种不同的分析管道。在第一种情况下,给定一个大小适中的多组学数据集(例如,小于2000个样本),SMRT执行以下子类型划分。它首先使用随机奇异值分解(RSVD)将每个数据类型投影到低维空间上,然后执行扰动聚类(PINS)(29,30)以确定每个数据级别中的子类型。它还为每个数据类型构建了一个成对连接矩阵,表示患者之间的连接(参见补充第5节SMRT和PINS之间的差异)。接下来,该方法将连通性矩阵组合成单个相似性矩阵,然后使用多个基于相似性的方法集成来确定最终的子类型。在第二种情况下,当数据有2000多个样本时,SMRT将数据分为两个不同的患者集:一个采样集和一个传播集。然后,它对采样集执行分型,然后将传播集中的患者分配给已识别的亚型。请注意,选择数字2000是为了平衡该方法的准确性和时间复杂性。此中等数量的样本允许SMRT在有限的内存中执行快速准确的分析(请参阅补充第3节). 我们的模拟研究表明,当我们改变这个数字时,结果不会改变。但是,用户在使用R包时可以自由更改此参数。以下是每个分析模块的描述。
2.2使用随机奇异值分解进行降维
这一步的目标是使用随机奇异值分解(RSVD)将多组学数据投影到低维空间。对于具有数十万个维度的数据(例如,Illumina 450k),此步骤大大降低了所需的计算能力,同时保持了聚类精度。让我们表示X(X)∈ ℝn个×米作为输入矩阵,其中n个是样本/患者数量,以及米是基因/特征的数量。简单地说,RSVD方法从生成随机投影矩阵开始P(P)∈ ℝ米×第页标准正态分布,其中第页≪米。然后进行投影X(X) ∈ ℝn个×米到的列空间P(P)得到矩阵Z轴这样的话Z轴=XP(极限编程).由于随机投影,Z轴和X(X)将具有大致相同的主列(功能)。现在,我们可以得到正交化矩阵问属于Z轴通过使用二维码分解,其中问大小与相同Z轴属于n个×第页。在下一步中,该方法预测X(X)进入较小的空间以获得矩阵Y(Y)∈ ℝ第页×米这样的话Y(Y)=问^T型*X(X)然后计算奇异值分解(SVD)Y(Y)作为Y(Y)=U∑V(V)*使用传统的SVD方法(33).U型和V(V)矩阵最多只能保持第页特征向量的大小U型是第页×第页以及V(V)*是米 ×第页最后,将原始矩阵的低秩旋转数据X(X)可以使用以下公式计算:X(X)′ =十五*.
实际上,RSVD比传统SVD更快,所需内存更少。为了进一步加快我们的方法,我们实现了RSVD的并行版本,该版本可以有效地利用现代处理器中可用的多个内核。请注意,当输入数据较大时(例如,超过2000个样本),我们不会对整个输入执行RSVD。相反,我们将数据分为两组患者:采样集和传播集。我们首先对采样集执行RSVD,然后将原始数据矩阵(采样集和传播集)投影到采样集将其与从采样集的RSVD获得的旋转矩阵相乘。此实现允许我们最多在几秒钟内执行SVD,即使对于包含数十万个样本和功能的数据集也是如此。
此模块的输出是多个矩阵-每个数据类型一个矩阵。在每个矩阵中,行表示患者,而列表示主成分(PCA)。这些矩阵将作为下一个模块的输入:扰动聚类,将在下一节中描述。这将计算扰动连接性矩阵并确定子类型。
2.3使用一种数据类型的子类型发现
给定单一数据类型,SMRT利用我们先前开发的扰动聚类(PINS)(29,30)对数据进行分区。简单地说,我们扰动数据(通过添加高斯噪声)并重复划分患者(默认情况下使用k-means)。对于每个分区,我们构建一个0和1的成对连接矩阵,其中1表示两个患者属于同一集群,否则为0。通过多次扰动和聚类数据,我们获得了多个连接矩阵,这些矩阵表示患者对之间的连接的稳定性。最后,我们选择对数据扰动最稳定的分区。该算法自动确定集群和患者子组的数量。
当样本数较大时,扰动聚类变得缓慢且内存效率低下。扰动聚类算法依赖于大小的成对连通性n个×n个用于群集(n个是患者数量)。当样本数量增加时,该方法的时间和空间复杂性(运行时间和内存使用)呈二次增长。因此,当样本数量较大时(默认设置为n个>2000年),我们对原始数据进行亚抽样处理,以获得2000名患者/样本的子集。接下来,我们将数据转换到低维空间,并使用扰动聚类来划分这些患者。在这一步之后,2000名患者中的每一位都有一个亚型。让我们将这组选定的2000名患者称为采样集。下一步是确定其余患者的亚型,称为传播集为此,我们使用快速k近邻搜索算法(FKNN)(34,35)将传播集中的每个患者分配给采样集中的一个子类型。简单地说,FKNN方法计算新患者到k个样本集中最近的患者。接下来,FKNN方法使用投票计数对新患者进行分类(即,它选择了在k个邻居)。默认情况下,k个使用Elbow方法对使用5倍交叉验证的采样集进行确定。采样集被随机分成5个相等的较小集。在每一轮中,将4个集合的组合用作训练集,另一个集合用作KNN算法的验证集k个范围从5到最大50。这个k个产生最低平均分类错误率的将被用作最佳分类错误率k个。但是,用户也可以自由修改此参数的值。补充第6节提供了有关使用弯头方法性能的更多详细信息与使用固定数量的k个.
需要注意的是,数据的维数可能很高,从而减缓距离计算和邻居查找的过程。因此,我们不是计算原始空间中患者之间的距离,而是计算采样集的主成分(PC)空间中患者间的距离。如上所述,我们将原始数据矩阵(采样集和传播集)投影到采样集将其与从采样集的RSVD获得的投影矩阵相乘。经过这种转换,患者之间的成对距离将在维数低得多的新空间中计算。
2.4使用多奥密克戎数据的亚型发现
当样本数较少时(默认情况下,当n个≤2000),我们使用集合策略来划分患者。该方法首先对每个数据类型进行聚类(使用第2.3节中描述的算法),并构造扰动连接性矩阵。然后,它将所有数据类型的连通性矩阵合并为一个单一的相似性矩阵,该相似性矩阵通过平均每对样本的连通性值来表示所有数据类型中患者之间的相似性。接下来,为了对相似矩阵进行聚类,它使用了几种基于相似性的算法,包括层次聚类、围绕medoids进行分区(36)、和动态树切割(37)然后选择最符合单个数据类型分区的分区。这种集成策略确保了所识别的子类型在所有数据类型中都是一致的,并且对聚类算法的选择具有鲁棒性。
当样本数量较大时(默认情况下,当n>2000时),我们执行类似于第2.3节中描述的算法的子采样和分类过程。这里的区别在于涉及多个数据类型。首先,我们随机选择2000个样本/患者,然后应用上述多组学算法对所选样本进行分区。我们将这组选定的2000名患者称为采样集剩下的病人传播集。下一个任务是确定传播集中患者的亚型。给定传播集中的患者,我们对每个数据类型执行FKNN程序,以使用从最近邻居处获得的标签来获得其属于每个子类型的概率。通过对所有数据类型的概率求平均值来计算最终概率。最后,我们将患者分为概率最高的亚型。当集成每个数据类型具有不同样本数的多组学数据时,也会应用此策略。这里的采样集是所有数据类型中都有数据的患者集(默认情况下最多2000名患者),其余患者将位于传播集中。
2.5 SMRT Web界面
该web应用程序可在http://SMRT.tinnguyen-lab.com.网站使用R Shiny框架构建(38). Shiny是一个R包,允许开发人员使用R编程语言直接构建交互式web界面。我们使用web界面将用户的数据和请求转发到新的SMRT方法,以执行数据集成和聚类。由于SMRT方法的效率,即使是具有数十万个样本的数据集,网站也可以在几分钟内返回结果。
使用web应用程序进行分析简单明了。用户可以使用左侧面板上的上传功能上传.csv文件或单个.rds文件中的表达式数据。每种数据类型都表示为一个矩阵,其中行表示样本,列表示基因/特征。SMRT可以自动确定子类型的数量。它不需要任何额外的配置或参数来执行分析。请参见补充第4节和图S6,第7部分以获取web应用程序的更详细描述。
3结果
为了评估SMRT的性能,我们使用39个癌症数据集和模拟数据进行了广泛的分析。首先,我们证明SMRT能够识别具有显著不同生存特征的癌症亚型。其次,我们对胶质瘤数据集进行了深入分析。最后,我们通过分析数十万个样本的模拟数据集来说明SMRT的可扩展性。我们还对SMRT和PAM50分类器在三个乳腺癌数据集(TCGA-BRCA、METABRIC_Discovery和METABRIC_Validation)中发现的亚型进行了比较分析补充第7节.
3.1使用39个癌症数据集的实验研究
在本文中,我们分析了37个TCGA和2个METABRIC数据集。对于TCGA数据集,我们从TCGA数据门户下载了匹配的mRNA、DNA甲基化和miRNA表达数据。对于METABRIC数据集,我们能够从欧洲基因组-表型档案中获得匹配的mRNA和拷贝数变化数据。我们还下载了每个患者的临床数据和生存信息,这些数据将用于评估分型方法的性能。补充表1,2提供数据集的更多详细信息。
我们将SMRT与八种最先进的子类型算法进行了比较:SNF(24)、CIMLR(32)、NEMO(28),moCluster(16)、iClusterBayes(9)、LRA群集(6)、MCCA(23)和IntNMF(20). 在我们的比较中使用了以下软件包:SNFtool v2.3.0 on CRAN for SNF,CIMLR v1.0.0 athttps://github.com/danro9685/CIMLR网站对于CIMLR,NEMO v0.1.0 athttps://github.com/Shamir-Lab/NEMO网站对于NEMO,moCluster的Bioconductor上的mogsa v1.16.0、iClusterPlus的Bioconductor v1.18.0和iCluster Bayes的LRACluster v1.18.00http://bioinfo.au.tsinghua.edu.cn/member/jgu/lracluster/对于LRACluster,MCCA的CRAN上的PMA v1.2.1,以及IntNMF的CRAN v1.2.0上的IntNMF。当维度数超过2000时,我们仅使用iClusterBayes、IntNMF和MCCA方差最大的前2000个变量,因为这些方法无法在全基因组尺度上分析数据。对于所有方法,我们都使用默认参数,并让所有方法自动确定最佳簇数。对于MCCA,它本身不是一种集群方法,我们遵循以下实现https://github.com/Shamir-Lab/Multi-Omics-Cancer-Benchmark(https://github.com/沙米尔实验室)用于聚类分析。
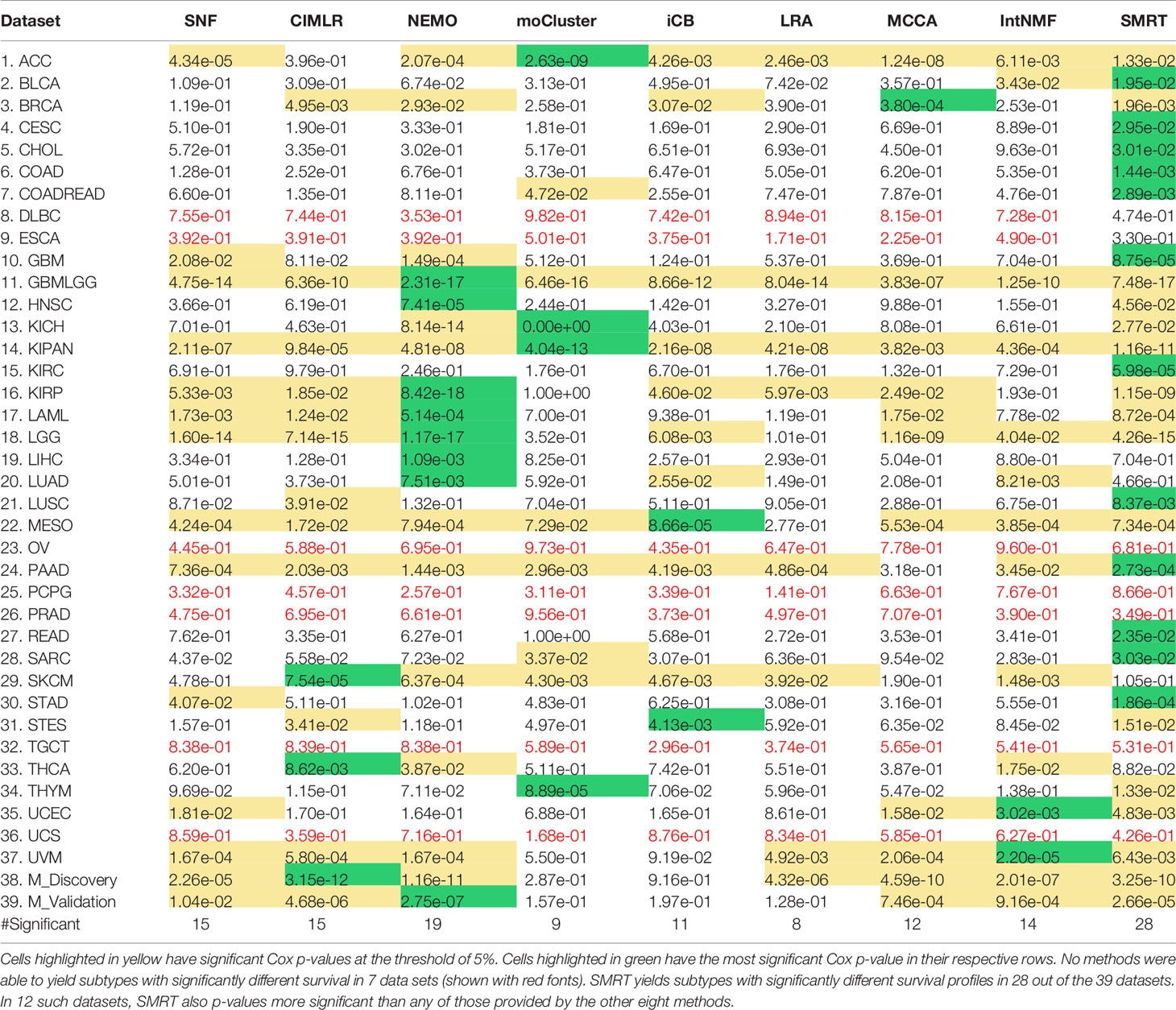
使用每种方法,我们在每个数据集中划分患者,然后使用Cox回归评估发现的患者组的生存差异(39). 总生存率数据用于TCGA数据集,无病生存率数据用于METABRIC数据集。表1显示了从每个数据集和方法获得的Cox p值(参见补充第9节,图S10–第17条每个数据集的Kaplan-Meier生存曲线)。在七个数据集中,没有一种方法能够识别Cox p值显著的亚型。对于剩余的32个数据集,SMRT在28个数据集中具有显著的p值,而NEMO在19个数据集中具有显著的p-值,所有其他方法在15个或更少的数据集中都具有显著的p-值。在这28个数据集中,SMRT在12个数据集中具有最显著的p值,而SNF、CIMLR、NEMO、moCluster、iClusterBayes、LRACluster、MCCA和IntNMF分别在0、3、8、4、2、0、1和2个数据集中具有最显著的p值。
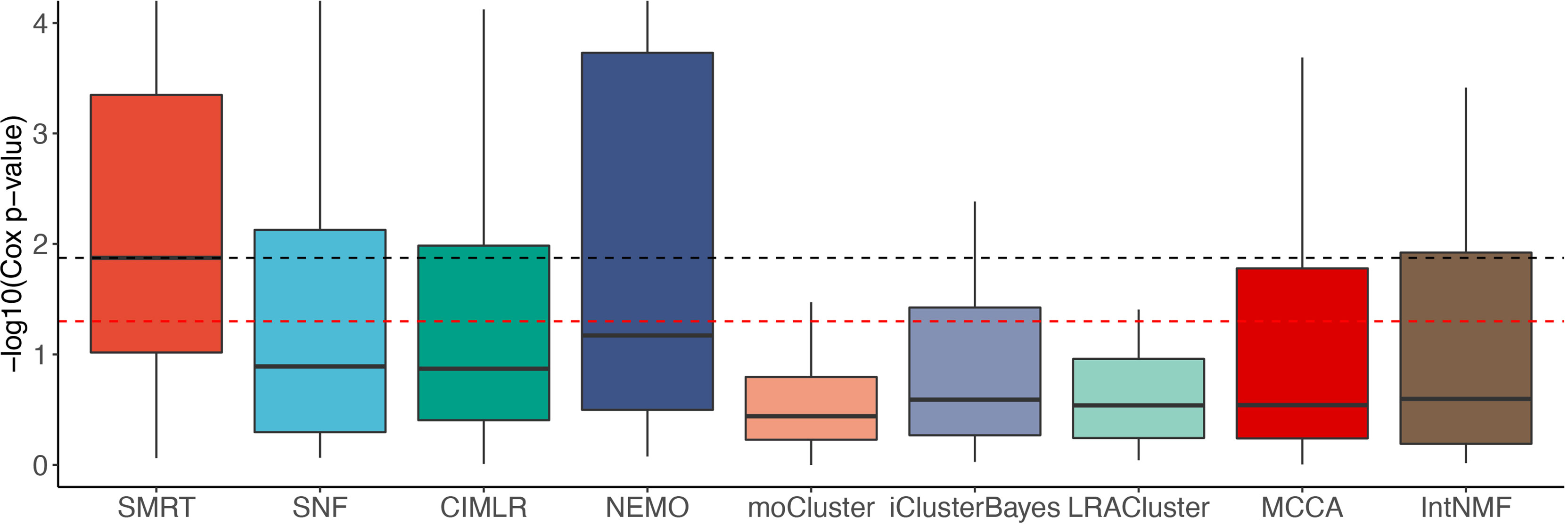
图2显示了对数10标度中Cox p值的分布。总的来说,SMRT的中位数-log10 p值接近2(即,中位数p值为0.01),而次优方法(NEMO)的中位数-log10 p值则接近1(即,中值p值为0.1)。Wilcoxon检验也证实SMRT的p值明显小于其他方法获得的p值(第页=0.0002,使用单尾Wilcoxon检验)。
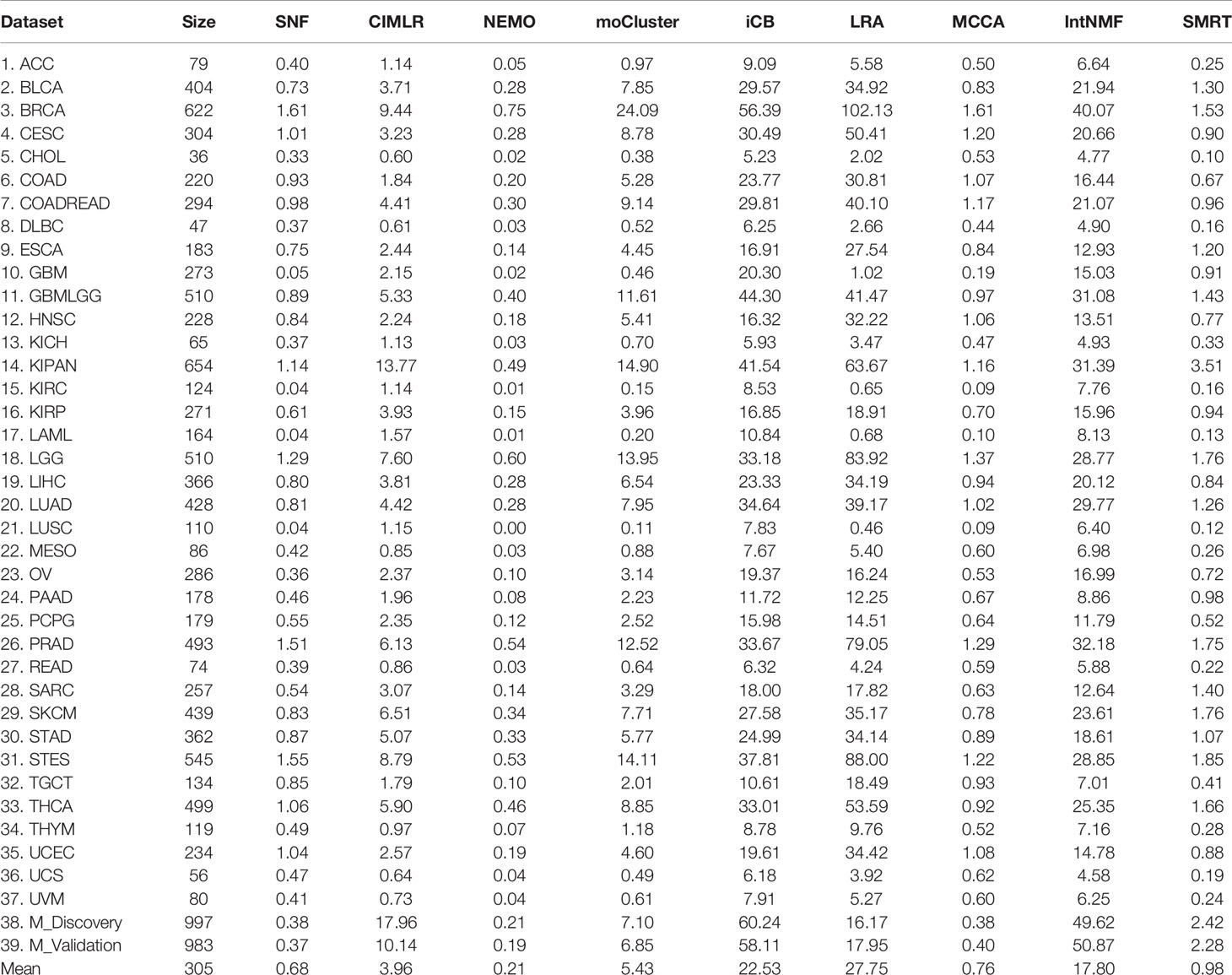
每种方法的运行时间如所示表2。前39行显示每个数据集中每个方法的运行时间,最后一行显示平均运行时间。平均而言,SMRT、SNF、NEMO和MCCA速度快,能够在一分钟内完成每个分析。其余的方法速度较慢,尤其是iClusterBayes和IntNMF,尽管它们的分析仅限于2000个最变异的基因。
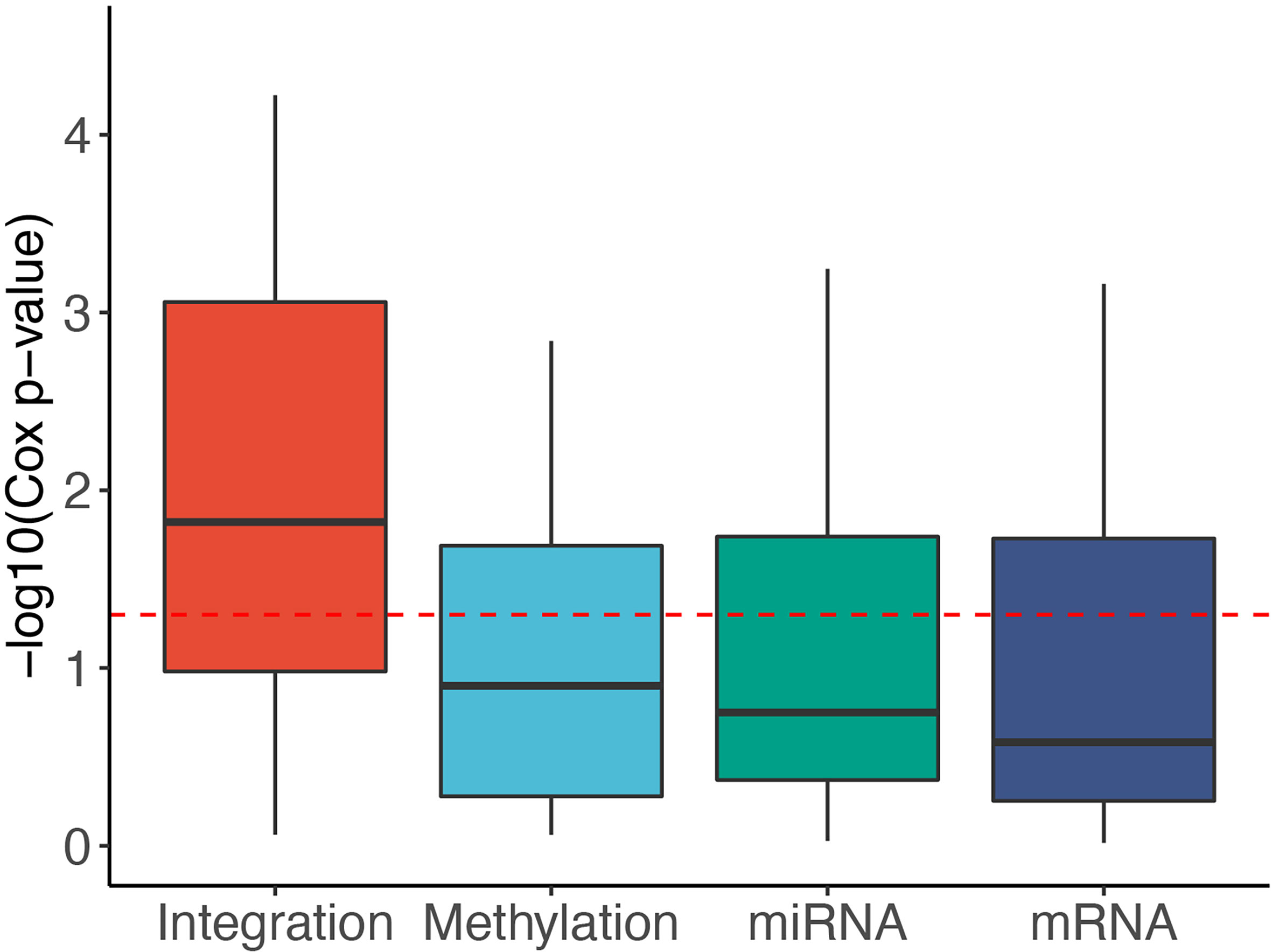
为了揭示每种数据类型的贡献,我们使用SMRT分别使用每种数据类别对患者进行分区。接下来,我们计算了从每种数据类型中获得的Cox p值,并将其与从多组学数据的分型中获得的Cox p值进行了比较。图3显示了37个TCGA数据集各数据类型的子类型-log10 p值的分布。从多组学数据中获得的p值比从单个数据类型中获得的值显著得多。从多组学数据中获得的中值p值接近0.01(-log10值接近2),而每种数据类型的中值p甚至高于0.1(-log 10值接近1)。这表明SMRT能够利用每种数据类型中的互补信息来确定具有显著生存差异的亚型。补充第10节和表S15提供有关每个数据集中各个数据类型的贡献的更多详细信息。
接下来,我们调查了发现的亚型与临床变量之间的关联。我们对性别、年龄、癌症分期和肿瘤分级进行了分析,这些数据至少可用于15个数据集。我们进行了以下分析:(1)Fisher精确检验,以评估性别(男性和女性)与发现的亚型之间的相关性的重要性;(2) 方差分析评估发现的亚型之间的年龄差异;最后(3)使用归一化互信息(NMI)计算发现的亚型与已知癌症分期和肿瘤分级之间的一致性。-的分布日志性别和年龄的10个p值如所示补充图S8(请参见补充表11-12精确的p值)。除NEMO和iClusterBayes外,聚类方法通常不会在聚类中产生性别或年龄差异。对于性别,iCluster Bayes在31个数据集中的17个数据集中具有显著的p值。对于年龄,NEMO和iClusterBayes在29个数据集中分别有17个和15个具有显著的p值。这一结果表明,在待发现的数据中存在有意义且与生存相关的分子特征,并且这些方法并不是简单地基于一些可见的临床变量(如性别或年龄)来分离患者。补充图S9和补充表13,14显示NMI值,该值表示发现的亚型与已知癌症分期和肿瘤分级之间的一致性。对于癌症阶段,SMRT和NEMO的NMI中值具有可比性,并且高于其他值。对于肿瘤分级,SMRT的NMI中位数最高。然而,对于癌症分期和肿瘤分级,所有方法的NMI值都很低,这意味着已知分期/分级与使用任何子分型方法发现的亚型之间的一致性很低。总之,从SMRT和其他分型方法中发现的亚型与性别、年龄、癌症分期和肿瘤分级等临床变量几乎没有一致性。
3.2 GBMLGG数据集的案例研究
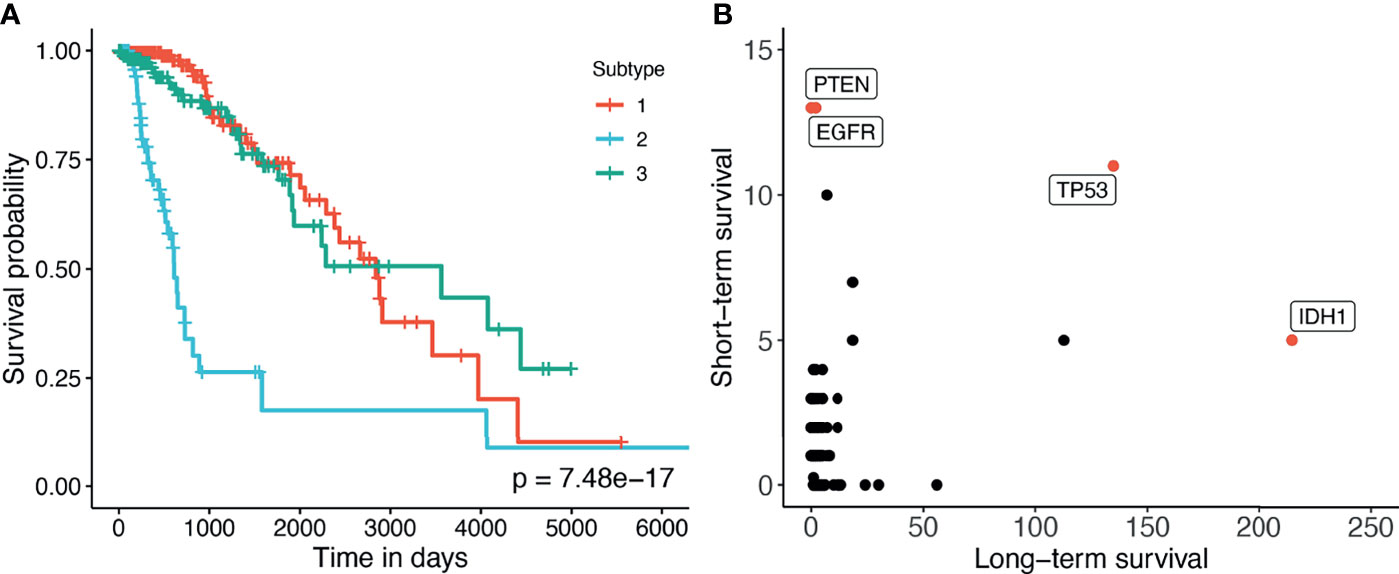
这里我们对GBMLGG(胶质瘤)进行了深入分析。图4A显示了所发现亚型的Kaplan–Meier生存分析。对于这个数据集,SMRT发现了三个亚型,其中一个亚型(第2组)的存活率非常低,在第3年,这一组患者的存活率仅为26%,而其他两个亚型的患者(第1组和第3组)的生存率为84%。我们还对数据集进行了变异分析,以发现短期存活患者组(第2组)中高度发生的突变,但长期存活患者组中(第1组和第3组)中不发生,反之亦然。图4B显示了每组的突变,其中每个点都是一个基因,其坐标表示相应组中具有该突变的患者数量。原则上,我们想调查图左上角或右下角的突变基因。在这个图中,我们可以很容易地识别出与GBMLGG疾病相关的四个标记基因:IDH1、TP53、PTEN和EGFR。其中,IDH突变(底部-右侧)被认为是导致低级胶质瘤(LGG)的因素,并已用于WHO分类系统(40)对预后较差的IDH突变型和IDH野生型进行分类。另一方面,EGFR不是LGG的常见突变,而是GBM(胶质母细胞瘤)的常见突变(41)存活率很低(42). EGFR扩增可引起PTEN基因突变(43)哪一个是肿瘤抑制基因(44). 有趣的是,长期存活组中没有患者出现PTEN突变。EGFR突变基因的出现可能是导致短期存活组患者存活率低的另一个原因。
我们使用共识路径分析平台上发现的亚型进一步进行路径分析(45)使用FGSEA方法(46)和KEGG路径数据库。补充图S4显示了显著的路径,显著性阈值为0.5%。在这个相连的网络中,每个节点都是一条通路,如果两条通路有共同的基因,那么它们之间就有一条边。如图所示,胶质瘤途径受到显著影响。其他与胶质瘤通路有共同成分的通路,包括MAPK信号通路、ErbB信号通路、钙信号通路和癌症通路,也受到显著影响。这证实了SMRT发现的亚型在胶质瘤和癌症相关通路的活性上存在显著差异。补充第2节和图S1–S4系列提供此数据集的更详细分析。
3.3子类型方法的可扩展性
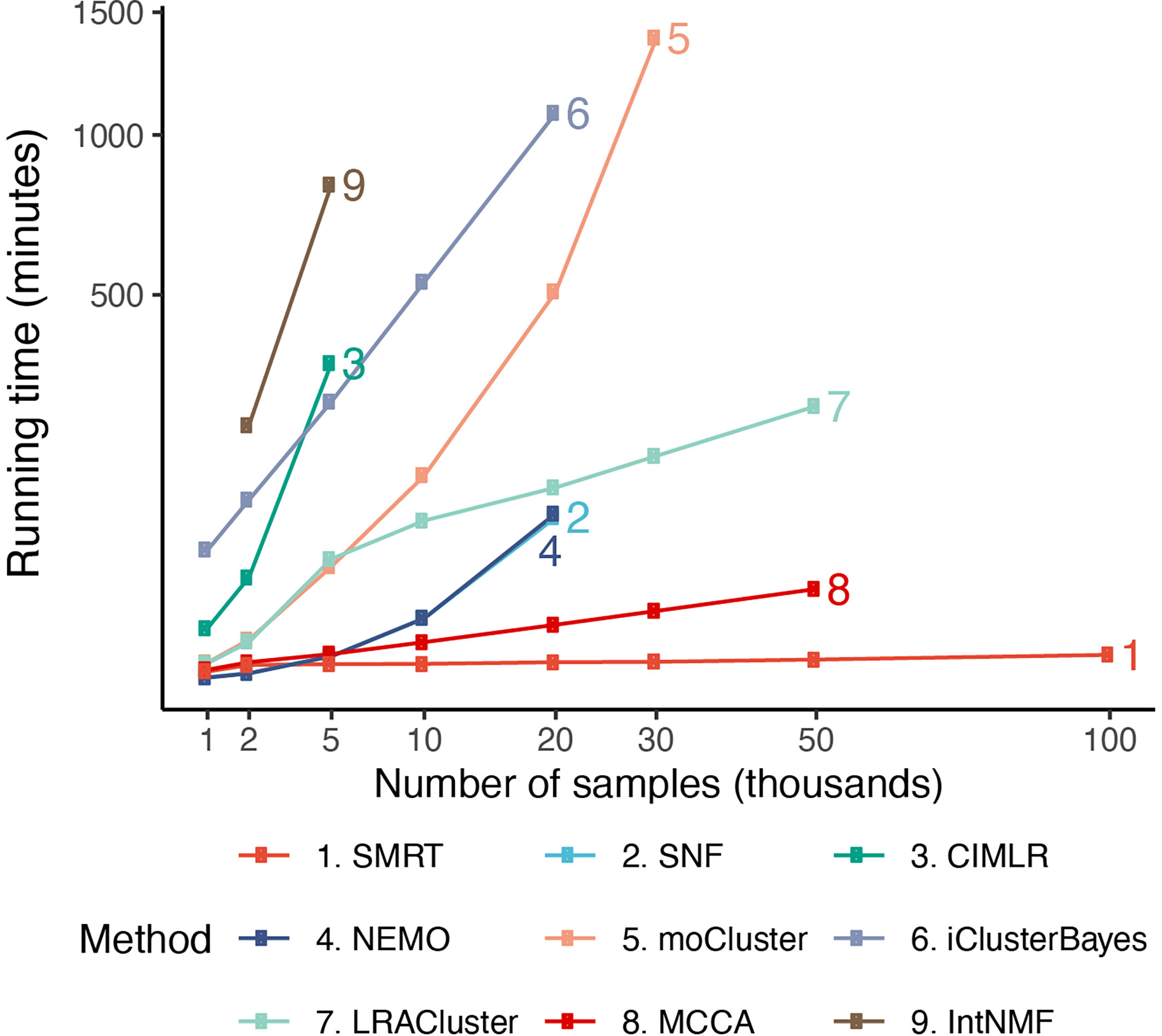
为了评估九种分型方法的可扩展性,我们生成了一些模拟数据集,其中基因/特征的固定数量为5000,样本数量不同(从1000到100000)。在生成的每个数据集中,有三类样本——每个样本都有一组不同的上调基因。使用了真实的类信息后部评估每种聚类方法的准确性。我们服务器的内存限制为376 GB。
图5显示了具有不同样本数的方法的运行时间。SNF、CIMLR、NEMO和moCluster的时间复杂性随样本大小呈指数级增加。这些方法无法分析样本数超过30000个的数据集(内存不足、产生错误或分析单个数据集的时间超过24小时)。MCCA和LRACluster能够分析50000个样本的数据集,但无法分析更大的数据集。只有SMRT能够分析所有大型数据集,包括100000个样本的数据集。SMRT比其他方法快得多,可以在三分钟内分析100000个样本的数据集。请参见补充第3节,图S5、和表4,5有关模拟和结果的详细信息。
4结论
在本文中,我们介绍了SMRT,这是一种快速而准确的数据集成和子类型发现方法。在使用39个癌症数据集进行的广泛分析中,我们发现SMRT在发现具有显著不同生存特征的新亚型方面优于其他最先进的方法。我们还证明了该方法可以在几分钟内以较低的内存需求准确地划分数十万个样本。同时,所提供的web应用程序对于缺乏计算背景或资源的生命科学家来说非常有用。尽管该软件是为癌症分型而开发的,但其他领域的研究人员可以使用web应用程序和R包进行无监督学习和数据集成。
数据可用性声明
本研究分析了公开可用的数据集。此数据可在此处找到:http://smrt.tinnguyen-lab.com/.
作者贡献
HN和TN构想并设计了该方法。HN、DT和BT在R中实现了该方法,并进行了数据分析和计算实验。MR、AC和SDa有助于数据准备和一些数据分析。HN、DT、SD和TN撰写了手稿。所有作者都审查并批准了手稿。
基金
这项工作得到了NIH NIGMS(拨款编号GM103440)和NSF(拨款编号2001385和2019609)的部分支持。
作者免责声明
本材料中表达的任何意见、发现、结论或建议均为作者的意见、发现和结论,并不一定反映任何资助机构的意见。
利益冲突
作者声明,该研究是在没有任何可能被解释为潜在利益冲突的商业或金融关系的情况下进行的。
出版商备注
本文中表达的所有声明仅为作者的声明,不一定代表其附属组织的声明,也不一定代表出版商、编辑和审稿人的声明。任何可能在本文中进行评估的产品,或制造商可能提出的索赔,都不受出版商的保证或认可。
补充材料
本文的补充材料可以在以下网站上找到:https://www.frontiersin.org/articles/10.3389/fonc.2021.725133/full#补充-材料
工具书类
2.Grossman RL、Heath AP、Ferretti V、Varmus HE、Lowy DR、Kibbe WA等,《癌症基因组数据共享愿景》。新英格兰医学杂志(2016) 375:1109–12. doi:10.1056/NEJMp1607591
PubMed摘要|CrossRef全文|谷歌学者
3.Curtis C、Shah SP、Chin S-F、Turashvili G、Rueda OM、Dunning MJ等。2000年乳腺肿瘤的基因组和转录组结构揭示了新的亚群。自然(2012) 486:346–52. doi:10.1038/nature10983
PubMed摘要|CrossRef全文|谷歌学者
4.Sudlow C、Gallacher J、Allen N、Beral V、Burton P、Danesh J等。《英国生物银行:一种用于确定中老年多种复杂疾病病因的开放获取资源》。公共科学图书馆-医学(2015)12:e1001779。doi:10.1371/journal.pmed.1001779
PubMed摘要|CrossRef全文|谷歌学者
5.Wilkerson MD,Hayes DN。ConsensusClusterPlus:一个具有置信度评估和物品跟踪功能的类别发现工具。生物信息学(2010) 26:1572–3. doi:10.1093/bioinformatics/btq170
PubMed摘要|CrossRef全文|谷歌学者
6.Wu D,Wang D,Zhang MQ,Gu J.使用低秩近似对多组数据进行快速降维和集成聚类:在癌症分子分类中的应用。BMC基因组学(2015) 16:1022. doi:10.1186/s12864-015-2223-8
PubMed摘要|CrossRef全文|谷歌学者
7.Speicher NK,Pfeifer N.通过正则化无监督多核学习整合不同数据类型,并应用于癌症亚型发现。生物信息学(2015)31:i268–75。doi:10.1093/生物信息系统/btv244
PubMed摘要|CrossRef全文|谷歌学者
8.Mo Q,Wang S,Seshan VE,Olshen AB,Schultz N,Sander C等。整合癌症基因组数据中的模式发现和癌症基因识别。国家科学院程序(2013) 110:4245–50. doi:10.1073/pnas.1208949110
PubMed摘要|CrossRef全文|谷歌学者
9.Mo Q,Shen R,Guo C,Vannucci M,Chan KS,Hilsenbeck SG.多类型Omics数据综合聚类分析的完全贝叶斯潜在变量模型。生物统计学(2018) 19:71–86. doi:10.1093/biostatistics/kxx017
PubMed摘要|CrossRef全文|谷歌学者
10.Coretto P,Serra A,Tagliaferri R.用于患者亚型的噪声高维基因表达数据的稳健聚类。生物信息学(2018) 34:4064–72. doi:10.1093/bioinformatics/bty502
PubMed摘要|CrossRef全文|谷歌学者
11.Ahmad A,Fröhlich H.通过基于生存率的贝叶斯聚类法对患者异质性进行临床更相关的分析。生物信息学(2017) 33:3558–66. doi:10.1093/bioinformatics/btx464
PubMed摘要|CrossRef全文|谷歌学者
13.柯克·P、格里芬·JE、萨维奇·RS、加拉马尼·Z、威尔德·DL。贝叶斯相关聚类集成多个数据集。生物信息学(2012) 28:3290–7. doi:10.1093/bioinformatics/bts595
PubMed摘要|CrossRef全文|谷歌学者
14.Lock EF、Hoadley KA、Marron JS、Nobel AB。多数据类型综合分析的联合和个体变异解释(Jive)。Ann应用统计(2013) 7:523. doi:10.1214/12-AOAS597
PubMed摘要|CrossRef全文|谷歌学者
15.Meng C、Kuster B、Culhane AC、Ghoma AM。多组数据集集成的多元方法。BMC生物信息(2014) 15:162. doi:10.1186/1471-2105-15-162
CrossRef全文|谷歌学者
16.Meng C、Helm D、Frejno M、Kuster B.Mocluster:识别多个Omics数据集的关节模式。蛋白组学研究杂志(2016) 15:755–65. doi:10.1021/acs.jproteome.5b00824
PubMed摘要|CrossRef全文|谷歌学者
17.李伟、张S、刘C-C、周晓杰。从多维基因组数据中识别多层基因调控模块。生物信息学(2012) 28:2458–66. doi:10.1093/bioinformatics/bts476
PubMed摘要|CrossRef全文|谷歌学者
18.于恩,高Y-L,刘J-X,尚J,朱R,戴L-Y。癌症基因组数据中基于图正则化多视图NMF的共差异基因选择和聚类。基因(2018) 9:586. doi:10.3390/genes9120586
CrossRef全文|谷歌学者
19.刘J,王C,高J,韩J.通过联合非负矩阵分解进行多视图聚类。在:2013年SIAM数据挖掘国际会议(SIAM)会议记录美国宾夕法尼亚州费城:工业和应用数学学会(2013)。第252-60页。
谷歌学者
20.Chalise P,Fridley BL.基于非负矩阵分解算法的多层次Omic数据集成聚类。公共图书馆(2017)12:e0176278。doi:10.1371/journal.pone.0176278
PubMed摘要|CrossRef全文|谷歌学者
21.Yang Z,Michailidis G.一种检测异构Omics多模态数据中模的非负矩阵分解方法。生物信息学(2016) 32:1–8. doi:10.1093/bioinformatics/btv544
PubMed摘要|CrossRef全文|谷歌学者
22.张S、刘C-C、李伟、沈H、莱尔德·普华永道、周晓杰。通过癌症基因组数据的综合分析发现多维模块。核酸研究(2012) 40:9379–91. doi:10.1093/nar/gks725
PubMed摘要|CrossRef全文|谷歌学者
23.Witten DM,Tibshirani RJ.稀疏典型相关分析的扩展及其在基因组数据中的应用。统计应用基因分子生物学(2009) 8:28. doi:10.2202/1544-6115.1470
CrossRef全文|谷歌学者
24.Wang B,Mezlini AM,Demir F,Fiume M,Tu Z,Brudno M,et al.在基因组尺度上聚合数据类型的相似网络融合。Nat方法(2014) 11:333–7. doi:10.1038/nmeth.2810
PubMed摘要|CrossRef全文|谷歌学者
25.Yuan Y,Savage RS,Markowetz F.患者特异性数据融合定义预后癌症亚型。公共科学图书馆计算生物学(2011)7:e1002227。doi:10.1371/journal.pcbi.1002227
PubMed摘要|CrossRef全文|谷歌学者
26.石Q,张C,彭M,于X,曾T,刘J,等.基于多异质Omics数据自适应对齐的模式融合分析。生物信息学(2017) 33:2706–14. doi:10.1093/bioinformatics/btx176
PubMed摘要|CrossRef全文|谷歌学者
27.Hoo Z,Tseng G.在疾病亚型发现的基因组应用中具有重叠群套索的整合稀疏K-Means。Ann应用统计(2017) 11:1011. doi:10.1214/17-AOAS1033
PubMed摘要|CrossRef全文|谷歌学者
28.Rappoport N,Shamir R.NEMO:通过整合部分多组数据进行癌症分型。生物信息学(2019) 35:3348–56. doi:10.1093/bioinformatics/btz058
PubMed摘要|CrossRef全文|谷歌学者
29.Nguyen T,Tagett R,Diaz D,Draghici S.数据整合和疾病分型的新方法。基因组研究(2017) 27:2025–39. doi:10.1101/gr.215129.116
PubMed摘要|CrossRef全文|谷歌学者
30.Nguyen H、Shrestha S、Draghici S、Nguyem T.PINSPlus:综合基因组数据中肿瘤亚型发现的工具。生物信息学(2019) 35:2843–6. doi:10.1093/bioinformatics/bty1049
PubMed摘要|CrossRef全文|谷歌学者
31.Tran D,Nguyen H,Le U,Bebis G,Luu HN,Ngueen T。使用一致性因子分析进行癌症分型和风险预测的新方法。前Oncol(2020) 10:1052. doi:10.3389/电话:2020.01052
PubMed摘要|CrossRef全文|谷歌学者
32.Ramazzotti D,Lal A,Wang B,Batzoglou S,Sidow A.多器官肿瘤数据揭示了与生存相关的分子机制的多样性。国家公社(2018) 9:4453. doi:10.1038/s41467-018-06921-8
PubMed摘要|CrossRef全文|谷歌学者
33.Golub G,Kahan W.计算矩阵的奇异值和伪逆。工业应用数学期刊B辑:数值分析(1965) 2:205–24. doi:10.1137/0702016
CrossRef全文|谷歌学者
34.Beygelzimer A、Kakadet S、Langford J、Arya S、Mount D、Li S。FNN:快速最近邻搜索算法和应用。R包1.1.3版《奥地利维也纳:综合R档案网络》(2019年)。
谷歌学者
35.雷普利银行。现代应用统计学美国纽约州纽约市:施普林格出版社(2002年)。
谷歌学者
36.考夫曼L,卢梭P.通过Medoids聚类。图片来源:Dodge Y,编辑。基于L1-范数及相关方法的统计数据分析阿姆斯特丹:北荷兰(1987)。第405-16页。
谷歌学者
37.Langfelder P,Zhang B,Horvath S.从层次聚类树定义聚类:R的动态树切割包。生物信息学(2008) 24:719–20. doi:10.1093/bioinformatics/btm563
PubMed摘要|CrossRef全文|谷歌学者
38.Chang W、Cheng J、Allaire J、Xie Y、McPherson J。闪亮:R.R包1.4.0.2版的Web应用程序框架《奥地利维也纳:综合R档案网络》(2020年)。
谷歌学者
39.Therneau TM,Grambsch PM。生存数据建模:扩展考克斯模型美国纽约州纽约市:施普林格出版社(2000年)。
谷歌学者
40.Louis DN、Perry A、Reifenberger G、Von Deimling A、Figarella Branger D、Cavenee WK等。2016年世界卫生组织中枢神经系统肿瘤分类:总结。神经病理学学报(2016) 131:803–20. doi:10.1007/s00401-016-1545-1
PubMed摘要|CrossRef全文|谷歌学者
41.郝Z,郭D.Egfr突变:与低度胶质瘤免疫浸润相关的新预后因子;探索性研究。BMC癌症(2019) 19:1–13. doi:10.1186/s12885-019-6384-8
PubMed摘要|CrossRef全文|谷歌学者
42.Stupp R、Hegi ME、Mason WP、van den Bent MJ、Taphoorn MJ、Janzer RC等。联合和佐剂替莫唑胺放射治疗的效果与美国相比胶质母细胞瘤放疗对生存率的影响:一项随机Ⅱ期研究:Eortc-Ncic试验的5年分析。柳叶刀Oncol(2009) 10:459–66. doi:10.1016/S1470-2045(09)70025-7
PubMed摘要|CrossRef全文|谷歌学者
43.Ohgaki H,Kleihues P.原发性和继发性胶质母细胞瘤的遗传途径。美国病理学杂志(2007) 170:1445–53. doi:10.2353/ajpath.2007.070011
PubMed摘要|CrossRef全文|谷歌学者
44.Ali IU,Schriml LM,Dean M.Pten/Mmac1基因的突变谱:一种具有脂肪酶活性的肿瘤抑制剂。美国国家癌症研究所(1999) 91:1922–32. doi:10.1093/jnci/91.22.1922
CrossRef全文|谷歌学者
45.Nguyen H、Tran D、Galazka JM、Costes SV、Beheshti A、Draghici S等。CPA:基于网络的共识路径分析和交互式可视化平台。核酸研究(2021)49:gkab421。doi:10.1093/nar/gkab421
CrossRef全文|谷歌学者
46.Korotkevich G、Sukhov V、Budin N、Shpak B、Artyomov MN、Sergushichev A.快速基因集富集分析。生物Rxiv(2021), 060012. 数字对象标识代码:10.1101/060012
CrossRef全文|谷歌学者