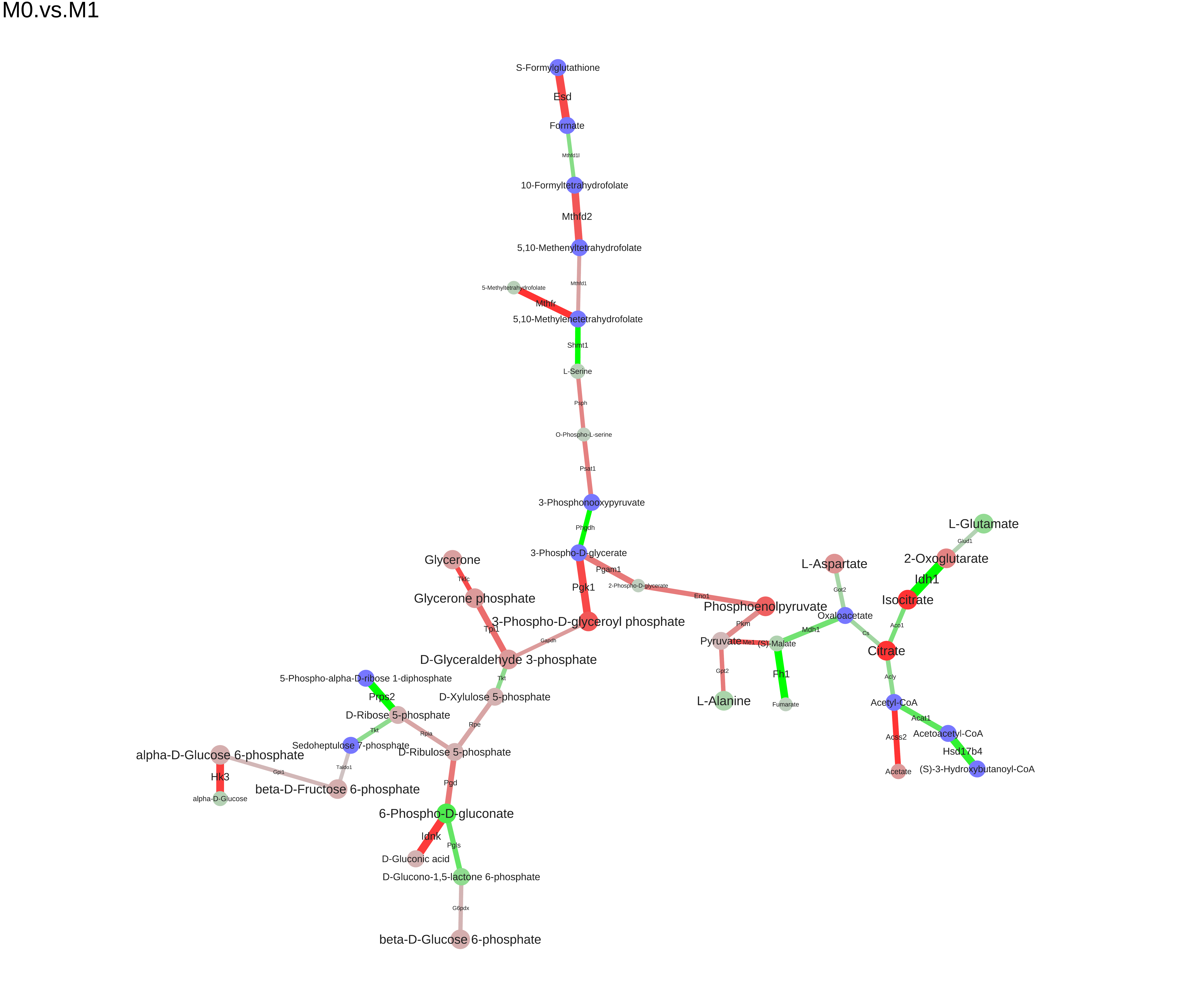
[](https://github.com/ctlab/gatom/actions/workflows/R-CMD-check.yaml)#关贸总协定寻找原子跃迁中活性代谢模块的R包网络。可以找到完整的渐晕图[此处](https://rpubs.com/asergushichev/gatom-tutorial网站).###安装```第页库(devtools)安装github(“ctlab/gatom”)```###快速入门```第页图书馆(gatom)库(data.table)库(igraph)图书馆(mwcsr)```首先,让我们用原子映射(`network`对象)、酶加载数据小鼠注释(`org.Mm.eg.gatom`)和代谢物注释(`met.kegg.db.rda`):```对数据(“networkEx”)数据(“org.Mm.eg.gatom.annoEx”)数据(“met.kegg.dbEx”)```加载输入数据:```第页数据(“met.de.rawEx”)数据(“gene.de.rawEx”)```获取原子图:```第页g<-makeMetabolicGraph(网络=网络Ex,拓扑=“atoms”,org.gatom.anno=org.Mm.eg.gatom.annoEx,gene.de=基因.de.rawEx,met.db=met.kegg.dbEx,met.de=met.de.rawEx)```##找到具有RefSeq ID的基因的DE表##找到HMDB ID代谢物的DE表```第页打印(g)```##IGRAPH b23a8ad联合国——176 190——##+属性:名称(v/c)、代谢物(v/c##|(v/c),pval(v/n),原点##|(v/n)、logPval(v/n)、信号(v/c)、标签(e/c),pval(e/n),来源##|(e/n),RefSeq(e/c),基因,##|reaction_equation(e/c),url(e/c),reaction(e/c),log2FC(e/n),##|baseMean(e/n),logPval##+来自b23a8ad的边(顶点名称):##[1]C00025_-0.3248_2.8125-C00026_-0.3248 _2.8125##[2]C00025_-1.6238_3.5625-C00026_-1.628_3.5625##[3]C00025_-2.9228_2.8125-C00026_-2.9225_2.8125## + ... 省略了几个边得分图,获得SGMWCS(信号广义最大权重子图)问题实例:```第页gs<-评分图(g,k.gene=25,k.met=25)```初始化SMGWCS解算器(启发式松弛与剪切解算器`rnc_solver `用于简化,请查看`mwcsr`包更多选项的文档):```第页解算器<-rnc_solver()```查找模块:```第页res<-solve_mwcsp(解算器,gs)m<-res$图形``````第页打印(m)```##IGRAPH e74bc24联合国——37 36——##+属性:信号(g/n)、名称(v/c)、代谢物,##|标签(v/c)、url(v.c)、pval(v/n##|(v/n),baseMean(v/n.),logPval(v/n:),signal(v/c),score(v/n-),##|标签(e/c),pval(e/n,##|酶(e/c),反应_名称(e/c),反作用_等式(e&c),url##|(e/c),反应(e/c),log2FC(e/n),基本平均值(e/n),logPval(e/m),##|signal(e/c),signalRank(e/n),score(e/n)##+来自e74bc24的边(顶点名称):##[1]C00025_-2.9228_2.8125-C00026_-2.9225_2.8125##[2]C00024_15.0644_27.8518-C00033_-1.6238_0.5625## + ... 省略了几个边```第页头部(E(m)$标签)```##[1]“Psat1”“Acss2”“Gpt2”“Got2”“Pkm”“Tpi1”```第页头(V(m)$标签)```##[1]“丙酮酸”“乙酰-CoA”“L-谷氨酸”“2-氧化戊二酸”##[5]“醋酸盐”“草酸酯”我们可以将模块保存为不同的格式(dot、xgmml、svg、pdf):```第页saveModuleToPdf(m,file=“M0.vs.M1.pdf”,name=“M0.vs.M1”,n_iter=100,force=1e-5)```