介绍
公共化学(https://pubchem.ncbi.nlm.nih.gov网站) (Kim等人,2016a;Kim等人,2016年b;Kim等人,2019年;Kim等人,2021年)是美国国家卫生研究院化学品及其生物活性的公共储存库。与其他NCBI数据库一起(Sayers等人,2019年;Sayers等人,2021年)PubChem为生物医学发现提供了广泛的资源。PubChem每月都有数百万用户访问,为广泛的用户提供服务,包括研究科学家、专利代理人、化学卫生官员、化学教育工作者、学生和许多其他人。PubChem数据量的巨大增长,以及其日益增加的异质性和质量可变性,需要一种新的探索性方法来快速检索相关、非冗余和可靠的信息,并以易于理解的形式呈现,围绕生物医学社区最有用的内容进行组织。
PubChem用户经常想发现和探索化学物质、基因、蛋白质和疾病之间的重要关系,同行评审的期刊文章证明了这一点。考虑到PubChem中包含的数据的大小和范围,这项任务并不容易。为了满足这一需求,PubChem开发并实施了文献知识小组。对于给定的实体(即化学品、基因或蛋白质),文献知识面板显示了几个最相关的“邻居”,即与实体一起提及的化学品、基因、蛋白质或疾病。这些小组还提供了PubMed记录的样本,其中提到了该实体及其邻国。需要从公共数据库中提取关于这些实体之间关系的信息,然后断言和汇总。如此复杂的相互关联实体集合通常称为知识图(Singhal,2012年;Ehrlinger和Wöß,2016年;苏利文,2020年;谷歌,2021年;SciBite,2021年). 为了使集合有用,需要查找、组织和向用户显示与查询相关的数据片段。在本文中,我们描述了通过分析生物医学文献中术语的共现现象来揭示和总结化学品、基因、蛋白质和疾病之间重要关系的方法。
为文献知识面板生成数据的第一步是识别非结构化文本中的相关命名实体。虽然可用的数据包括可信的精选集、不同储户提供的实验数据,以及由索引器手动注释的文献和生物医学出版物(MEDLINE,2021年); 使用命名实体识别软件可以从非结构化文本中提取大量数据(Ratinov,2009年). 当前命名实体识别方法包括字典匹配、使用规则识别专门术语以及使用统计和神经语言模型进行上下文分析(Sayle等人,2011年;Vazquez等人,2011年;Jessop等人,2012年;Rocktäschel等人,2012年;Gurulingappa等人,2013年;Lowe和Sayle,2015年;Pletscher-Frankild等人,2015年;Song等人,2018年;Devlin等人,2019;Lee等人,2020年;田等人,2020年). 为了为PubChem文献知识小组生成数据,使用第三方命名实体识别软件LeadMine在PubMed记录中注释实体(Lowe和Sayle,2015年),并与PubChem Compound数据库中的化学同义词以及基因、蛋白质和疾病名称相匹配,如材料和方法.
通过统计分析和基于相关性的抽样确定最相关的信息,并以紧凑的形式进行汇总。对于每个查询实体,知识图中显示了几个最相关的邻居,以及每个查询-邻居对的几个最相关PubMed记录,其中,查询是为其构建面板的实体(即,化合物、基因或蛋白质),而相邻的是共生实体(即化合物、基因、蛋白质或疾病)。每个记录样本附带的附加信息以及下载链接有助于用户检查已确定关系的上下文及其可靠性。中列出了文献知识小组示例的链接表1,屏幕截图如所示图1——三.
使用排除规则对PubChem化合物进行冗余和近冗余消除。我们讨论了三种不同的对共现进行评分的方法:最简单的是基于两个实体同时被提及的记录数量的评分,另外两种是更高级的基于信息的评分,允许校正数据库中的大量邻居。为了让用户具有一定的灵活性,同时确保PubChem的效率,我们允许用户从有限的选项中进行选择,并为每个选项预先计算数据。
我们的方法、数据源和实现的详细信息在材料和方法中讨论了该方法在实际数据中的应用结果中讨论了当前方法的局限性以及增强和扩展使用来自可信的人工管理源以及各种异构数据源的信息的机会讨论.
材料和方法
NCBI PubMed记录(PubMed,2021年;Sayers等人,2021年)使用LeadMine(实体识别软件程序)下载并注释,该程序使用字典匹配和专用化学术语(“语法”)规则来识别相关文本实体(Lowe和Sayle,2015年)3.15版。使用LeadMine提供的词典以及我们的内部词典,可以在多个类别(如化学品、基因、蛋白质和疾病)中执行注释。虽然允许嵌套注释(例如在蛋白质名称中找到的化学名称,如“水杨酸”,如“水杨酸结合蛋白2”),但只有非嵌套注释用于构建知识面板。嵌套在其他带注释实体中的带注释实体将保留以供内部使用,例如质量控制和消除歧义。
PubMed记录中识别的实体被规范化并与PubChem Compound数据库同义词匹配。类似地,通过下面描述的过程,基因名称和蛋白质名称分别与相应的PubChem gene和PubChem-protein页面匹配。疾病实体与医学主题标题(MeSH)标题和补充概念相匹配(MeSH,2021年)使用LeadMine。对于每个查询实体(化合物、基因或蛋白质),将根据查询和相邻实体之间的共现分数选择几个非冗余相邻实体(化合物,疾病,基因或蛋白)。分数取决于PubMed记录的计数,同时提及查询邻居对。根据相关性得分对共同提及它们的PubMed记录进行抽样,相关性得分反映了共同提及的实体在PubMed记录中的位置和频率,以及PubMet记录的特征(例如,文章类型和出版日期最近度)。下面将详细讨论匹配算法和评分方案。
文本实体匹配
使用LeadMine提供的字典和解析器将疾病文本实体与MeSH标题和补充概念匹配(Lowe等人,2016),并进行了一些更正,以适应最近MeSH的变化。使用下面段落中描述的匹配算法,将其他注释实体与化学品、基因和蛋白质的名称进行匹配。
虽然使用LeadMine的区分大小写字典注释的实体是以区分大小写的方式匹配的,但在使用区分大小写词典匹配LeadMine注释的实体时,会考虑大写。执行了一个规范化步骤,其中所有括号都变成了圆括号。此外,在匹配之前,尽可能使用Open Parser for Systematic IUPAC Nomenclature(OPSIN)项目的功能将文本实体和数据库条目转换为ASCII(从UTF-8或Unicode字符集)(Lowe等人,2011年).
如果实体具有相同的字母数字字符串,则认为它们是匹配的(对于由使用区分大小写字典注释的实体生成的字母数字串,也使用区分大小大小写的匹配,否则使用区分大小码的匹配),如果对齐分数较高,则允许在非字母数字符号中使用一些灵活性。使用Needleman–Wunsch算法对齐一对具有相同字母数字字符串的文本实体(Needleman和Wunsch,1970年)权重:1表示精确匹配,-1表示不匹配和间隙。对于要接受的两个对齐实体,通过实体最大长度标准化的匹配字符数应大于或等于接受阈值0.9(对于化合物)和0.7(对于基因和蛋白质)。这些阈值是在对各种案例进行实验后根据经验确定的。
目前,尚不可能将非结构化文本(例如PubMed记录)中的基因或蛋白质实体与生物体信息进行可靠连接。在注释基因和蛋白质实体时,LeadMine经常将它们的名称解析为模糊生物体中的名称(例如,人类基因和蛋白质的旧名称解析为其他物种的当前名称)。我们决定优先考虑人类基因和蛋白质。已实施以下策略,以将基因和蛋白质文本实体解析为最合理的基因、蛋白质或酶符号(如果可能,对应于人类):
-尝试在人类基因组组织(HUGO)基因命名委员会(HGNC)名称之间找到匹配项(Braschi等人,2019年;HUGO,2021年);
-试着在IUPHAR/BPS药理学指南中找到名字的匹配(Armstrong等人,2020年;IUPHAR/BPS,2021年);
-尝试在UniProt中查找名称之间的匹配项(贝特曼等人,2017年);
-否则,尝试匹配酶名称并解析为EC编号(拜罗赫,2000;2021年Expassy).
一般来说,要区分基因的名称和该基因编码的蛋白质的名称是非常困难的,而且通常是不可能的。因此,基因和蛋白质的名称并没有严格区分,而是被视为一个类别。因此,本研究中考虑的注释可分为三类:化学品、基因/蛋白质和疾病。
PubMed记录中一对实体的相关性得分
PubMed记录中共同提到的两个实体的相关度得分用于对PubMed记录进行采样,这些记录可能会提供关于它们之间关系的上下文。相关性评分方案经过精心设计,以反映以下因素:
-标题中出现的查询邻居对显著增加了出版物的相关性;
-文本中靠近的带注释实体更有可能关联(Manning和Schütze,1999年);
-在所有其他因素都相同的情况下,最近的出版物可能比旧出版物更重要;
-与“综述”文章相对应的PubMed记录略微增加了出版物的相关性。
相关性得分对于匹配的实体和PubMed记录中由以下经验公式计算
哪里,
如果匹配的实体为1在记录的标题中,否则为0(定义类似);
如果匹配的实体为1记录中存在多次,否则为0(定义类似);
如果两个实体都是2和出现在摘要中的两个或多个句子中,如果只有一个句子,则为1,否则为0;
如果记录为1标记为与评论文章对应,否则为0;
等于:
出版年限在一年以内的;
如果出版年龄在一年至两年之间;
如果出版年龄在2至5年之间;
如果出版年龄在5至10年之间;
如果出版年龄在10至15年之间;
如果出版年龄在15至20年之间;
如果出版年龄超过20年。
使用的重量等式1是经过仔细考虑后选定的。PubChem生产中当前使用的值为:;;;;;;;;.
等式1通过研究各种具有代表性的PubMed记录并确定影响因素的相对重要性,确定了权重值。由于实体关系解释的主观性和策划数据的相对稀缺性,很难客观地检查公式和权重的准确性和可靠性。尽管公式是启发式的,权重值是主观的,但它们可以进一步优化以处理特定的用例。
选择出版日期的时间段
虽然协调不同的相关性因素是一个复杂的问题,但平衡出版日期与其他相关性因素可能特别困难,并且在很大程度上取决于用户的需求。在默认设置下使用预先计算的数据进行操作时,用户可以从有限的选项中选择首选的发布时间段(目前有三个选项:自去年以来、过去5年内或过去10年内)。根据用户的选择,在web浏览器中根据预先计算的数据形成页面视图。这种方法在确保系统效率的同时允许一些灵活性。
对共同事件进行评分
两个实体之间的共现分数用于为给定实体选择最常见的实体。已经测试了三种方法来为共现分数开发合适的公式。
考虑查询实体和相邻实体,属于类别我和二,并让和是一组PubChem记录,从类别中提到我和二分别为和.让和是一组提及实体的PubChem记录和,、和.表示是一组PubChem记录,其中实体和是共同提到的(很容易看出以及)。
对于共现分数,我们考虑了以下选项.首先,我们使用
哪里.这种简单的评分方案适用于在数据集中相对较少出现然而,当邻居经常出现在PubMed的文章中(例如,化学名称“water”或疾病术语“cancer”),这种方案往往使其位于相邻实体列表的顶部,即使和不是很具体。这可以通过切换到由公式计算的基于信息增益的共现分数来避免
哪里是数据集的大小,并且是内的记录数where实体被提及。分数(3)来自Kullback–Leibler散度,也称为相对熵(库尔贝克和莱布勒,1951年;Manning等人,2008年). 它可以被视为术语频率的变体–反向文档频率(TF-IDF)分数(Aizawa,2003年;罗伯逊,2004年;Manning等人,2008年;Rajaraman和Ullman,2011年). 在撰写本文时,等式3PubChem共现显示屏使用的是什么。
为了定义更高级的评分公式,让我们表示实体的相关性得分和在记录中作为,
和
然后,共现分数由以下公式定义
而共现分数由定义方程式2,三仅取决于物品数量,分数由等式4也取决于相关性得分的分布。有关这些评分方案的进一步解释,请参阅结果.
冗余消除
PubChem中的一些化合物非常相似(例如,同一母体化合物的不同盐形式),如果这些化合物碰巧与知识图中的查询化合物相邻,面板将被冗余信息堵塞,从而降低其效用。通过从每个具有相同父连接组的邻居“组”中选择一个具有代表性的邻居,可以消除这种冗余(Fu等人,2015年)或者(更选择性地)相同的化学名称。使用以下规则来选择具有代表性的邻居:
-不考虑与查询复合词属于同一父连接性组或名称相同的所有复合词;
-当PubChem化合物作为相邻化合物添加到知识面板时,重复应用相同的规则。在每次迭代中,共现得分最高的化合物(基于等式3)从候选列表中选择(在同现分数值相同的情况下,选择是任意的)。所选化合物将作为代表性相邻化合物添加到知识面板中,而与该化合物属于同一亲子连接性组或与其同名的所有化合物都将从列表中删除(仅考虑PubChem同义词列表中与PubMed记录匹配的名称)。
这些规则的应用导致查询实体的“非冗余”邻居列表。请注意,这些规则确保邻域之间以及查询之间不太相似。
实施
预计算的共现数据加载到一组数据库中,并提供给PubChem Compound、Gene和Protein页面相应文献部分中的知识小组。创建给定PubChem记录的摘要页面时,将查询这些数据库,以查看是否存在有关特定记录的共现信息。如果是这样,则在摘要中添加适当的标题(在目录的文献部分)。当用户滚动到摘要页面的该部分时,将查询数据库以收集面板中显示的信息。
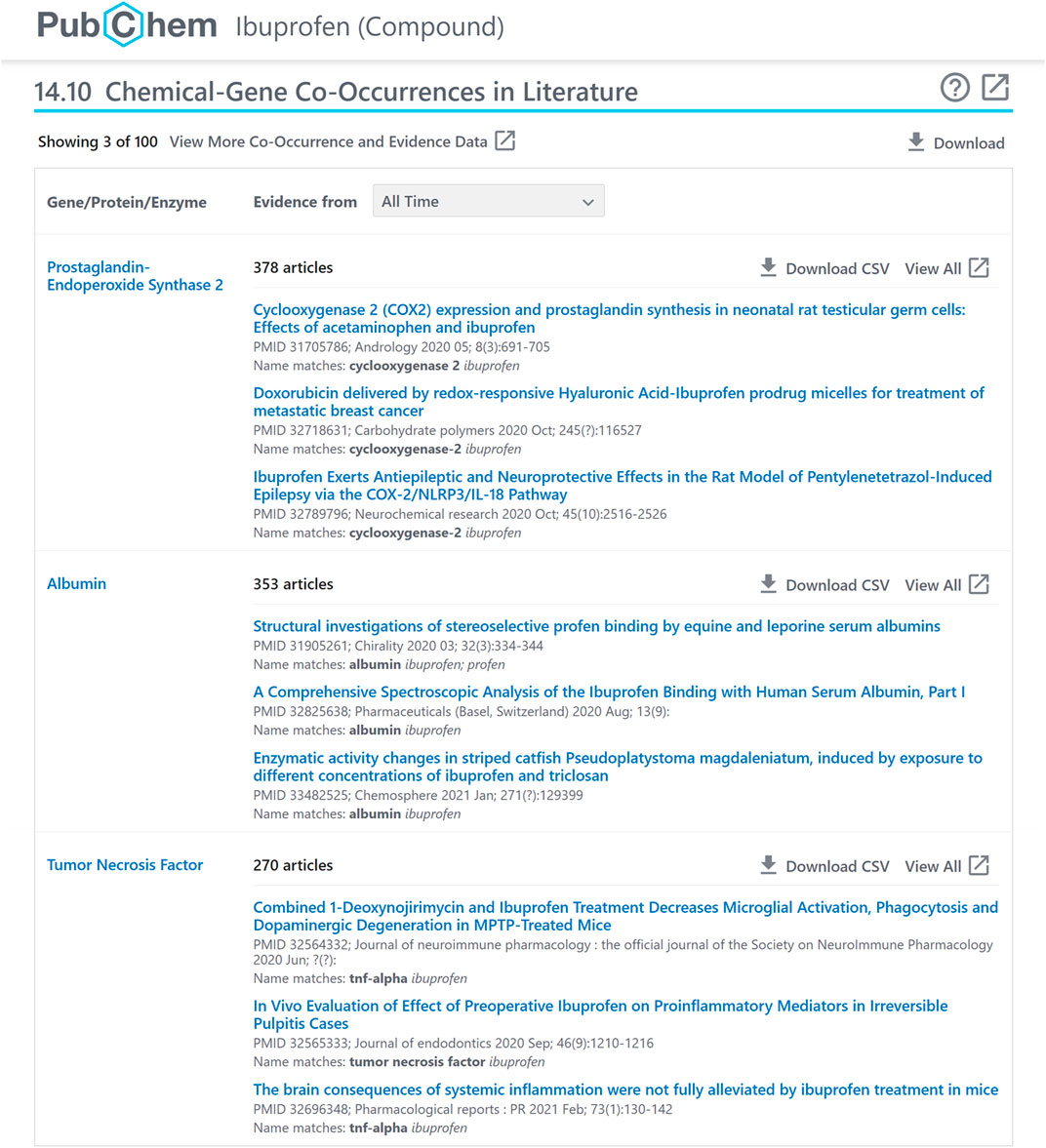
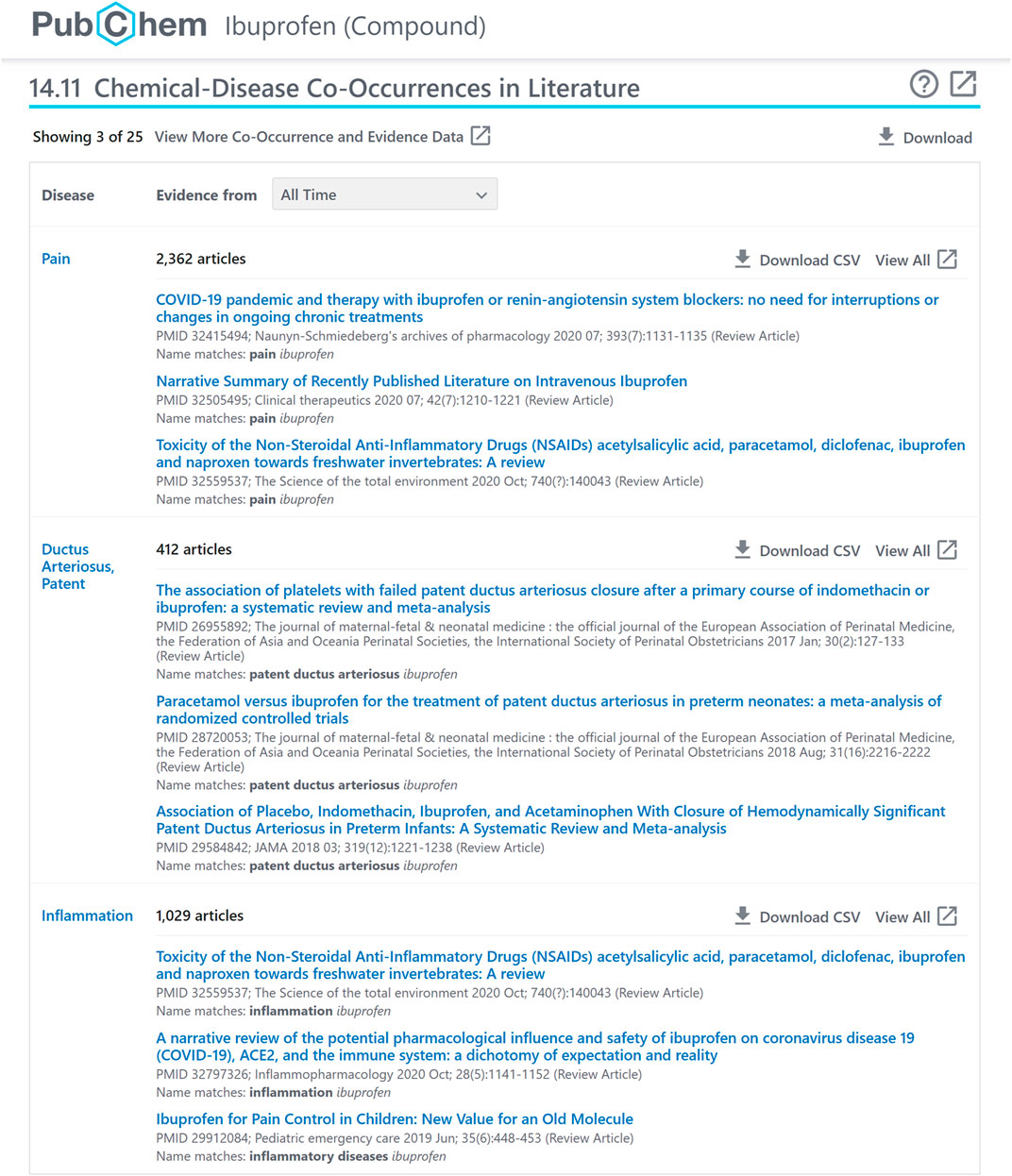
与许多其他基于NLP的工具一样,我们不区分蛋白质名称和编码基因的名称,因为它们经常互换使用。六类文献共现面板的示例如所示表1.
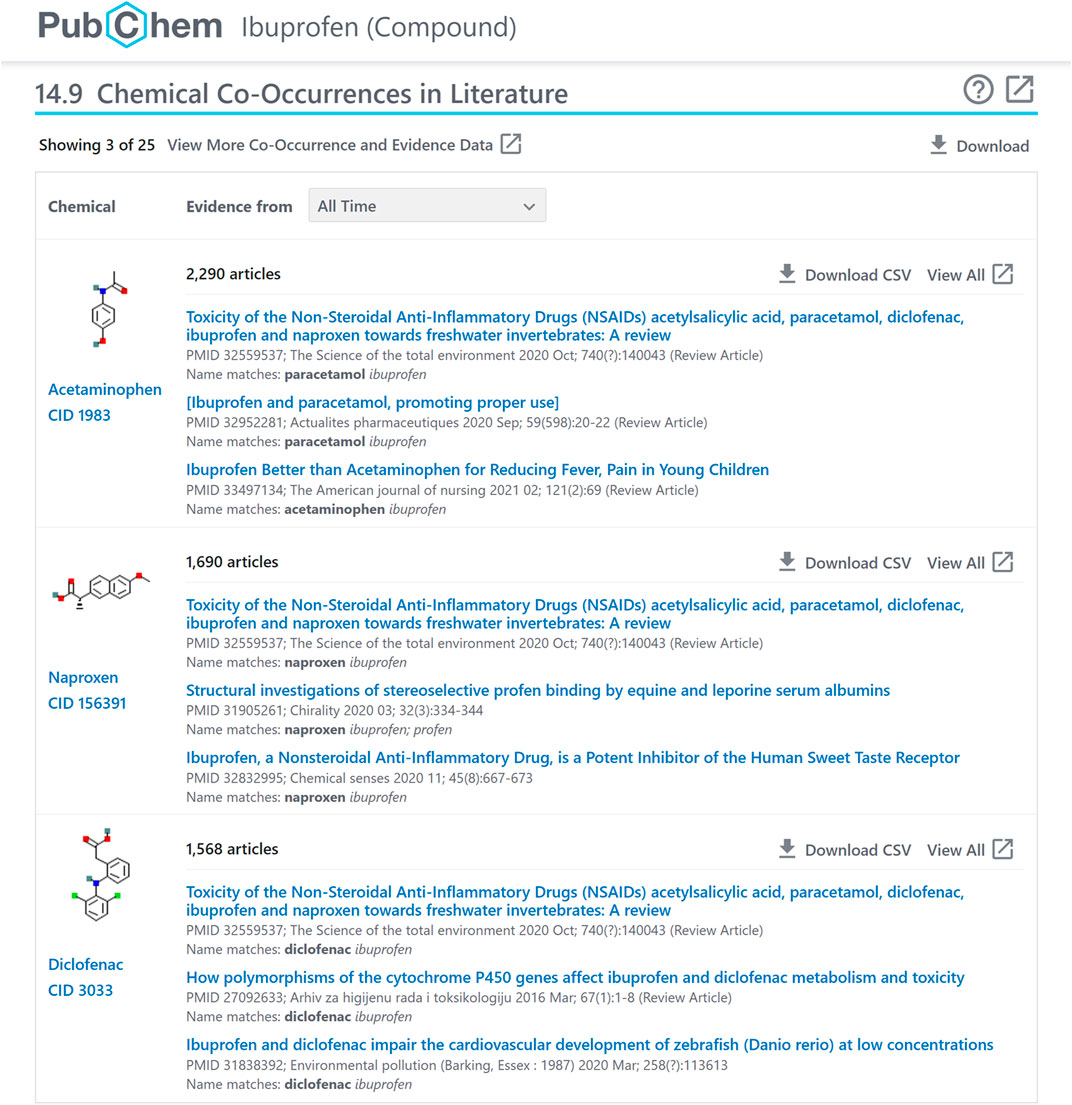
图4显示了布洛芬的化学共现面板(https://pubchem.ncbi.nlm.nih.gov/composite/3672#section=化学-文学中的共现现象)通过面板和控制选项中包含的信息注释,帮助用户检查关系的上下文和可靠性:
① 查询复合;
② 一些最近的非冗余相邻化合物(基于使用等式3)带有指向相应记录的链接(例如。,https://pubchem.ncbi.nlm.nih.gov/compound/1983);
③ 带有链接的相关PubMed记录样本(例如。,https://www.ncbi.nlm.nih.gov/pubmed/32559537);
④ 选择时间间隔(所有时间,或过去1年、5年或10年);
⑤ 切换到具有更多邻居和采样记录的扩展视图(例如。,https://pubchem.ncbi.nlm.nih.gov/composite/3672#section=化学-文学作品与全屏同步=真);
⑥ 下载JSON、XML或ASNT格式的共现数据(例如。,https://pubchem.ncbi.nlm.nih.gov/link_db/link_db_server.cgi?format=JSON&type=ChemicalNeighbor&operation=GetAllLinks&id_1=3672&response_type=显示);
⑦ 打开帮助;
⑧ 在新的浏览器窗口中打开面板(例如。,https://pubchem.ncbi.nlm.nih.gov/composite/3672#section=化学-文学作品与全屏同步=真);
⑨ 以CSV格式(例如。,https://pubchem.ncbi.nlm.nih.gov/link_db/link_db_server.cgi?response_type=save&type=ChemicalNeighborAll&operation=GetCSV&id_1=3672&id_2=1983);
⑩ 查看查询和相邻化合物同时出现的PubMed记录(例如。,https://pubchem.ncbi.nlm.nih.gov/link_db/link_db_server.cgi?type=ChemicalNeighborAll&operation=RedirectToEntrez&id_1=3672&id_2=1983).
文献知识小组的基础数据每周定期更新。下一节中的数据是在2021年2月底生成的。
结果
PubMed记录注释和匹配的一般统计数据如所示表2在3220万份PubMed记录中(截至2021年2月底),有140万份记录具有化学注释,其中1142万份记录的化学注释与PubChem化合物相匹配,2946万份Pub Chem化合物与Pub Med记录相匹配。请注意,疾病字典中的几乎所有疾病术语,包括MeSH树的所有级别,都解析为MeSH标题和补充概念记录(Lowe等人,2016年).
PubMed记录注释中化合物、基因/蛋白质和疾病的出现分布如所示图5PubMed记录中最常提及的三类实体列在表3——5。请注意,大多数注释用于少数经常提及的实体。例如,79.9%的唯一CID-PMID对只包含294.6K个CID中的1%。五种最常见的化学物质是水(CID 962)、D-葡萄糖(CID 5793)、氧(CID 977)、乙醇(CID 702)和钙(CID 5460341)。特别是,823.7K份PubMed记录中对水进行了注释,这相当于用化学品注释的所有PubMed记录的5.9%,以及所有PubMet记录的2.6%。基因/蛋白质和疾病类别中最常提及的实体分别是胰岛素和肿瘤,分别出现在329.4K和246M PubMed记录中。
图6显示了化学物质、基因/蛋白质和疾病的邻域数量分布(化学邻域计数用于非冗余化学邻域,使用中描述的方法生成冗余消除). 因为经常提到的实体可能与其他实体一起出现,所以它们通常有成千上万个邻居。例如,最常提到的化学物质水有47.2K个非冗余化学邻居、15.9K个基因/蛋白质邻居和4.5K个疾病邻居(表3). 对于基因/蛋白质类别,胰岛素是最常见的,它有13.4K个非冗余化学邻居、12.3K个基因/蛋白质邻居和3.9K个疾病邻居(表4). 最常见的疾病术语“肿瘤”出现在47.1K非冗余化学邻居、28.3K基因/蛋白邻居和6.0K疾病邻居中(表5).
重要的是,当使用等式2这种偏见是通过使用基于信息增益的共现评分方案来解决的,等式3,4为了说明校正的重要性,请考虑丙酮的化学共现面板(CID:180):
https://pubchem.ncbi.nlm.nih.gov/composite/180#section=化学-文学中的共现现象.
丙酮的三个最接近的非冗余化学邻居是甲醇(CID 887)、乙醇(CID 702)和水(CID 962)。请注意,水被列为第三个最接近的,尽管它与丙酮的共同提及频率高于其他两个邻居(水记录为4.59K,甲醇记录为3.30K,乙醇记录为4.3K)。这是因为公式3.
图7说明了CID1-CID2-PMID三联体的相关得分值的分布。请注意,右边的一组列说明了最重要的共现现象:240以上的得分值通常是在标题中注释了两个复合词并在摘要中的两个或多个句子中一起提到时产生的。例如,维生素B2和钴在PMID 33053716中的相关性得分非常高,为275分,标题为:“维生素B12和家畜钴代谢的关系:最新进展”(Gonzalez-Montana等人,2020年),因为中列出的所有因素PubMed记录中一对实体的相关性得分有助于相关性得分,如下图所示图8这两种化学物质同时出现在标题中以及摘要中的多个句子中。此外,这篇论文是一年前发表的最新评论文章。重要但不太重要的共现模式产生的相关性得分在140-240之间,对应于图7。通常,当标题中注释了两个化合物并在摘要中的一句话中同时提及时,会产生140–240范围内的得分值。
理解和比较等式3,4,让我们用另一种形式重写它们。等式3可以写为
其中修正系数定义为
和
修正系数的值在里面等式5a当邻居在数据集中表现良好(即PubMed文章中经常提到)可与。对于(具有匹配化学注释的PubMed记录的数量;表3),什么时候、和什么时候.约有1.78K种化合物值为1/2或以下。其中82种化合物具有值1/3或以下。水是最小的值,等于0.161。修正系数的值在里面等式5a对于化学邻居,如图9.
类似于等式3,等式4,可以写在表格中
其中修正系数定义为
和
利率的值由定义等式5a,6b条等于相应修正系数的速率和:
自为所有人,为所有人.因此,.
举例说明使用等式6a,将D-葡萄糖(CID:5793)视为胆固醇的邻居(CID:5997)。在提到D-葡萄糖的447.6K PubMed记录中,有37.8K记录中同时提到胆固醇和D-葡萄糖。D-葡萄糖的值为0.80,校正系数的值为0.20。费率的价值取决于PubMed记录的计数,以获取两组PubMed记录中的相关得分值:D-葡萄糖与胆固醇共存的记录集和D-葡萄糖和任何PubChem化合物共存的记录集中(如果D-葡萄糖与多个化合物同时出现,则取相关性得分的最大值)。相应的条形图如所示图10.结果值为1.18,修正系数的值为0.24。
如中所述冗余消除,从每组具有相同父连接性的化合物中选择一个具有代表性的化合物,以避免冗余信息阻塞知识面板。在PubMed记录中匹配的294.6K CID中,101.1K CID(34%)与另一个匹配化合物具有相同的父连接性,形成31.5K组。其余193.5K个CID(66%)是单例。这导致总共225.0K个CID组(即31.5K个多CID组加上193.5K个单CID组),从每个CID组中选择一个代表性CID以生成非冗余的化学邻居。最大的一组包含191个CID,对应于柠檬酸(CID 311)及其各种盐形式(柠檬酸钠、柠檬酸钙、柠檬酸钾等)。
讨论
PubChem文献知识面板在PubChem-Compound、Gene和Protein页面中实现,它是一个探索工具,显示生物医学文献中共同提到的几个最相关的非冗余实体,用于查看各个记录(查询实体),以及一些最相关的PubMed记录。这些面板帮助用户快速发现化学物质、基因、蛋白质和疾病之间的重要关系,并快速了解一组论文中的关系。当数据集太大而无法检查时,这尤其有益。PubMed记录的一个样本共同提到了这些实体,这有助于用户了解关系的性质和可靠性。此外,用户可以下载感兴趣的论文列表并阅读,以获得更深入的理解。
用于开发知识面板的方法的局限性包括当前共现模型本身的局限性以及用于命名实体识别和数据库匹配的技术的局限性。虽然基于PubChem记录中命名实体共现的方法是一种有用的数据探索工具,但它基于一个简单的众所周知的语言模型。有理由认为,即使是更复杂的模型也可能产生更好的结果。
当同一个单词可能具有多个含义和多个匹配时,基于词典的方法和我们使用的术语匹配过程会出现歧义。例如,lead在普通英语中除了是CID:5352425复合词的同义词外,还有多种含义。视网膜是CID:638015化合物的同义词,也是MeSH ID:D012164下与各种视网膜疾病相关的解剖学术语。CAT是过氧化氢酶(NCBI基因ID:847)的基因符号,这是一种生物家猫的通用名称,NCBI分类ID:9685,计算机辅助断层扫描在MeSH ID:D014057下。MP2是化合物的同义词,CID:15942661,成熟多肽的基因符号(NCBI基因ID:547827),二阶Møller–Plesset扰动理论的缩写,以及视频文件格式,也称为MPEG-2。在基于词典的方法中,减少歧义的方法包括将术语放在区分大小写的词典中,决定始终指定“最常见”的含义,或将术语放进否定词典中。然而,在许多情况下,词义与上下文相关,需要能够解决上下文敏感情况的新消歧方法。我们正在研究算法和方法,以便更好地理解和利用歧义术语的上下文含义。与许多其他知识资源一样,PubChem方法可以从合并来自更可信、更人性化的数据源的信息中获益。额外的策划信息将允许进一步交叉验证数据,并通过改进评分促进可信和可靠的信息。
目前,用于知识面板的共现分数使用等式3,但我们正在使用更先进的评分方案,如等式4处理实体的邻居列表中的(近)冗余也是未来开发中需要解决的一个重要问题。特别是,化学名称-结构关联是一个需要改进的重要领域。由于各种原因,一个化学名称通常与多个彼此略有不同的化学结构相关联(例如,在立体化学、同位素组成、共振形式、互变结构、混合物/盐形式等方面)(Hahnke等人,2018年). 虽然PubChem化学结构和化学名称处理试图处理此类问题,但它并不完善。因此,单个化学名称对(每个化学名称都可以映射到多个CID)通常会导致许多结构相似的CID-CID对,从而增加了化学品之间相邻关系的冗余。为化学名称选择好的代表性结构的改进算法将加强对这种冗余的处理。
当PubMed文章中注释了更具体的疾病名称(例如乳腺癌)时,考虑是否自动注释更广泛的疾病术语(例如癌症)也很有趣。虽然这种对更广泛疾病术语的扩展注释将有助于发现实体之间的新关系,但也会增加相邻关系冗余。
PubMed记录包含生物医学和生命科学出版物的标题、摘要和一些其他元数据。为了支持更广泛的科学界,我们正在努力将我们的方法扩展到PubMed记录之外。这可能包括在PubMed未涵盖的科学领域发表的论文(如化学、物理、材料科学和纳米技术)或PubMed-Central和其他公共存储库中提供的全文文章(如政府报告或策划的文本注释)。专利文件也很有趣。然而,将该方法扩展到高度异构的数据集需要深入了解数据的相对重要性,并重新设计评分方案和数据表示。
总之,我们相信本文所述的努力为用户提供了一种有效的手段,可以快速高效地了解与给定PubChem记录相关的关键生物医学实体。用户可以快速浏览一组相关PubMed论文,了解相关实体(化学品、基因/蛋白质或疾病)。可下载的内容允许用户进一步探索和分析所提供的链接。虽然可以做出许多改进,但它已经非常有帮助,并受到用户的欢迎。
数据可用性声明
研究中提出的原始贡献包含在文章/补充材料中,可以向通讯作者进行进一步询问。
作者贡献
LZ和EB提出了该方法。LZ开发并实现了所描述的计算算法。所有作者都参与了数据流的开发、实施和集成,以便在PubChem内使用知识小组,为起草文章做出了贡献,并提供了关键修订。所有作者都同意提交文章的最终版本。
基金
国立卫生研究院国家医学图书馆的校内研究项目。开放存取费用资助:国立卫生研究院国家医学图书馆的校内研究项目。
利益冲突
作者声明,该研究是在没有任何可能被解释为潜在利益冲突的商业或金融关系的情况下进行的。
致谢
我们感谢我们与NextMove Software的卓有成效的合作,并感谢Roger A.Sayle、Daniel Lowe、Noel O’Boyle和John W.Mayfield的响应能力和持续支持。我们感谢与Terence Murphy、Jean Thierry-Mieg、Dan-Sung Cho和Chih-Hsuan Wei进行的富有成效的讨论。
缩写
CID,PubChem化合物标识符(整数);PMID、PubMed标识符(一个整数);MeSH,医学主题标题;OPSIN,系统IUPAC命名的开放解析器;国际纯化学和应用化学联合会;人类基因组组织;HGNC、HUGO基因命名委员会;国际基础和临床药理学联合会;英国药理学学会BPS;EC编号、酶委员会编号;TF-IDF,术语频率–反转文档频率;自然语言处理;JSON、JavaScript对象表示法;XML,可扩展标记语言;CSV,逗号分隔值;ASNT,抽象语法符号文本。
工具书类
Aizawa,A.(2003年)。Tf-Idf测度的信息论视角。信息处理。管理。39 (1), 45–65. doi:10.1016/s0306-4573(02)00021-3
交叉引用全文|谷歌学者
Armstrong,J.F.、Faccenda,E.、Harding,S.D.、Pawson,A.J.、Southan,C.、Sharman,J.L.等人(2020年)。2020年IUPHAR/BPS药理学指南:扩展免疫药理学内容并引入IUPHAR/MMV疟疾药理学指南。核酸研究。48(D1),D1006–D1021。doi:10.1093/nar/gkz951
PubMed摘要|交叉引用全文|谷歌学者
Bairoch,A.(2000年)。2000年的ENZYME数据库。核酸研究。28 (1), 304–305. doi:10.1093/nar/28.1.304
PubMed摘要|交叉引用全文|谷歌学者
Bateman,A.、Martin,M.J.、O'Donovan,C.、Magrane,M.、Alpi,E.、Antunes,R.等人(2017年)。UniProt:通用蛋白质知识库。核酸研究。45(D1),D158–D169。doi:10.1093/nar/gkw1099
PubMed摘要|交叉引用全文|谷歌学者
Braschi,B.、Denny,P.、Gray,K.、Jones,T.、Seal,R.、Tweedie,S.等人(2019年)。Genenames.org:2019年HGNC和VGNC资源。核酸研究。47(D1),D786–D792。数字对象标识代码:10.1093/nar/gky930
PubMed摘要|交叉引用全文|谷歌学者
Devlin,J.、Chang,M.-W.、Lee,K.和Toutanova,K.(2019年)。《BERT:语言理解深度双向变形金刚的预训练》,in计算语言学协会北美分会2019年会议论文集:人类语言技术,第一卷(长短论文),美国明尼苏达州明尼阿波利斯宾夕法尼亚州斯特劳德斯堡:计算语言学协会, 4171–4186.
谷歌学者
Ehrlinger,L.和Wöß,W.(2016)。《走向知识图的定义》第十二届语义系统国际会议——SEMANTiCS2016和第一届语义变化和演变语义国际研讨会海报和演示轨道联合会议记录(SuCCESS16). (德国莱比锡)2016年9月12日至15日, 13–16.
谷歌学者
Fu,G.、Batchelor,C.、Dumoniter,M.、Hastings,J.、Willighagen,E.和Bolton,E.(2015)。PubChemRDF:面向PubChem化合物和物质数据库的语义注释。J.化学信息7 (1), 34. doi:10.1186/s13321-015-0084-4
PubMed摘要|交叉引用全文|谷歌学者
González-Montaña,J.-R.、Escalera-Valente,F.、Alonso,a.J.、Lomillos,J.M.、Robles,R.和Alonso.M.E.(2020年)。家畜维生素B12与钴代谢的关系:最新进展。动物10 (10), 1855. doi:10.3390/ani10101855
交叉引用全文|谷歌学者
Gurulingappa,H.、Mudi,A.、Toldo,L.、Hofmann-Apitius,M.和Bhate,J.(2013)。挖掘化学信息文献的挑战。RSC高级。3, 16194–16211. doi:10.1039/c3ra40787j
交叉引用全文|谷歌学者
Hähnke,V.D.、Kim,S.和Bolton,E.E.(2018年)。PubChem化学结构标准化。J.化学信息10 (1), 36. doi:10.1186/s13321-018-0293-8
PubMed摘要|交叉引用全文|谷歌学者
Jessop,D.、Adams,S.、Willighagen,E.、Hawizy,L.和Murray-Rust,P.(2012年)。OSCAR4:一个灵活的化学文本挖掘架构。化学信息学杂志3, 11. doi:10.1186/1758-2946-3-41
交叉引用全文|谷歌学者
Kim,S.、Chen,J.、Cheng,T.、Gindulyte,A.、He,J.和He,S.等人(2021年)。PubChem in 2021:新的数据内容和改进的Web界面。核酸研究。49(D1),D1388–D1395。doi:10.1093/nar/gkaa971
PubMed摘要|交叉引用全文|谷歌学者
Kim,S.、Chen,J.、Cheng,T.、Gindulyte,A.、He,J.和He,S.等人(2019年)。PubChem 2019更新:改进化学数据访问。核酸研究。47(D1),D1102–D1109。doi:10.1093/nar/gky1033
PubMed摘要|交叉引用全文|谷歌学者
Kim,S.、Thiessen,P.A.、Bolton,E.E.、Chen,J.、Fu,G.、Gindulyte,A.等人(2016a)。PubChem物质和化合物数据库。核酸研究。第44页,D1202–D1213。doi:10.1093/nar/gkv951
PubMed摘要|交叉引用全文|谷歌学者
Kim,S.、Thiessen,P.A.、Cheng,T.、Yu,B.、Shoemaker,B.A.、Wang,J.等人(2016b)。公共化学文献信息:公共化学记录与科学文章之间的联系。J.化学信息8, 32. doi:10.1186/s13321-016-0142-6
PubMed摘要|交叉引用全文|谷歌学者
Kullback,S.和Leibler,R.A.(1951年)。关于信息和充分性。安。数学。统计师。22 (1), 79–86. doi:10.1214/aoms/1177729694
交叉引用全文|谷歌学者
Lee,J.、Yoon,W.、Kim,S.、Kim和D.、Kim、S.、So和C.H.等人(2020年)。BioBERT:用于生物医学文本挖掘的预训练生物医学语言表示模型。生物信息学36 (4), 1234–1240. doi:10.1093/bioinformatics/btz682
PubMed摘要|交叉引用全文|谷歌学者
Lowe,D.M.、Corbett,P.T.、Murray-Rust,P.和Glen,R.C.(2011年)。结构化学名称:OPSIN,一种开源解决方案。化学杂志。信息模型。51(3),739-753。doi:10.1021/ci100384d文件
PubMed摘要|交叉引用全文|谷歌学者
Lowe,D.M.、O'Boyle,N.M.和Sayle,R.A.(2016)。使用维基百科提高召回率的高效化学疾病识别和关系提取。数据库2016年,baw039。doi:10.1093/数据库/baw039
PubMed摘要|交叉引用全文|谷歌学者
Lowe,D.M.和Sayle,R.A.(2015)。LeadMine:一种语法和字典驱动的实体识别方法。J.化学信息第7章第5节。doi:10.1186/1758-2946-7-s1-s5
PubMed摘要|交叉引用全文|谷歌学者
Manning,C.D.和Schütze,H.(1999)。统计自然语言处理基础马萨诸塞州剑桥市:麻省理工学院出版社.
Manning,C.、Raghavan,P.和Schütze,H.(2008)。信息检索导论纽约州纽约市:剑桥大学出版社.doi:10.1017/cbo9780511809071
交叉引用全文
Needleman,S.B.和Wunsch,C.D.(1970年)。一种适用于搜索两种蛋白质氨基酸序列相似性的通用方法。分子生物学杂志。48 (3), 443–453. doi:10.1016/0022-2836(70)90057-4
PubMed摘要|交叉引用全文|谷歌学者
Pletscher-Frankild,S.、Pallejá,A.、Tsafou,K.、Binder,J.X.和Jensen,L.J.(2015)。疾病:疾病基因关联的文本挖掘和数据集成。方法74, 83–89. doi:10.1016/j.meth.2014.11.020
PubMed摘要|交叉引用全文|谷歌学者
Rajaraman,A.和Ullman,J.(2011年)。海量数据集的挖掘。纽约州纽约市:剑桥大学出版社.doi:10.1017/cbo9781139058452
交叉引用全文
Ratinov,L.R.D.(2009)。《命名实体识别中的设计挑战和误解》第十三届计算自然语言学习会议,科罗拉多州博尔德,2009年6月宾夕法尼亚州斯特劳德斯堡:计算语言学协会.doi:10.3115/1596374.1596339
交叉引用全文|谷歌学者
Robertson,S.(2004)。理解反向文档频率:关于IDF的理论论证。J.文件60 (5), 503–520. doi:10.1108/00220410410560582
交叉引用全文|谷歌学者
Rocktäschel,T.、Weidlich,M.和Leser,U.(2012年)。ChemSpot:一个用于化学命名实体识别的混合系统。生物信息学28, 1633–1640. doi:10.1093/bioinformatics/bts183
PubMed摘要|交叉引用全文|谷歌学者
Sayers,E.W.、Agarwala,R.、Bolton,E.E.、Brister,J.R.、Canese,K.、Clark,K.等人(2019年)。国家生物技术信息中心的数据库资源。核酸研究。47(数据库问题),D23–D28。doi:10.1093/nar/gky1069
PubMed摘要|交叉引用全文|谷歌学者
Sayers,E.W.、Beck,J.、Bolton,E.E.、Bourexis,D.、Brister,J.R.、Canese,K.等人(2021年)。国家生物技术信息中心的数据库资源。核酸研究。49(D1),D10-D17。doi:10.1093/nar/gkaa1892
PubMed摘要|交叉引用全文|谷歌学者
Sayle,R.、Xie,P.H.和Muresan,S.(2011年)。使用无限词典和自动拼写更正改进专利的化学文本挖掘。化学杂志。信息模型。52, 51–62. doi:10.1021/ci200463r
PubMed摘要|交叉引用全文|谷歌学者
Song,H.-J、Jo,B.-C、Park,C.-Y、Kim,J.-D和Kim,Y.-S(2018)。生物医学文献中命名实体识别方法的比较。生物识别。工程在线17(补充2),158。doi:10.1186/s12938-018-0573-6
PubMed摘要|交叉引用全文|谷歌学者
田毅、沈伟、宋毅、费熙、何明、李凯(2020)。利用句法信息改进生物医学命名实体识别。BMC生物信息学21, 539. doi:10.1186/s12859-020-03834-6
PubMed摘要|交叉引用全文|谷歌学者
Vazquez,M.、Krallinger,M.,Leitner,F.和Valencia,A.(2011年)。药物和化学化合物的文本挖掘:方法、工具和应用。摩尔Inf。30, 506–519. doi:10.1002/minf.201100005年
交叉引用全文|谷歌学者