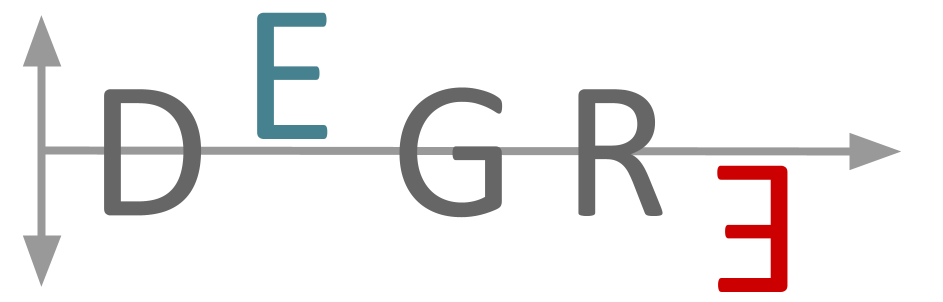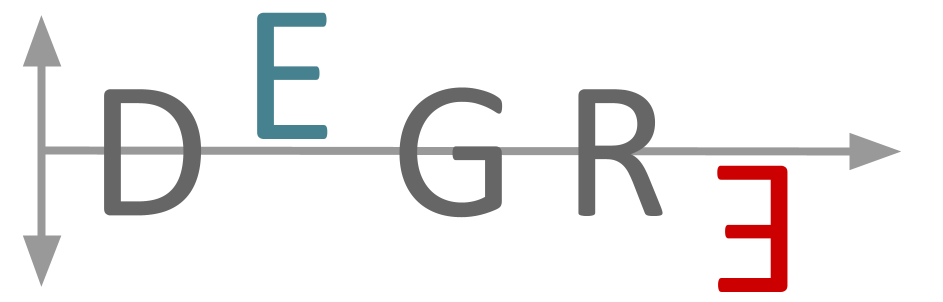推断基于广义线性混合模型的差异表达基因DEGRE R包
范围
 替换文本
替换文本
DEGRE是一个R包,旨在识别差异以成对方式表达基因(DEG)并考虑插入实验设计中个体的随机效应。这个包中有一个预处理步骤,负责筛选以下基因可能会削弱DEG的推断。为此,DEGRE使用广义负二项分布的线性混合模型(GLMM)。
DEGRE的存储库位于GitHub上https://github.com/lipinfo-lncc/DEGRE。在此网站中,您可以报告bug并获得帮助。
引用
DEGRE软件包正在发布中。如果你使用它,请引用它在你的研究中。
你可以在下面引用它:
#要在出版物中引用包“DEGRE”,请使用:
#Machado等人(2022年)。DEGRE:使用DEGRE R包中的广义线性混合模型推断差异表达基因。R软件包版本1.0。
#LaTeX用户的BibTeX条目是
#@手动{,
#title={使用DEGRE R包中的广义线性混合模型推断差异表达基因},
#作者={Douglas Terra Machado、Otavio Jose Bernardes Brustolini、Yasmin Cortes Martins、Marco Antonio Grivet Mattoso Maia、Ana Tereza Ribeiro Vasconcelos},
#年份={2022},
#注意={R包版本1.0},
# }
#注意:此引文信息是从包中自动生成的
#描述文件,可能需要手动编辑,请参阅“help(“引文”)”。
DEGRE的安装包裹
要安装DEGRE软件包,必须运行:
图书馆(开发工具)
安装github(“labinfo lncc/DDEGRE”,参考=“主要”)
所需的程序包
DEGRE包具有以下几个功能包装:
- parglm,版本0.1.7
- glmmTMB,版本1.1.3
- doParallel,版本1.0.17
- foreach,1.5.2版
- tibble,3.1.6版
- dplyr,版本1.0.9
- ggplot2,版本3.3.5
- ggpubr,版本0.4.0
- ggreset,版本0.9.1
注意:如果您在glmmTMB程序包方面遇到问题,您可以尝试删除并重新安装它。
快速入门
要使用此包,必须有一个包含读取内容的矩阵每个生物复制和设计矩阵中每个基因的计数包含样本信息。在计数矩阵中,行表示基因,而柱表示这两者的生物复制实验条件。在设计矩阵中,列表示样品的相关信息。
要开始使用包,您需要加载库:
然后您可以应用DEGRE的快速启动功能:
#阅读计数矩阵和设计矩阵的示例:
目录<- system.file(系统文件)(“数据”,包装= “度”)
选项卡<- 读取.csv(文件路径(目录,“计数矩阵示例.csv”))
行名称(选项卡)<-选项卡[,1]; 选项卡<-选项卡[,-1]
des公司<- 读取.csv(文件路径(目录,“design_matrix_for_example.csv”))
#运行DEGRE功能:
结果<- DEGRE(度)(count_矩阵=选项卡,
p_value_调整= “伯克希尔哈撒韦”,
设计矩阵=数据集,
公式= “条件+(1|性别)”)
DEGRE的输出功能
其输出(表示为结果以上)是一个数据帧对象。此对象具有以下列:
- ID-用户插入的基因ID
计数矩阵.
- log2FC-log2折叠更改。
- P(P)-值-P(P)-中每个基因的计算值Wald测试。
- 问-值-修正值P(P)-使用Benjamini的值Hochberg(BH)或Bonferroni(BON)方法。
- averagelogCPM-每个基因的log2CPM。
关于DEGRE的更多信息功能
此函数具有以下参数:
结果<- DEGRE(度)(计数矩阵,
设计矩阵,
公式,
p_value_调整)
如您所见,DEGRE函数具有重要参数,这些参数如下所述:
count_矩阵-一个数据帧对象。它接收矩阵作为输入。设计矩阵-一个数据帧对象。它接收实验设计矩阵。样本名称必须为在第一列中标识。此矩阵也可以有更多列包含固定效应和随机效应的信息样品。公式-它接受固定和随机效果描述。p_value_调整-所有P(P)-计算的值必须进行纠正,DEGRE软件包提供了两种可能性:“BH”(Benjamini-Hochberg)校正(默认)和“BON”(Bonferroni)修正。
一个实用的固定效应和随机效应示例
在计划周密的实验设计中,固定效应是其特点这在被调查的个体中是恒定的。假设我们正在进行一项RNA-Seq实验,以确定接受特殊治疗的人群和只接受水而不接受药物。这方面的说明对比如下:

请注意,我们知道A组中的所有人都收到了治疗。相比之下,B组的所有人只接受了水。这个我们可以控制两者之间效果的比较小组理解固定效应。
假设在两组中,我们都有一些与生物性,其中有不同性别的男性和女性比例。如下所示:

这种效果可以作为随机效果处理,以消除任何与男性和女性之间差异相关的潜在偏差1²³。其他难以控制的随机效应示例可能包括体内不同浓度的生化分子这两组都与基因表达有关。
托拉尔、伊丽莎、昆汀·奎罗斯、达米安·罗塞尔、吉尔伯特·达托,埃里克·加斯特(Eric Gasset)、大卫·麦肯齐(David J.McKenzie)、卡罗琳·罗梅斯坦(Caroline Romestaing)、珍妮·马奇(Jean-Marc)弗罗门汀、克莱尔·萨劳和洛伊克·特利耶。2021.“牧草的变化猎物数量下降引起的模式具有主要的生物能量小型远洋鱼的后果。”动物生态学杂志90(10): 2289–2301.
Kleyheeg、Erik、Bart A.Nolet、Sandra Otero Ojea和Merel B。很快。2018年,“肠道之间关系的机械评估水禽的形态和虫内种子散布。”生态学和进化8(22):10857–67。
多诺万(Donovan)、玛格丽特·K·R·阿格涅斯卡(Margaret K.R.)、阿格涅斯·达安托尼奥·科洛诺斯卡(Agnieska D’Antonio-Chronowska)、马特奥(Matteo)D’Antonio和Kelly A.Frazer。2020年,“GTEx的蜂窝反褶积组织推动疾病的发现和细胞类型相关调控变体。”《自然通讯》11(1):955。
您可以使用一些应用程序!
筛选来自最终数据帧的基因ID
这里有一些代码可以用于筛选特定的基因ID。
假设您要筛选与ENSMUSG00000000881基因ID。然后,您可以尝试:
过滤基因<-结果[结果$身份证件== “ENSMUSG00000000881”,]
打印(过滤基因)
应用此筛选步骤,您将得到以下结果:
#ID log2FC P值Q值
#ENSMUSG0000000881-3.71189 1.83797593742305e-64 7.765448e-64
然而,假设你在载体中有特定的基因ID示例:
发电机IDs_to_filter<- c(c)(“ENSMUSG00000000881”,
“ENSMUSG00000002820”,
“ENSMUSG00000005610”,
“ENSMUSG00000015484”)
我们可以使用合并函数来获得基因结果。首先,考虑将其转换为data.frame对象:
发电机IDs_to_filter<- 数据帧(ID编号=gene_IDs_to_filter)
打印(基因_IDs_to_filter)
#ID编号
#ENSMUSG00000000881号机组
#ENSMUSG00000002820
#ENSMUSG00000005610号
#ENSMUSG00000015484号
然后,您可以使用合并功能:
过滤基因<- 合并(结果,gene_IDs_to_filter,由= “ID”)
打印(过滤基因)
#ID log2FC P值Q值
#ENSMUSG00000000881-3.7118904 1.83797593742305e-64 7.765448e-64号
#ENSMUSG00000002820-4.4200312 5.1046515023083e-105 2.974780e-104号
#ENSMUSG00000005610 1.4922973 2.41432633104465e-115 1.522467e-114
#ENSMUSG00000015484-0.6455689 7.54077969856174e-06 1.098614e-05
获取结果来自问-价值截止点
要筛选问-值,您可以执行以下内容:
结果q_value_cutoff<-结果[结果$`Q值` < 0.05,]
在这里,我们根据5%的重要性筛选出一个临界值,但你可以改变它。
要检查它是否正常工作,可以检查最大值问-值来自结果和结果q_value_cutoff:
最大值(结果_q_value_cutoff$`Q值`)
将其与结果即数据帧如果不进行过滤,您将得到:
其中它向您显示过滤器工作正常。
获取biomeRt包中的基因注释
如果您只有基因ID但想要基因名称,可以使用获取信号群信息的biomRt包。
首先,如果您没有安装biomaRt,请安装它遵循建议在这里.
然后,您必须执行库操作:
使用以下代码,您可以从穆斯肌肉物种:
合奏<- useMart网站(“合奏”,
数据集=“mmusculus_gene_ensembl”)
基因<- 获取BM(属性=c(c)('集合基因id',“external_gene_name”),
过滤器= '集合基因id',
值=结果$ID中,
市场=合奏)
您可以使用merge函数带来DEGRE输出信息基因。注意:您需要重命名基因数据的列以使具有基因ID的列具有相同的名称在两个数据帧中:结果从DEGRE函数(DEGRE包装)和基因从getBM函数(biomeRt包装)。
列名(基因)[1]<- “ID”
学位结果_基因名称<- 合并(结果、基因、,由= “ID”)
数据帧DEGRE结果_基因名称现在有了基因信号群的名称和DEGRE函数的所有信息输出。
绘制DEGRE向您提供的套餐
火山图
DEGRE包具有VolcanoDEGRE功能,可以可视化应用log2FC计算下调和上调基因的比例切断。
下面是默认功能的快速启动参数:
火山爆发(结果=结果,
log2FC_切断= 1,
巴德(padj)= 0.05,
德拉贝尔= "",
字体.x= 10,
字体.y= 10,
字体.tickslab= 10,
下调颜色= “珊瑚2”,
上调颜色= “矢车菊蓝”,
xlab公司= “log2Foldchange”,
伊拉布= “-log10(P值)”,
图例_位置= “正确”,
图例.标题= “法规”)
在结果参数,必须指定数据帧你想要绘图。下面显示了以下结果。

我们建议您使用以下png函数保存它参数:
png公司(文件名= “Volcano_DEGRE_example.png”,
宽度= 1700,
高度= 1200,
资源= 350)
#然后,粘贴代码以绘制条形图。
在此处粘贴代码以绘制条形图并运行它。
#因此,您可以关闭绘图:
开发关闭()
可以自由更改关于VolcanoDEGRE的任何论点功能。
解释的参数如下所述:
结果-一个数据帧对象。它接收DEGRE函数的输出,无论是否过滤输入。
log2FC_切断-它存储了log2折叠更改。
巴德(padj)-它存储了P(P)-调整后的值(问-值)。
字体。x-x轴的字体大小。
字体。年-y轴的字体大小。
字体.tickslab-刻度的字体大小实验室。
下调_颜色-的颜色下调基因。默认值为“coral2”。
上调的颜色-上调后的颜色基因。默认为“矢车菊蓝”。
xlab公司-x实验室文本。默认值为“log2Foldchange”。
伊拉布-y实验室文本。默认值为“-log10(P值)”。
图例_位置-您需要在此处指定图例的位置。默认值为“right”。
图例.标题-图例的标题。
条形图
DEGRE包具有BarGraphDEGRE函数以获取条形图显示下调和上调基因的数量。
下面是默认功能的快速启动参数:
条形图度数(结果=结果,
log2FC_切断= 1,
下调颜色= “珊瑚2”,
上调颜色= “矢车菊蓝”,
xlab公司= “法规”,
伊拉布= “基因数量”,
字体.x= 10,
字体.y= 10,
字体.tickslab= 10,
图例_位置= “正确”,
图例.标题= “法规”)
在结果参数,必须指定数据帧你想要绘图。下面显示了以下结果。

我们建议您使用以下png函数保存它参数:
png公司(文件名= “BarPlot_DEGRE_example.png”,
宽度= 1500,
高度= 1200,
资源= 350)
#然后,粘贴代码以进行条形图绘制。
在此处粘贴代码以绘制条形图并运行它。
#因此,您可以关闭绘图:
开发关闭()
可以自由更改BarGraphDEGRE上的任何参数功能。
解释的参数如下所述:
结果-一个数据帧对象。它接收DEGRE函数的输出,过滤或不过滤,作为输入。log2FC_切断-它存储了log2折叠更改。下调颜色-与下调基因的数量。默认值为“coral2”。上调颜色-与数字相关的条形图颜色上调基因。默认为“矢车菊蓝”。xlab公司-x实验室文本。默认值为“法规”。伊拉布-y实验室文本。默认值为“数量基因”。字体。x-x轴的字体大小。字体。年-y轴的字体大小。方尖碑-扁虱实验室的字体大小。图例_位置-你需要在这里指定位置传说中的。默认值为“right”。图例.标题-图例的标题。