Kaur Alasoo是爱沙尼亚塔尔图大学的小组组长。他率先创建了eQTL目录,统一处理的eQTL汇总统计和精细制图结果的最大简编。
他们刚刚发布了他们的自然遗传学方法,所以我们与Kaur坐下来了解更多关于项目是如何合并的。
表达数量性状位点(eQTL)表达量性状是蛋白质mRNA转录物的数量。解释附近基因表达水平变化的基因组变体称为eQTL。
eQTL目录是如何产生的?
我第一次接触Open Targets是通过我在Wellcome Sanger Institute的Dan Gaffney博士介绍的。我的博士毕业后不久,丹·加夫尼(Dan Gaffney)、欧洲生物信息学研究所(EMBL-EBI)的丹尼尔·泽比诺(Daniel Zerbino)和我提交了一份提案,构建一条处理和存储人类eQTL数据的管道。我们的项目得到了资助,从而创建了eQTL目录。
我们不是简单地在一个地方收集所有研究的总结结果,而是希望能够访问原始研究数据,以便使用我们自己的工作流重新分析一切。我记得Open Targets的董事们有一些——可能是合理的——怀疑:我们必须以艰难的方式做到这一点吗?
一旦我们开始研究这个问题,我们很快意识到我们不可能用另一种方式来做。处理后的数据集之间差异太大,很难进行比较。重新处理数据很复杂,但最终数据要干净得多,也更容易使用。研究人员可以利用这些数据进行协同定位研究或查找,而无需花费数周或数月时间查找和协调数据。
爱沙尼亚和英国之间的项目是什么样的?
老实说,这是我参与过的最好的合作之一。我在塔尔图的团队申请访问单个数据集,然后进行数据处理。既然我们有了可以轻松重新运行的健壮工作流,那么集成新数据集就相对容易了。
一旦数据被处理,它就不再包含个人可识别数据,并且可以公开共享。我们将数据传输到EMBL-EBI,James Hayhurst在那里通过FTP提供数据。
James还建立了一个API,使用户能够跨所有数据集查询特定的变体或基因。这相当具有挑战性:在上一版本中,汇总统计数据约为7 TB,相当于3500部高清电影。这只在当前版本中有所增加。构建一个能够高效查询这种规模的数据库的API是一项艰巨的任务。

你会说这是该项目最具挑战性的方面吗?
我想说,运行Catalogue最具挑战性的部分是访问单个原始数据集,因为它们位于非常不同的存储库中,每个存储库都有自己的数据共享系统和门户。有时,如NCBI的基因型和表型数据库(数据库间隙P),申请流程规范。其他存储库的组织性较差,有时我们需要等待6个月才能收到电子邮件的回复。特别令人沮丧的是,在经历了整个过程后,却发现数据要么不可用,要么无法用于此目的。
事实上,收集原始数据的过程非常繁重,这可能就是我们没有直接竞争对手的原因。我们现在已经处理了29份出版物的数据,这是三年半工作的结果。
如何选择要集成的数据集?
当我们向Open Targets提交提案时,我们根据合作伙伴的需求列出了优先数据集列表。我们已经集成了列表中的大多数批量eQTL数据集,所以我们现在正在尝试尽可能全面,并正在应用我们已知的所有数据集。
数据收集的一个问题是,我们不能总是访问单个级别的数据。例如,获得参与者的同意有时会使研究人员无法分享个体基因型。虽然在一个地方有单独的数据可以更容易地统一处理,但这并不是一个严格的要求。因此,我们计划转向数据分析的联邦模型,在该模型中,我们将工作流发送给其他组。然后,他们可以使用我们的方法处理数据,并将汇总结果发送给我们。这种方法的另一个优点是,我们不再需要承担本地存储数据的成本。不幸的是,实现这一目标的标准和技术还不够成熟,无法实现完全无摩擦。
有关eQTL Catalogue的联合分析计划的更多信息,请参阅https://www.ebi.ac.uk/eqtl/联邦分析/.
eQTL目录的下一步是什么?
自从我们提交论文以来,我们又整合了8个数据集,使我们处理的研究总数达到29个。Open Targets Genetics团队正在努力将这些新数据集集成到Genetics Portal的下一版本中。
我们还正在开发新的工作流来集成单单元eQTL数据集。第一批此类研究已经发表,包括最近的一个开放目标项目多巴胺能神经元分化的群体规模分析同样重要的是,这些都要统一处理和提供。
除此之外,还有其他有前途的数据类型,例如染色质和蛋白质数据,我们还需要开发新的工作流。我应该说,这并不是说我们什么都想做——如果有人做了eQTL目录的工作,我们会很高兴!
推特是我新的有趣论文的主要来源,我发现自己参与了越来越多的科学讨论。例如,几周前,克里斯·华莱士(Chris Wallace)、金井雅弘(Masahiro Kanai)和我辩论过如何最好地使用克里斯的共定位方法对eQTL目录进行精细映射。
我最近也有与埃里克·福曼的讨论关于他发现的一些关联,表明变异的效应大小随着你远离DNA的距离而减弱。Eric给出了3819817卢比变体,HAL基因的eQTL。这个eQTL目录显示这只与三个精细映射的可信集重叠,所有这些都是仅针对HAL和仅针对皮肤的eQTL。相同的数据现在也可以是在FiveX浏览器中浏览.
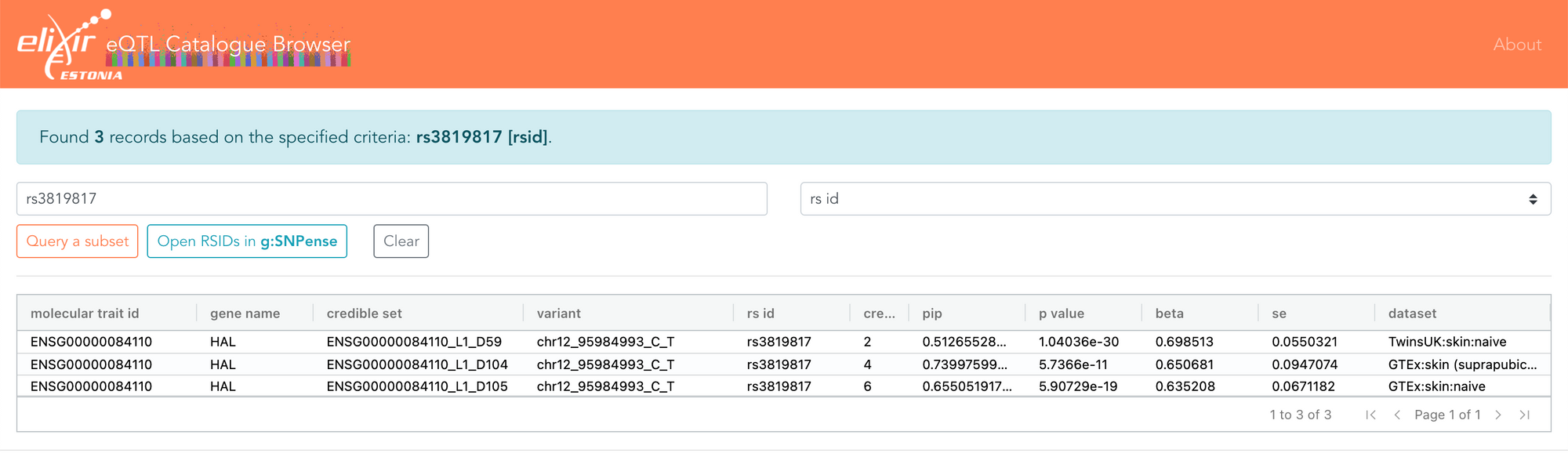 eQTL目录浏览器显示3819817卢比变体与皮肤中HAL的三个精确可信集重叠
eQTL目录浏览器显示3819817卢比变体与皮肤中HAL的三个精确可信集重叠精细映射有助于您估计变异的因果概率,在这种情况下,rs3819817对HAL基因具有51-74%的因果概率。它也是与皮肤中HAL表达最密切相关的变体。
HAL是一种将组氨酸转化为反式葡萄糖酸的酶,是一种参与皮肤色素沉着的代谢物。埃里克的例子表明,如果你携带这种变体,你的反式尿路酸盐水平会更高,维生素D水平会更低,晒伤的次数会更少,你更容易晒黑,这意味着你不太可能需要防晒霜。当eQTL分析在相关组织(皮肤)中指向正确的基因(HAL)时,令人满意。
这些类型的讨论可以促进新的合作,或让您快速获得工作反馈。
eQTL Catalogue团队目前正在寻找愿意以联合方式贡献其数据集的合作者。联系他们eqtlcataloge@ebi.ac.uk.
引用
克里莫夫,N.,海赫斯特,J.D.,佩科娃,K。等。一统一处理的人类基因表达和数量性状基因座剪接简编.自然基因53,1290–1299 (2021). https://doi.org/10.1038/s41588-021-00924-w网址






